19.7A: Transcripción
- Page ID
- 55496
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Defina lo siguiente:
- gen
- transcripción
- Describa brevemente la función de lo siguiente en términos de transcripción:
- mRNA
- extremo 3'
- extremo 5'
- ARN polimerasa
- enlace fosfodiéster
- promotor
- secuencia líder
- secuencia de codificación
- terminador de transcripción
- codón
- Definir lo siguiente en términos de transcripción en células eucariotas:
- intrones
- exones
- ARNm precursor
- gorra
- Cola poli-A
- mRNA maduro
Transcripción en células procariotas
Descripción: El ARN mensajero (ARNm) se sintetiza mediante el apareamiento de bases complementarias de ribonucleótidos con desoxirribonucleótidos para que coincidan con una porción de una cadena de ADN llamada gen. Aunque los genes están presentes en ambas cadenas de ADN, solo se transcribe una cadena para cualquier gen dado. Después de la transcripción, las subunidades ribosómicas 30S y 50S se unen al ARNm y el ARNt inserta los aminoácidos correctos que posteriormente se unen para formar un polipéptido o una proteína a través de un proceso llamado traducción.
La enzima ARN polimerasa transcribe ADN. Esta enzima inicia la transcripción, une los nucleótidos del ARN y termina la transcripción. Para iniciar la transcripción en bacterias, una variedad de proteínas llamadas factores sigma se unen a las ARN polimerasas. Este complejo puede entonces unirse a una secuencia específica de generalmente alrededor de 40 bases desoxirribonucleotídicas llamadas el promotor ubicado a lo largo del ADN antes de la región codificante del gen. El promotor determina qué región del ADN y qué cadena de ADN se transcribirá en ARN.

Al igual que la ADN polimerasa, la ARN polimerasa solo puede sintetizar ácido nucleico en una dirección 5' a 3' mientras “lee” un molde de ADN en la dirección 3' a 5'. Como se mencionó anteriormente en esta unidad, el extremo 3' de una cadena de ácido nucleico tiene un grupo hidroxilo (OH) en el carbono 3' de la desoxirribosa o ribosa y no está unido a otro nucleótido. El extremo 5' de esa cadena tiene un grupo fosfato unido al carbono 5' del azúcar y no está unido a otro nucleótido (Figura\(\PageIndex{1}\)).
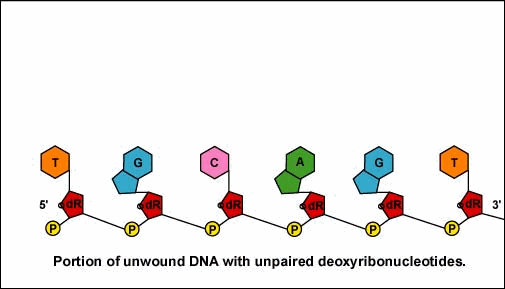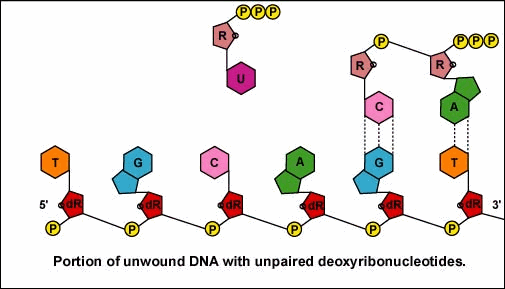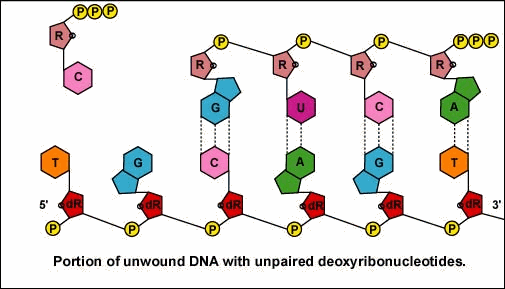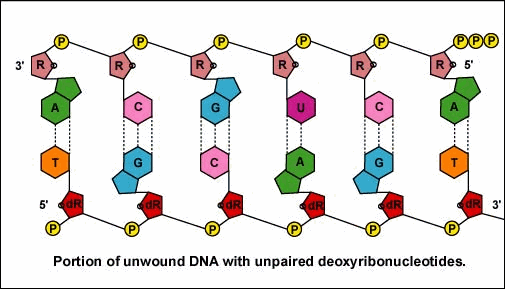
Una vez que el complejo ARN polimerasa/factor sigma reconoce el promotor correcto, el factor sigma se disocia de la ARN polimerasa y la enzima comienza a desenrollar la hélice del ADN creando una región de desoxirribonucleótidos no apareados que sirven como molde para la síntesis de ARN (Figuras 2 y 3).

Desenrollamiento de la hélice de ADN por ARN polimerasa
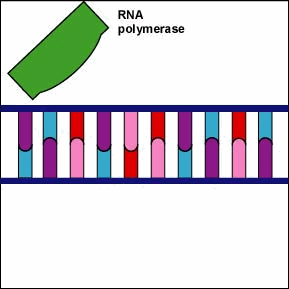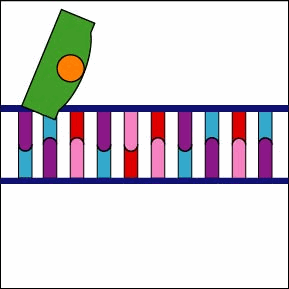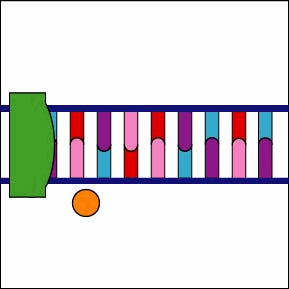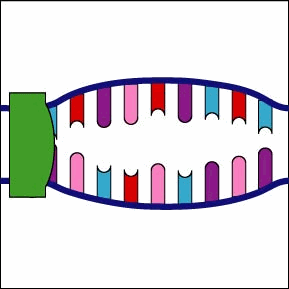
Una vez que el complejo ARN polimerasa/factor sigma reconoce el promotor correcto, el factor sigma se disocia de la ARN polimerasa y la enzima comienza a desenrollar la hélice del ADN creando una región de desoxirribonucleótidos no apareados que sirven como molde para la síntesis de ARN
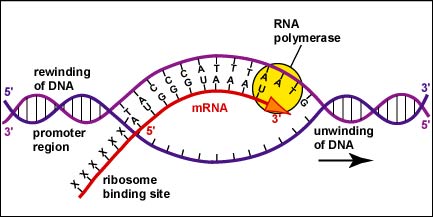
Una vez que comienza la transcripción real, los ribonucleótidos que contienen 3 grupos fosfato se unen hidrógeno a través del proceso de apareamiento de bases complementarias con los desoxirribonucleótidos expuestos en la cadena desenrollada que se va a transcribir (Figura\(\PageIndex{4}\)). Los ribonucleótidos se unen entonces covalentemente entre sí mediante enlaces fosfodiéster, siendo la energía suministrada por la escisión de dos grupos fosfato del ribonucleótido trifosfato (Figura\(\PageIndex{5}\)). (El enlace fosfodiéster se refiere al fosfato en el 5'C del nucleótido recién insertado que se une covalentemente al 3'C del último ribonucleótido en la cadena de ARNm). El ARNm polimeriza a una velocidad de aproximadamente 30 nucleótidos por segundo.

A medida que la ARN polimerasa se mueve hacia abajo por el ADN, el tramo anterior de ADN nuevamente se empareja con su hebra complementaria. Este proceso continúa hasta que la ARN polimerasa encuentra una señal de “parada” o terminador de la transcripción al final del gen. Esto hace que el ARNm completo caiga del gen.
Transcripción de ARNm complementario al ADN
Una vez que el complejo ARN polimerasa/factor sigma reconoce el promotor correcto, el factor sigma se disocia de la ARN polimerasa y la enzima comienza a desenrollar la hélice del ADN creando una región de desoxirribonucleótidos no apareados que sirven como molde para la síntesis de ARN. La transcripción está bajo el control de la enzima ARN polimerasa que no se muestra aquí.
Una vez que la ARN polimerasa se mueve más allá de la región promotora, una nueva molécula de ARN polimerasa puede unirse al promotor e iniciar una nueva ronda de transcripción. De esta manera, un solo gen se puede transcribir múltiples veces.
|
Película de YouTube que ilustra el emparejamiento de bases complementarias durante la transcripción.
|
|
Película de YouTube que ilustra la transcripción y el ensamblaje de proteínas.
|
|
Película de YouTube que ilustra la transcripción en bacterias
|
La transcripción se resume en las Figs. 6 y 7.
Hay 22 aminoácidos que pueden ser codificados por la información genética transportada sobre el ARNm. La molécula de ARNm se divide en codones. Un codón es una serie de tres bases de ARNm consecutivas que codifican un aminoácido específico. Los diversos codones y los aminoácidos para los que codifican se muestran en la Tabla\(\PageIndex{16}\) .8.1. Hay 64 codones. Un codón, AUG, también sirve como codón de inicio para iniciar la traducción, y tres codones, UAG, UAA y UGA, funcionan como codones de parada o sin sentido para terminar la traducción. (Los codones de inicio alternativos son diferentes del codón AUG estándar y ocasionalmente se encuentran tanto en procariotas como en eucariotas).
|
U
|
C
|
A
|
G
|
||
|---|---|---|---|---|---|
|
U
|
UUU = Phe UUC = Phe UUA = Leu rumano = Leu rumano |
UCU = Ser UCC = Ser UCA = Ser UCG = Ser |
UAU = Tyr UAC = Tyr UAA = Parar UAG = Parar |
UGU = Cys UGC = Cys UGA = Parar UGG = Trp |
U C A G |
|
C
|
CUU = Leu cuc = Leu rumano CUA = Leu rumano CUG = Leu rumano |
CCU = CCC Pro = Pro CCA = Pro CCG = Pro |
CAU = Su CAC = Su CAA = Gln CAG = Gln |
CGU = Arg CGC = Arg CGA = Arg CGG = Arg |
U C A G |
|
A
|
AUU = Ile AUC = Ile AUA = Ile AUG = Met |
ACU = Thr ACC = Thr ACA = Thr ACG = Thr |
AAU = Asn AAC = Asn AAA = Lys AAG = Lys |
AGU = Ser AGC = Ser AGA = Arg AGG = Arg |
U C A G |
|
G
|
GUU = Val CUC = Val GUA = Val GUG = Val |
CCG = Ala CCG = Ala GCA = Ala GCG = Ala |
GAU = Asp GAC = Asp GAA = Glu GAG = Glu |
GGU = Gly GGC = Gly GGA = Gly GGG = Gly |
U C A G |
| Phe = fenilalanina Leu = leucina Ile = isoleucina Met = metionina Val = valina |
Ser = serina Pro = prolina Thr = treonina Ala = alanina Tyr = tirosina |
His = histidina Gln = glutamina Asn = asparagina Lys = lisina Asp = ácido aspártico |
Glu = ácido glutámico Cys = cisteína Trp = triptófano Arg = arginina Gly = glicina |
||
| AUG = codón de inicio, UAA, UAG y UGA = codones de parada (sin sentido) | |||||
En bacterias, un ARNm puede ser monocistrónico o policistrónico. Un ARNm monocistrónico es una transcripción de un solo gen. Un ARNm policistrónico porta una transcripción de múltiples genes, a menudo involucrados en una sola vía bioquímica. Los grupos de genes relacionados que se transcriben juntos para formar un ARNm policistrónico se conocen como operones. También hay genes específicos a lo largo del ADN a partir de los cuales se transcriben cada uno de los diferentes ARN de transferencia (ARNt) y los ARN ribosómicos (ARNr).
La mayoría de los ARNm en procariotas tienen una vida media del orden de unos pocos minutos. Las moléculas de ARNr y ARNt, por otro lado, son mucho más estabiles. Debido a que el ARNr y el ARNt son moléculas altamente plegadas, a diferencia del ARNm, son mucho más resistentes a la degradación por ribonucleasas. Una vez transcrito, el ARNm puede traducirse en proteína.
Transcripción en células eucariotas
La transcripción es más compleja en las células eucariotas que en las que son procariotas. Las proteínas activadoras se unen a genes conocidos como potenciadores que ayudan a determinar qué genes están activados y aceleran la transcripción. Las proteínas represoras se unen a genes llamados silenciadores que interfieren con las proteínas activadoras y ralentizan la transcripción. Los coactivadores, moléculas adaptadoras que coordinan señales de proteínas activadoras y represivas, transmiten esta información a factores basales que luego posicionan a la ARN polimerasa al inicio de la región codificante del gen para comenzar la transcripción.
Una vez que comienza la transcripción real, los ribonucleótidos que contienen 3 grupos fosfato forman enlaces de hidrógeno a través del proceso de emparejamiento de bases complementarias con los desoxirribonucleótidos expuestos en la cadena desenrollada que se va a transcribir. Los ribonucleótidos se unen entonces covalentemente entre sí mediante enlaces fosfodiéster, siendo la energía suministrada por la escisión de dos grupos fosfato del ribonucleótido trifosfato (Figura\(\PageIndex{16}\) .8B.5). (El enlace fosfodiéster se refiere al fosfato en el 5'C del nucleótido recién insertado que se une covalentemente al 3'C del último ribonucleótido en la cadena de ARNm).

A diferencia de los procariotas, la mayoría de los genes en las células eucariotas superiores contienen grandes cantidades -tanto como 98% en el genoma humano- de regiones llamadas intrones que no forman parte del código para la proteína final. Estos se intercalan entre las regiones codificantes o exones que realmente codifican para la proteína final.
La ARN polimerasa copia tanto los exones como los intrones para formar lo que se llama ARNm precursor o pre-ARNm. Al principio de la transcripción, se agrega una tapa en forma de un nucleótido inusual, 7-metilguanilato, al extremo 5' del pre-ARNm. Esta tapa ayuda a que los ribosomas se adhieran para su traducción. A medida que la transcripción está casi terminada, se agrega una serie de 100-250 ribonucleótidos de adenina llamada cola poli-A al extremo 3' del pre-ARNm. Se cree que esta cola poli-A ayuda a transportar el ARNm fuera del núcleo y puede estabilizar el ARNm contra la degradación en el citoplasma. Después de la transcripción del ARNm precursor, las regiones codificantes no proteicas (intrones) se escinden y las regiones codificantes (exones) se unen entre sí por complejos de ribonucleoproteínas llamados espliceosomas para producir lo que se denomina ARNm maduro como se muestra en la Figura\(\PageIndex{9}\). Este proceso se llama procesamiento de ARN.

|
Película de YouTube que ilustra el emparejamiento de bases complementarias durante la transcripción.
|
|
Película de YouTube que ilustra la transcripción y el ensamblaje de proteínas.
|
|
Película de YouTube que ilustra el procesamiento de ARN en células eucariotas #1.
|
|
Película de YouTube que ilustra el procesamiento de ARN en células eucariotas #2.
|
Luego, el ARNm maduro pasa a través de los poros de la membrana nuclear para ser traducido en proteína por el ARNt en ribosomas eucariotas 80S (compuestos por subunidades 60S y 40S) de manera similar a los procariotas.
La molécula de ARNm se divide en codones. Un codón es una serie de tres bases de ARNm consecutivas que codifican un aminoácido específico. Los diversos codones y los aminoácidos para los que codifican se muestran en la Figura\(\PageIndex{8}\). Hay 64 codones. Un codón, AUG, también sirve como codón de inicio para iniciar la traducción, y tres codones, UAG, UAA y UGA, funcionan como codones de parada o sin sentido para terminar la traducción. (Los codones de inicio alternativos son diferentes del codón AUG estándar y ocasionalmente se encuentran tanto en procariotas como en eucariotas).
Además de los genes que se transcriben en ARNm para traducirse en polipéptidos y proteínas, también hay genes específicos en el ADN a partir de los cuales se transcriben cada uno de los diferentes ARN de transferencia (ARNt) y los ARN ribosómicos (ARNs).
Una vez transcrito, el ARNm puede traducirse en proteína.
Como se mencionó anteriormente, los intrones constituyen la mayor parte del ADN en las células eucariotas superiores y durante décadas se consideró como “ADN basura” acumulado a lo largo de millones de años de evolución. Sin embargo, en los últimos años se ha descubierto que gran parte de este ADN intergénico, aunque no codifica para la síntesis de proteínas, se transcribe en moléculas funcionales de ARN con nombres como microARN antisentido, y ARN riboswitch que desempeñan papeles importantes en si realmente se elabora o no una proteína.
El ARN antisentido es ARN transcrito de la cadena de ADN complementaria a la que se transcribe en ARNm. En otras palabras, es una molécula de ARN complementaria a un ARNm y como tal puede emparejarse bases complementarias con el ARNm e impedir que se traduzca en proteína.
El microARN, a menudo transcrito a partir del ADN intrón, se pliega sobre sí mismo para parecerse al ARN bicatenario, una forma de ARN producida por muchos virus durante su ciclo de vida. El ARN bicatenario viral activa un mecanismo de defensa del huésped que degrada ese ARN viral. El microARN se une frecuentemente al ARNm y engaña este mecanismo de defensa para degradar ese ARNm para que no pueda traducirse en proteína.
El ARN riboswitch, a menudo transcrito a partir de intrones, existe en una forma inactiva hasta que se une una sustancia química diana específica. La unión de la sustancia química diana convierte el ARN riboswitch en una forma activa que puede traducirse en una proteína específica.
Resumen
- Durante la síntesis de proteínas, el orden de las bases nucleotídicas a lo largo de un gen se transcribe en una cadena complementaria de ARNm que luego se traduce por ARNt en el orden correcto de aminoácidos para ese polipéptido o proteína.
- El orden de las bases de desoxirribonucleótidos a lo largo del ADN determina el orden de los aminoácidos en las proteínas, es decir, su estructura primaria.
- Debido a que ciertos aminoácidos pueden interactuar con otros aminoácidos, el orden de los aminoácidos para cada proteína determina su forma tridimensional final, que a su vez determina la función de esa proteína.
- El ARN mensajero (ARNm) se sintetiza mediante el apareamiento de bases complementarias de ribonucleótidos con desoxirribonucleótidos para que coincidan con una porción de una cadena de ADN llamada gen.
- Aunque los genes están presentes en ambas cadenas de ADN, solo se transcribe una cadena para cualquier gen dado.
- La enzima ARN polimerasa transcribe ADN.
- Para iniciar la transcripción en bacterias, una variedad de proteínas llamadas factores sigma se unen a las ARN polimerasas. Este complejo puede entonces unirse a una secuencia de ADN específica llamada promotor localizado a lo largo del ADN antes de la región codificante del gen. El promotor determina qué región del ADN y qué cadena de ADN se transcribirá en ARN.
- Al igual que la ADN polimerasa, la ARN polimerasa solo puede sintetizar ácido nucleico en una dirección 5' a 3' mientras “lee” un molde de ADN en la dirección 3' a 5'.
- Una vez que el complejo ARN polimerasa/factor sigma reconoce el promotor correcto, el factor sigma se disocia de la ARN polimerasa y la enzima comienza a desenrollar la hélice del ADN creando una región de desoxirribonucleótidos no apareados que sirven como molde para la síntesis de ARN.
- Durante la transcripción, los ribonucleótidos se unen hidrógeno a través del proceso de emparejamiento de bases complementarias con los desoxirribonucleótidos expuestos en la cadena desenrollada que se va a transcribir. Los ribonucleótidos se unen entonces covalentemente entre sí mediante enlaces fosfodiéster.
- Este proceso continúa hasta que la ARN polimerasa encuentra una señal de “parada” o terminador de la transcripción al final del gen.
- Un solo gen se puede transcribir varias veces.
- La molécula de ARNm se divide en codones. Un codón es una serie de tres bases de ARNm consecutivas que codifican un aminoácido específico.
- Tres codones, UAG, UAA y UGA, funcionan como codones de parada o sin sentido para terminar la traducción.
- En bacterias, un ARNm puede ser monocistrónico o policistrónico. Un ARNm monocistrónico es un transcrito de un solo gen; un ARNm policistrónico porta un transcrito de múltiples genes, a menudo involucrados en una sola vía bioquímica. Una vez transcrito, el ARNm puede traducirse en proteína por ARNt en ribosomas 70S (compuesto por subunidades 50S y 30S).
- La transcripción es más compleja en las células eucariotas que en las que son procariotas. Las proteínas activadoras se unen a genes conocidos como potenciadores que ayudan a determinar qué genes están activados y aceleran la transcripción. Las proteínas represoras se unen a genes llamados silenciadores que interfieren con las proteínas activadoras y ralentizan la transcripción. Los coactivadores, moléculas adaptadoras que coordinan señales de proteínas activadoras y represivas, transmiten esta información a factores basales que luego posicionan a la ARN polimerasa al inicio de la región codificante del gen para comenzar la transcripción.
- La mayoría de los genes en las células eucariotas superiores contienen regiones llamadas intrones que no forman parte del código de la proteína final. Estos se intercalan entre las regiones codificantes o exones que realmente codifican para la proteína final.
- Después de la transcripción del ARNm precursor, las regiones codificantes no proteicas (intrones) se escinden y las regiones codificantes (exones) se unen entre sí por complejos de ribonucleoproteínas llamados espliceosomas para producir lo que se denomina ARNm maduro.
- Luego, el ARNm maduro pasa a través de los poros de la membrana nuclear para ser traducido en proteína por el ARNt en los ribosomas 80S (compuestos por subunidades 60S y 40S) de manera similar a los procariotas.


