12.1: Los microbios y las herramientas de la ingeniería genética
- Page ID
- 54890
Objetivos de aprendizaje
- Identificar herramientas de genética molecular que se derivan de microorganismos
- Describir los métodos utilizados para crear moléculas de ADN recombinante
- Describir los métodos utilizados para introducir ADN en células procariotas
- Enumerar los tipos de bibliotecas genómicas y describir sus usos
- Describir los métodos utilizados para introducir ADN en células eucariotas
Enfoque Clínico: Parte 1
Kayla, ingeniera eléctrica de 24 años y entusiasta del funcionamiento, acaba de mudarse de Arizona a New Hampshire para tomar un nuevo trabajo. En sus fines de semana libres, le encanta explorar su nuevo entorno, dando largas carreras por los bosques de pinos. En julio pasó una semana caminando por las montañas. A principios de agosto, Kayla desarrolló fiebre baja, dolor de cabeza y dolores musculares leves, y se sintió un poco fatigada. Sin pensarlo mucho, tomó un poco de ibuprofeno para combatir sus síntomas y se comprometió a descansar más.
Ejercicio\(\PageIndex{1}\)
¿Qué tipos de afecciones médicas podrían ser responsables de los síntomas de Kayla?
La ciencia del uso de los sistemas vivos para beneficiar a la humanidad se llama biotecnología. Técnicamente hablando, la domesticación de plantas y animales a través de prácticas agrícolas y reproductivas es un tipo de biotecnología. Sin embargo, en un sentido contemporáneo, asociamos la biotecnología con la alteración directa de la genética de un organismo para lograr rasgos deseables a través del proceso de ingeniería genética. La ingeniería genética implica el uso de la tecnología de ADN recombinante, proceso mediante el cual una secuencia de ADN es manipulada in vitro, creando así moléculas de ADN recombinante que tienen nuevas combinaciones de material genético. El ADN recombinante se introduce luego en un organismo huésped. Si el ADN que se introduce proviene de una especie diferente, el organismo huésped ahora se considera transgénico.
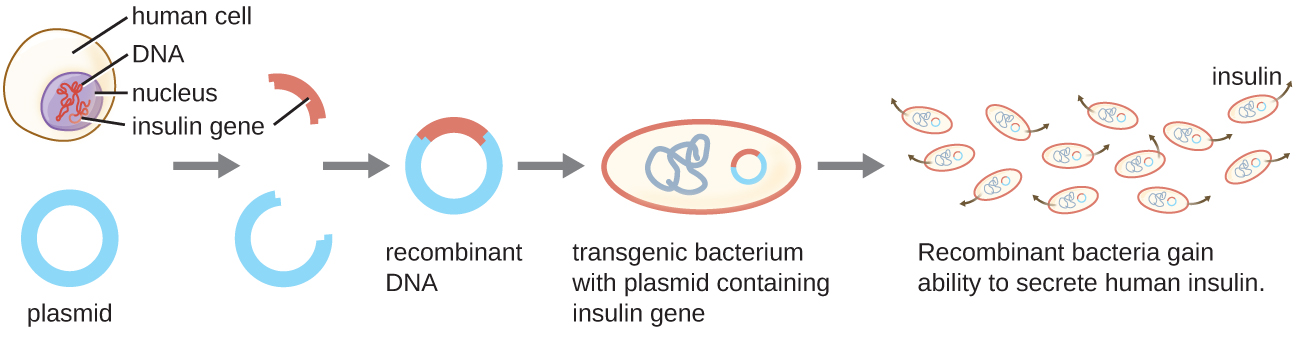
Un ejemplo de un microorganismo transgénico es la cepa bacteriana que produce insulina humana (Figura\(\PageIndex{1}\)). El gen de insulina de humanos se insertó en un plásmido. Este plásmido de ADN recombinante se insertó luego en bacterias. Como resultado, estos microbios transgénicos son capaces de producir y secretar insulina humana. Muchos procariotas son capaces de adquirir ADN extraño e incorporar genes funcionales en su propio genoma a través del “apareamiento” con otras células (conjugación), infección viral (transducción) y tomar ADN del ambiente (transformación). Recordemos que estos mecanismos son ejemplos de transferencia génica horizontal, la transferencia de material genético entre células de la misma generación.
Clonación Molecular
Herbert Boyer y Stanley Cohen demostraron por primera vez el proceso completo de clonación molecular en 1973 cuando clonaron con éxito genes de la rana africana (Xenopus laevis) en un plásmido bacteriano que luego se introdujo en el huésped bacteriano Escherichia coli. La clonación molecular es un conjunto de métodos utilizados para construir ADN recombinante e incorporarlo a un organismo huésped; hace uso de una serie de herramientas moleculares que se derivan de microorganismos.
Enzimas de restricción y ligasas
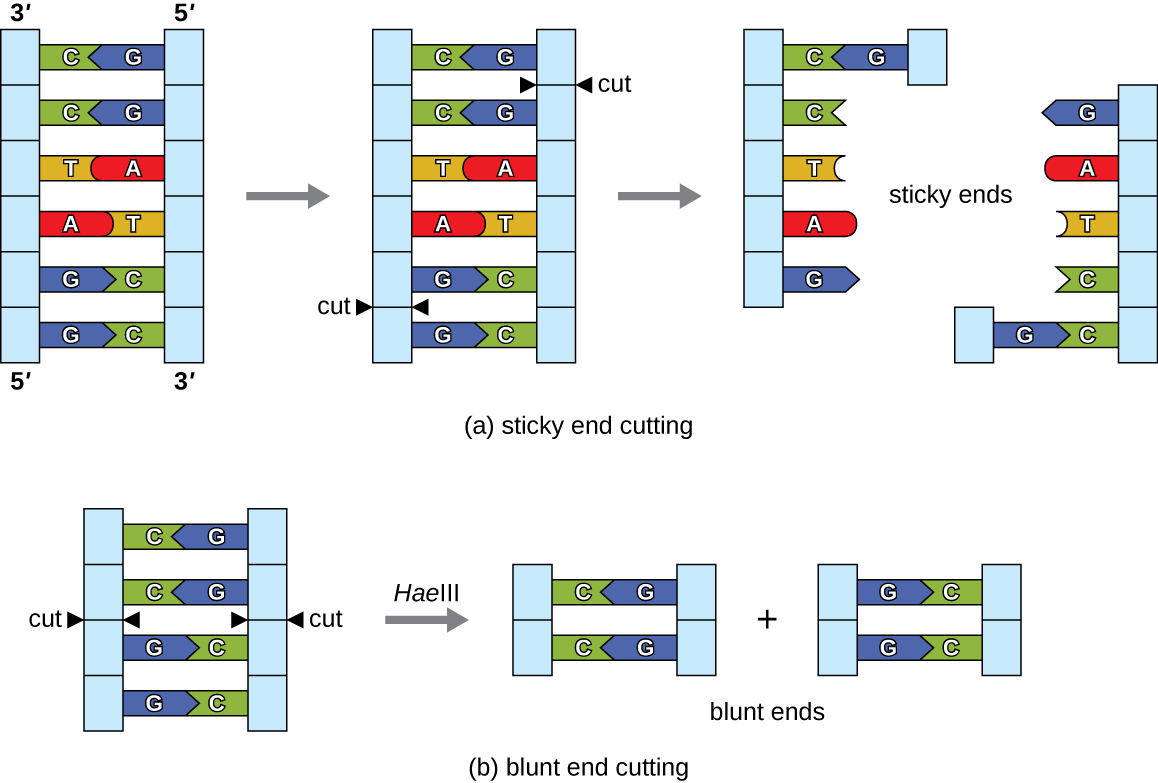
En la tecnología de ADN recombinante, las moléculas de ADN se manipulan utilizando enzimas naturales derivadas principalmente de bacterias y virus. La creación de moléculas de ADN recombinante es posible debido al uso de endonucleasas de restricción naturales (enzimas de restricción), enzimas bacterianas producidas como mecanismo de protección para cortar y destruir el ADN citoplásmico extraño que más comúnmente es resultado de la infección por bacteriófagos. Stewart Linn y Werner Arber descubrieron enzimas de restricción en sus estudios de la década de 1960 sobre cómo E. coli limita la replicación de bacteriófagos en la infección. Hoy en día, utilizamos enzimas de restricción extensamente para cortar fragmentos de ADN que luego se pueden empalmar en otra molécula de ADN para formar moléculas recombinantes. Cada enzima de restricción corta el ADN en un sitio de reconocimiento característico, una secuencia de ADN específica, generalmente palindrómica, típicamente entre cuatro y seis pares de bases de longitud. Un palíndromo es una secuencia de letras que lee lo mismo hacia adelante que hacia atrás. (La palabra “nivel” es un ejemplo de un palíndromo.) Las secuencias de ADN palindrómico contienen las mismas secuencias de bases en la dirección de 5 a 3 en una cadena que en la dirección de 5 a 3 en la cadena complementaria. Una enzima de restricción reconoce el palíndromo de ADN y corta cada cadena principal en posiciones idénticas en el palíndromo. Algunas enzimas de restricción cortan para producir moléculas que tienen salientes complementarios (extremos pegajosos) mientras que otras cortan sin generar tales salientes, en lugar de producir extremos romos (Figura\(\PageIndex{2}\)).
Las moléculas con extremos pegajosos complementarios pueden hibridarse fácilmente, o formar enlaces de hidrógeno entre bases complementarias, en sus extremos pegajosos. La etapa de hibridación permite la hibridación de los salientes monocatenarios. La hibridación se refiere a la unión de dos cadenas individuales complementarias de ADN. Los extremos romos también se pueden unir, pero de manera menos eficiente que los extremos pegajosos debido a la falta de voladizos complementarios que facilitan el proceso. En cualquier caso, la ligación por ADN ligasa puede entonces reunirse a las dos cadenas principales de azúcar-fosfato del ADN a través de enlaces covalentes, haciendo de la molécula una doble cadena continua. En 1972, Paul Berg, bioquímico de Stanford, fue el primero en producir una molécula de ADN recombinante utilizando esta técnica, combinando el virus del mono SV40 con el bacteriófago lambda de E. coli para crear un híbrido.
Plásmidos
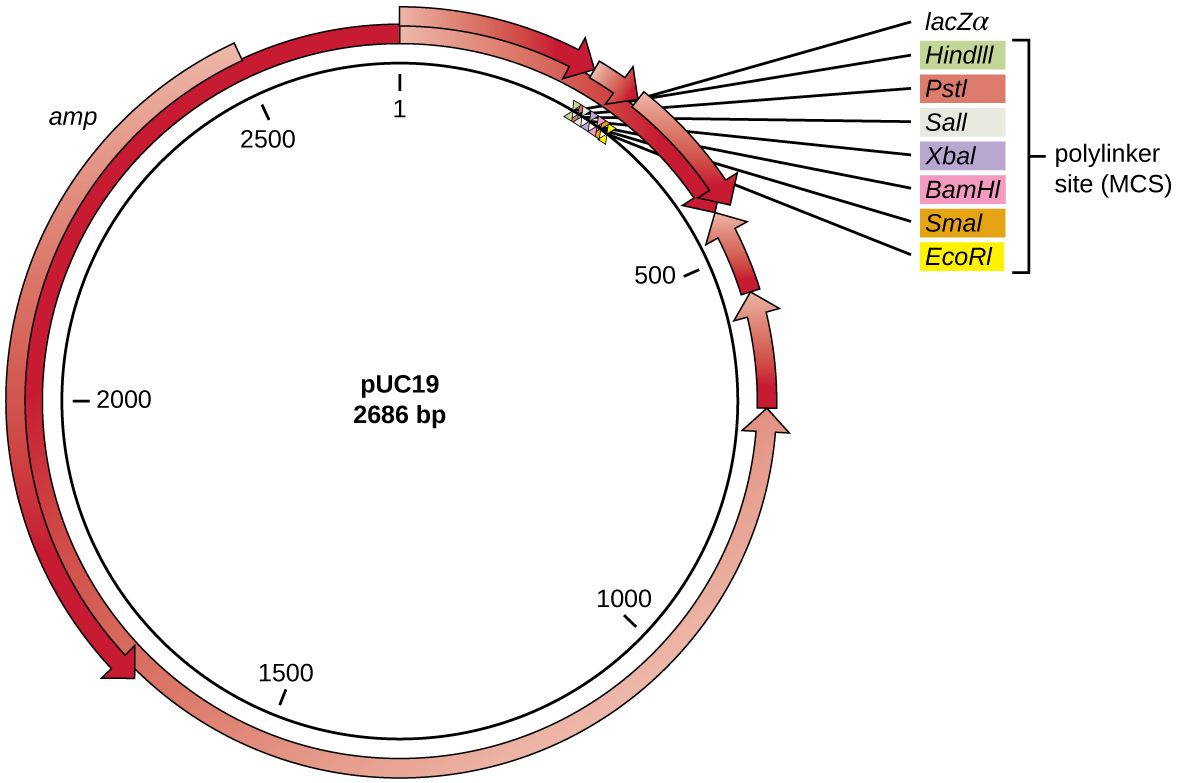
Después de la digestión de restricción, los genes de interés se insertan comúnmente en plásmidos, pequeños trozos de ADN bicatenario típicamente circular que se replican independientemente del cromosoma bacteriano (ver Características únicas de las células procariotas). En la tecnología de ADN recombinante, a menudo se utilizan plásmidos como vectores, moléculas de ADN que transportan fragmentos de ADN de un organismo a otro. Los plásmidos utilizados como vectores pueden ser modificados genéticamente por investigadores y empresas de suministro científico para que tengan propiedades especializadas, como lo ilustra el vector plasmídico de uso común pUC19 (Figura\(\PageIndex{3}\)). Algunos vectores plasmídicos contienen genes que confieren resistencia a antibióticos; estos genes de resistencia permiten a los investigadores encontrar fácilmente colonias que contienen plásmidos sembrándolas en placas en medios que contienen el antibiótico correspondiente. El antibiótico mata todas las células hospedadoras que no albergan el vector plasmídico deseado, pero las que contienen el vector son capaces de sobrevivir y crecer.
Los vectores plasmídicos utilizados para la clonación tienen típicamente un sitio polienlazador, o un sitio de clonación múltiple (MCS). Un sitio polienlazador es una secuencia corta que contiene múltiples sitios de reconocimiento de enzimas de restricción únicos que se usan para insertar ADN en el plásmido después de la digestión de restricción tanto del ADN como del plásmido. Tener estos múltiples sitios de reconocimiento de enzimas de restricción dentro del sitio polienlazador hace que el vector plasmídico sea versátil, por lo que puede usarse para muchos experimentos de clonación diferentes que involucran diferentes enzimas de restricción.
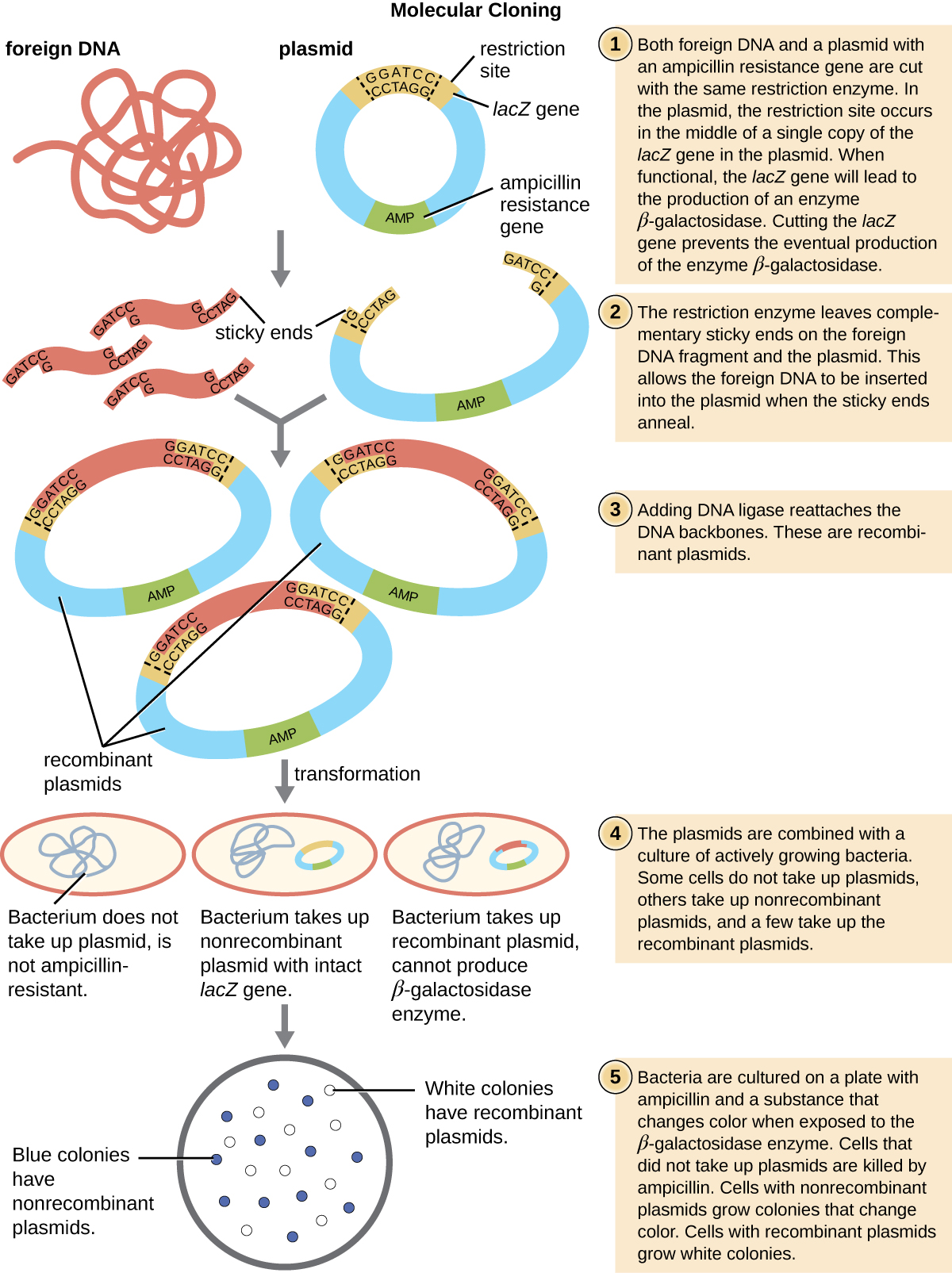
Este sitio polienlazador se encuentra a menudo dentro de un gen informador, otra secuencia génica modificada artificialmente en el plásmido que codifica una proteína que permite la visualización de la inserción de ADN. El gen informador permite a un investigador distinguir células hospedadoras que contienen plásmidos recombinantes con fragmentos de ADN clonados de células hospedadoras que solo contienen el vector plasmídico no recombinante. El gen indicador más común utilizado en los vectores plasmídicos es el gen lacZ bacteriano que codifica la beta-galactosidasa, una enzima que degrada naturalmente la lactosa pero que también puede degradar un análogo sintético incoloro X-gal, produciendo colonias azules en medios que contienen X-gal. El gen informador lacZ se inhabilita cuando el ADN recombinante se empalma en el plásmido. Debido a que la proteína lacZ no se produce cuando el gen está deshabilitado, X-gal no se degrada y se producen colonias blancas, que luego pueden aislarse. Este método de cribado azul-blanco se describe más adelante y se muestra en la Figura\(\PageIndex{4}\). Además de estas características, algunos plásmidos vienen predigeridos y con una enzima ligada al plásmido linealizado para ayudar en la ligación después de la inserción de fragmentos de ADN extraños.
Clonación molecular usando transformación
El mecanismo más utilizado para introducir plásmidos diseñados en una célula bacteriana es la transformación, un proceso en el que las bacterias toman el ADN libre de sus alrededores. En la naturaleza, el ADN libre generalmente proviene de otras células bacterianas lisadas; en el laboratorio, el ADN libre en forma de plásmidos recombinantes se introduce en el entorno de la célula.
Algunas bacterias, como Bacillus spp., son naturalmente competentes, lo que significa que son capaces de tomar ADN extraño. Sin embargo, no todas las bacterias son naturalmente competentes. En la mayoría de los casos, las bacterias deben hacerse artificialmente competentes en el laboratorio aumentando la permeabilidad de la membrana celular. Esto se puede lograr a través de tratamientos químicos que neutralizan las cargas en la membrana celular o mediante la exposición de las bacterias a un campo eléctrico que crea poros microscópicos en la membrana celular. Estos métodos producen bacterias químicamente competentes o electrocompetentes, respectivamente.
Siguiendo el protocolo de transformación, las células bacterianas se plaquean en un medio que contiene antibióticos para inhibir el crecimiento de las muchas células hospedadoras que no fueron transformadas por el plásmido que confiere resistencia a antibióticos. Luego se usa una técnica llamada cribado azul-blanco para vectores plasmídicos que codifican lacZ como pUC19. Las colonias azules tienen una enzima beta-galactosidasa funcional debido a que el gen lacZ es ininterrumpido, sin ADN extraño insertado en el sitio polienlazador. Estas colonias son típicamente el resultado del plásmido linealizado digerido que se religó consigo mismo. Sin embargo, las colonias blancas carecen de una enzima beta-galactosidasa funcional, lo que indica la inserción de ADN extraño dentro del sitio polienlazador del vector plasmídico, alterando así el gen lacZ. Por lo tanto, las colonias blancas resultantes de este cribado azul-blanco contienen plásmidos con un inserto y pueden cribarse adicionalmente para caracterizar el ADN extraño. Para asegurarse de que el ADN correcto se incorporó al plásmido, el inserto de ADN puede entonces ser secuenciado.
Visualizar una animación de clonación molecular desde el Centro de Aprendizaje de ADN.
Ejercicio\(\PageIndex{2}\)
En el cribado azul-blanco, ¿qué significa una colonia azul y por qué es azul?
Clonación molecular usando conjugación o transducción
El proceso bacteriano de conjugación (ver Cómo los procariotas asexuales logran la diversidad genética) también se puede manipular para la clonación molecular. Los plásmidos F, o plásmidos de fertilidad, se transfieren entre células bacterianas a través del proceso de conjugación. El ADN recombinante puede transferirse por conjugación cuando las células bacterianas que contienen un plásmido F recombinante se mezclan con células bacterianas compatibles que carecen del plásmido. Los plásmidos F codifican una estructura superficial llamada pilus F que facilita el contacto entre una célula que contiene un plásmido F y otra sin un plásmido F. Al contacto, se forma un puente citoplásmico entre las dos células y la célula que contiene el plásmido F replica su plásmido, transfiriendo una copia del plásmido F recombinante a la célula receptora. Una vez que ha recibido el plásmido F recombinante, la célula receptora puede producir su propio pilus F y facilitar la transferencia del plásmido F recombinante a una célula adicional. El uso de conjugación para transferir plásmidos F recombinantes a células receptoras es otra manera efectiva de introducir moléculas de ADN recombinante en células hospedadoras.
Alternativamente, se pueden usar bacteriófagos para introducir ADN recombinante en células bacterianas hospedadoras a través de una manipulación del proceso de transducción (ver Cómo los procariotas asexuales logran la diversidad genética). En el laboratorio, los fragmentos de ADN de interés pueden ser modificados por ingeniería genética en fagémidos, que son plásmidos que tienen secuencias de fagos que les permiten ser empaquetados en bacteriófagos. Las células bacterianas pueden entonces infectarse con estos bacteriófagos para que los fagémidos recombinantes puedan introducirse en las células bacterianas. Dependiendo del tipo de fago, el ADN recombinante puede integrarse en el genoma bacteriano del huésped (lisogenia), o puede existir como un plásmido en el citoplasma del huésped.
Ejercicio\(\PageIndex{2}\)
- ¿Cuál es la función original de una enzima de restricción?
- ¿Qué dos procesos se explotan para obtener ADN recombinante en una célula hospedadora bacteriana?
- Distinguir los usos de un gen de resistencia a antibióticos y un gen informador en un vector plasmídico.
Creación de una biblioteca genómica
La clonación molecular también se puede usar para generar una biblioteca genómica. La biblioteca es una copia completa (o casi completa) del genoma de un organismo contenido como plásmidos de ADN recombinante diseñados en clones únicos de bacterias. Tener una biblioteca de este tipo permite a un investigador crear grandes cantidades de cada fragmento cultivando el hospedador bacteriano para ese fragmento. Estos fragmentos pueden ser utilizados para determinar la secuencia del ADN y la función de cualquier gen presente.
Un método para generar una biblioteca genómica es ligar fragmentos genómicos individuales digeridos con enzimas de restricción en vectores plasmídicos cortados con la misma enzima de restricción (Figura\(\PageIndex{5}\)). Después de la transformación en un huésped bacteriano, cada célula bacteriana transformada toma un único plásmido recombinante y crece en una colonia de células. Todas las células de esta colonia son clones idénticos y portan el mismo plásmido recombinante. La biblioteca resultante es una colección de colonias, cada una de las cuales contiene un fragmento del genoma del organismo original, que son cada una separada y distinta y cada una puede ser utilizada para su posterior estudio. Esto hace posible que los investigadores seleccionen estos diferentes clones para descubrir el que contiene un gen de interés del genoma del organismo original.
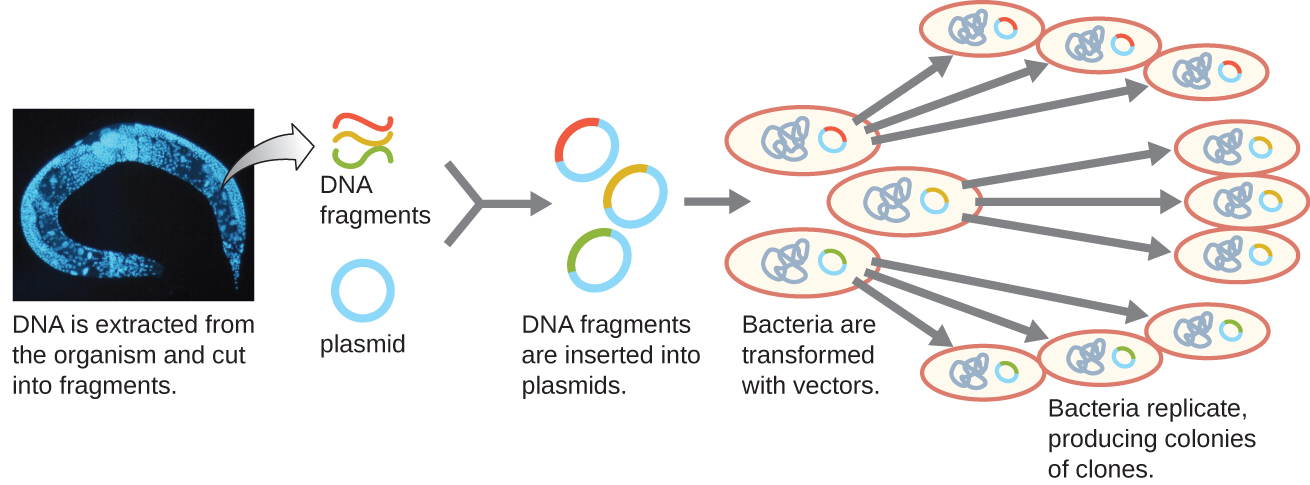
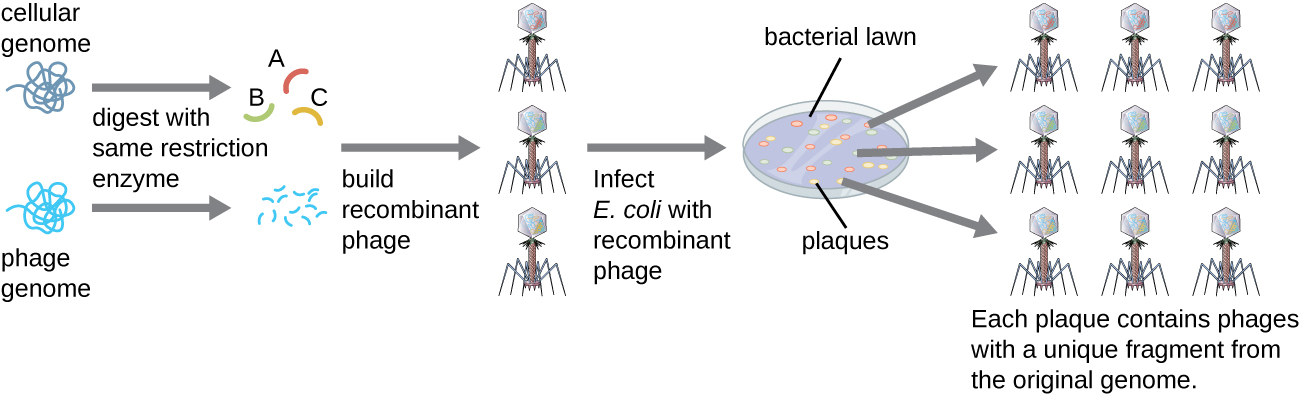
Para construir una biblioteca genómica usando fragmentos más grandes de ADN genómico, se puede usar como hospedador un bacteriófago de E. coli, tal como lambda (Figura\(\PageIndex{6}\)). El ADN genómico puede cortarse o digerirse enzimáticamente y ligarse en un vector de ADN de bacteriófago lambda predigerido. Entonces, estas moléculas de ADN de fago recombinante pueden empaquetarse en partículas de fago y usarse para infectar células hospedadoras de E. coli en una placa. Durante la infección dentro de cada célula, cada fago recombinante hará muchas copias de sí mismo y lisará el césped de E. coli, formando una placa. Así, cada placa de una biblioteca de fagos representa un fago recombinante único que contiene un fragmento de ADN genómico distinto. Las placas se pueden seguir cribando para buscar genes de interés. Una ventaja de producir una biblioteca usando fagos en lugar de plásmidos es que una partícula de fago contiene un inserto mucho más grande de ADN extraño en comparación con un vector plasmídico, requiriendo así un número mucho menor de cultivos para representar completamente el genoma completo del organismo original.
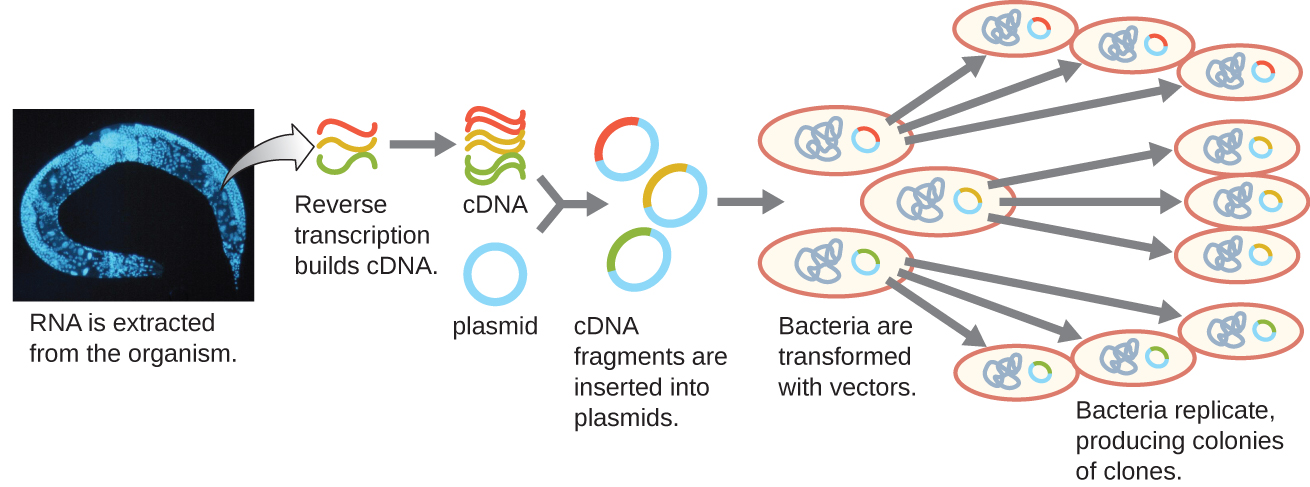
Para centrarse en los genes expresados en un organismo o incluso en un tejido, los investigadores construyen bibliotecas utilizando el ARN mensajero del organismo (ARNm) en lugar de su ADN genómico. Mientras que todas las células de un solo organismo tendrán el mismo ADN genómico, diferentes tejidos expresan diferentes genes, produciendo diferentes complementos de ARNm. Por ejemplo, el ADN genómico de todas las células humanas contiene el gen de la insulina, pero solo las células del páncreas expresan ARNm que dirige la producción de insulina. Debido a que el ARNm no puede clonarse directamente, en el laboratorio el ARNm debe ser utilizado como molde por la enzima retrovírica transcriptasa inversa para producir ADN complementario (ADNc). El complemento completo de ARNm de una célula se puede transcribir inversamente en moléculas de ADNc, las cuales se pueden usar como molde para la ADN polimerasa para hacer copias de ADN bicatenario; estos fragmentos se pueden ligar posteriormente en vectores plasmídicos o bacteriófagos para producir una biblioteca de ADNc. El beneficio de una biblioteca de ADNc es que contiene ADN de solo los genes expresados en la célula. Esto significa que los intrones, las secuencias de control como los promotores y el ADN no destinado a traducirse en proteínas no están representados en la biblioteca. El enfoque en las secuencias traducidas significa que la biblioteca no puede ser utilizada para estudiar la secuencia y estructura del genoma en su totalidad. La construcción de una biblioteca genómica de ADNc se muestra en la Figura\(\PageIndex{7}\).
Ejercicio\(\PageIndex{3}\)
- ¿Cuáles son los hospedadores para las bibliotecas genómicas descritas?
- ¿Qué es el ADNc?
Introducción de moléculas recombinantes en huéspedes eucariotas
El uso de hospedadores bacterianos para la ingeniería genética sentó las bases para la tecnología de ADN recombinante; sin embargo, los investigadores también han tenido un gran interés en la ingeniería genética de células eucariotas, particularmente las de plantas y animales. La introducción de moléculas de ADN recombinante en hospedadores eucariotas se denomina transfección. Las plantas genéticamente modificadas, llamadas plantas transgénicas, son de gran interés para fines agrícolas y farmacéuticos. La primera planta transgénica vendida comercialmente fue el tomate Flavr Savr de maduración retrasada, que salió al mercado en 1994. También se han producido con éxito ganado genéticamente manipulado, dando como resultado, por ejemplo, cerdos con mayor valor nutricional 1 y cabras que secretan productos farmacéuticos en su leche. 2
Electroporación
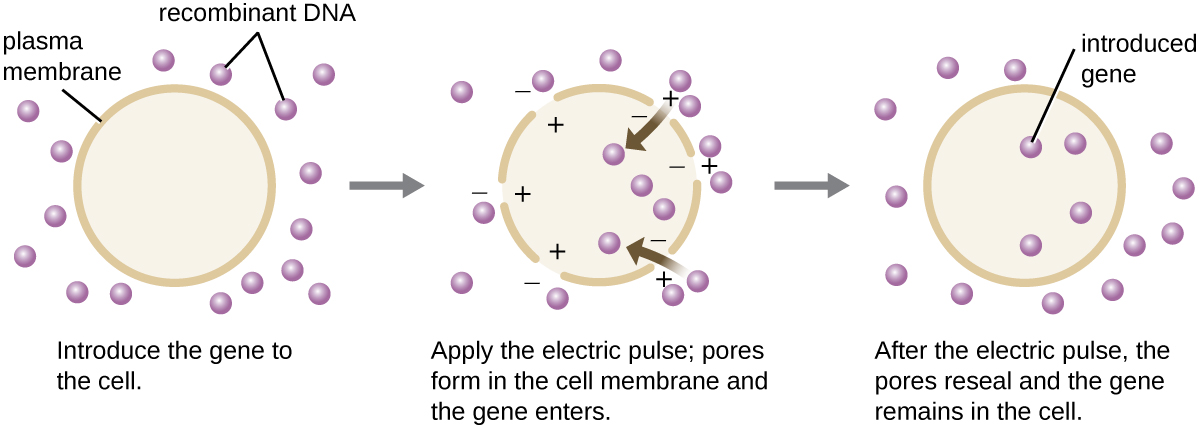
En comparación con las células bacterianas, las células eucariotas tienden a ser menos susceptibles como hospedadoras para moléculas de ADN recombinante. Debido a que los eucariotas normalmente no son competentes para tomar ADN extraño ni son capaces de mantener plásmidos, la transfección de huéspedes eucariotas es mucho más desafiante y requiere técnicas más intrusivas para el éxito. Un método utilizado para transfectar células en cultivo celular se llama electroporación. Un breve pulso eléctrico induce la formación de poros transitorios en las bicapas fosfolipídicas de las células a través de las cuales se puede introducir el gen. Al mismo tiempo, el pulso eléctrico genera una carga positiva de corta duración en un lado del interior de la célula y una carga negativa en el lado opuesto; la diferencia de carga atrae moléculas de ADN cargadas negativamente a la célula (Figura\(\PageIndex{8}\)).
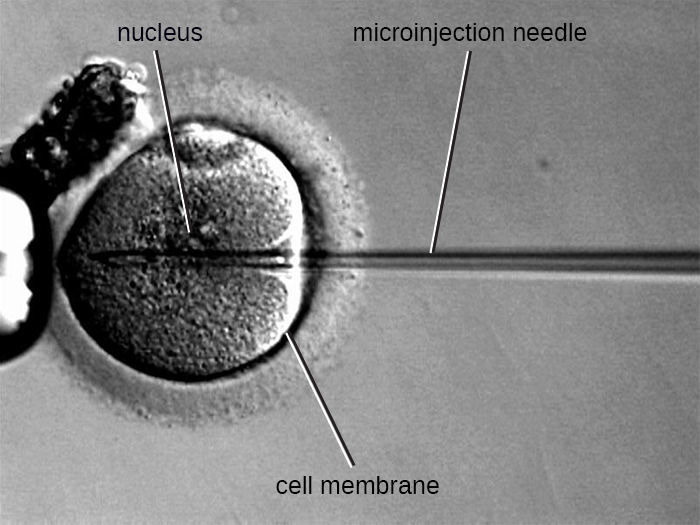
Microinyección
Un método alternativo de transfección se llama microinyección. Debido a que las células eucariotas son típicamente más grandes que las de los procariotas, a veces los fragmentos de ADN pueden inyectarse directamente en el citoplasma usando una micropipeta de vidrio, como se muestra en la Figura\(\PageIndex{9}\).
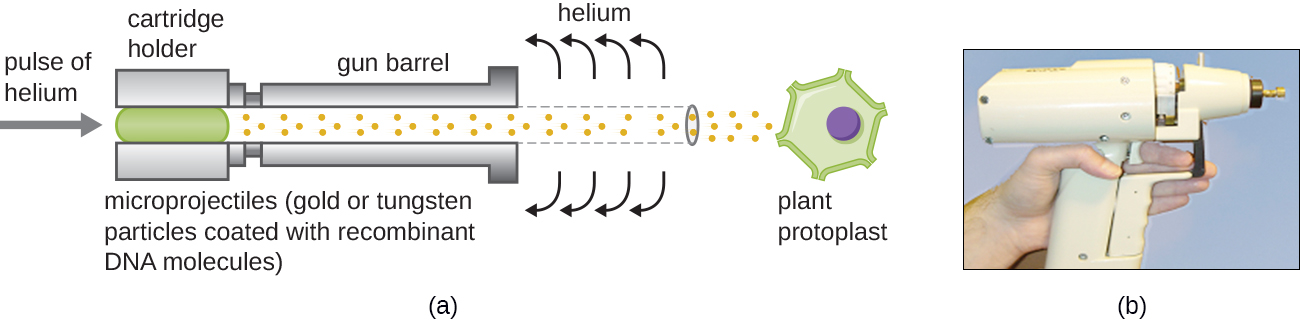
Pistolas génicas
Transfectar células vegetales puede ser incluso más difícil que las células animales debido a sus gruesas paredes celulares. Un enfoque consiste en tratar las células vegetales con enzimas para eliminar sus paredes celulares, produciendo protoplastos. Luego, se usa una pistola génica para disparar partículas de oro o tungsteno recubiertas con moléculas de ADN recombinante en los protoplastos de la planta a altas velocidades. Las células de protoplastos receptoras pueden entonces recuperarse y ser utilizadas para generar nuevas plantas transgénicas (Figura\(\PageIndex{10}\)).
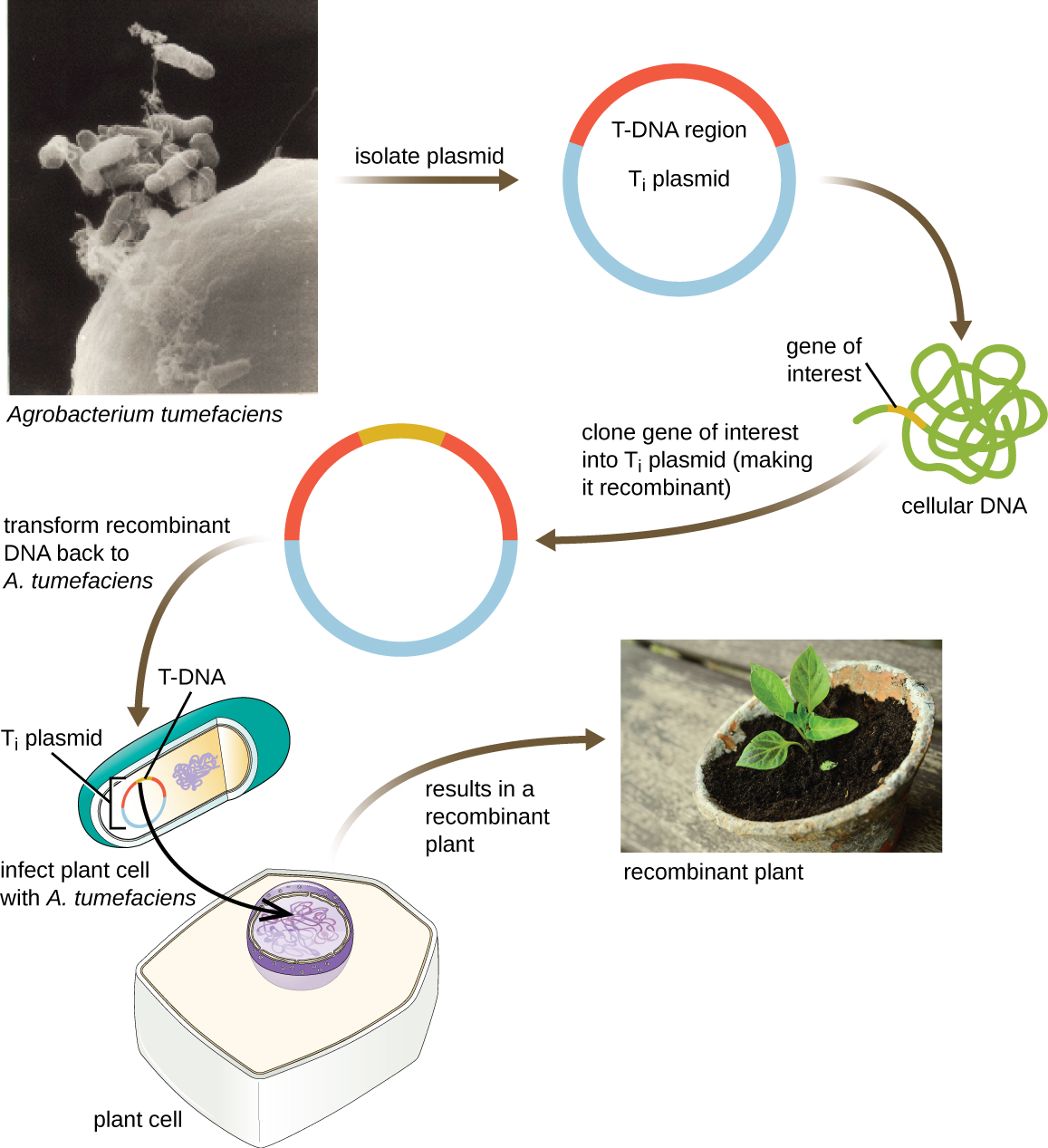
Vectores Shuttle
Otro método de transfectar plantas involucra vectores lanzadera, plásmidos que pueden moverse entre células bacterianas y eucariotas. Los plásmidos inductores de tumores (T i) originados a partir de la bacteria Agrobacterium tumefaciens se utilizan comúnmente como vectores lanzadera para incorporar genes en plantas (Figura\(\PageIndex{11}\)). En la naturaleza, los plásmidos T i de A. tumefaciens provocan que las plantas desarrollen tumores cuando se transfieren de células bacterianas a células vegetales. Los investigadores han sido capaces de manipular estos plásmidos de origen natural para eliminar sus genes causantes de tumores e insertar fragmentos de ADN deseables. Los plásmidos T i recombinantes resultantes pueden transferirse al genoma de la planta a través de la transferencia natural de plásmidos T i de la bacteria al huésped vegetal. Una vez dentro de la célula hospedadora vegetal, el gen de interés se recombina en el genoma de la célula vegetal.
Vectores Virales
También se pueden usar vectores virales para transfectar células eucariotas. De hecho, este método se utiliza a menudo en terapia génica (ver Terapia Génica) para introducir genes sanos en pacientes humanos que padecen enfermedades que resultan de mutaciones genéticas. Los genes virales se pueden eliminar y reemplazar con el gen que se va a administrar al paciente; 3 el virus infecta entonces la célula huésped y entrega el ADN extraño al genoma de la célula diana. Los adenovirus se utilizan a menudo para este propósito porque pueden cultivarse a un título alto y pueden infectar tanto a las células hospedadoras que no se dividen como en división. Sin embargo, el uso de vectores virales para la terapia génica puede plantear algunos riesgos para los pacientes, como se discute en Terapia Génica.
Ejercicio\(\PageIndex{4}\)
- ¿Cuáles son los métodos utilizados para introducir vectores de ADN recombinante en células animales?
- Comparar y contrastar vectores lanzadera y vectores virales.
Conceptos clave y resumen
- La biotecología es la ciencia de utilizar sistemas vivos para beneficiar a la humanidad. En los últimos años, la capacidad de alterar directamente el genoma de un organismo a través de la ingeniería genética ha sido posible gracias a los avances en la tecnología de ADN recombinante, lo que permite a los investigadores crear moléculas de ADN recombinante con nuevas combinaciones de material genético.
- La clonación molecular implica métodos utilizados para construir ADN recombinante y facilitar su replicación en organismos hospedadores. Estos métodos incluyen el uso de enzimas de restricción (para cortar tanto ADN extraño como vectores plasmídicos), ligadura (para pegar fragmentos de ADN juntos) e introducción de ADN recombinante en un organismo huésped (a menudo bacterias).
- El cribado azul-blanco permite la selección de transformantes bacterianos que contienen plásmidos recombinantes utilizando el fenotipo de un gen informador que se deshabilita por la inserción del fragmento de ADN.
- Las bibliotecas genómicas se pueden hacer clonando fragmentos genómicos de un organismo en vectores plasmídicos o en bacteriófagos.
- Se pueden generar bibliotecas de ADNc para representar las moléculas de ARNm expresadas en una célula en un punto dado.
- La transfección de hospedadores eucariotas se puede lograr a través de diversos métodos usando electroporación, pistolas génicas, microinyección, vectores lanzadera y vectores virales.
Notas al pie
- 1 Liangxue Lai, Jing X. Kang, Rongfeng Li, Jingdong Wang, William T. Witt, Hwan Yul Yong, Yanhong Hao et al. “Generación de Cerdos Transgénicos Clonados Rico en Ácidos Grasos Omega-3”. Nature Biotechnology 24 núm. 4 (2006): 435—436.
- 2 Raylene Ramos Moura, Luciana Magalhães Melo, y Vicente José de Figueirêdo Freitas. “Producción de Proteinas Recombinantes en Leche de Cabras Transgénicas y No Transgénicas” Archivos Brasileños de Biología y Tecnología 54 núm. 5 (2011): 927—938.
- 3 William S.M. Wold y Karoly Toth. “Vectores de Adenovirus para Terapia Génica, Vacunación y Terapia Génica Terapia Génica Actual 13 núm. 6 (2013): 421.


