2.9: Respuestas a los ejercicios
- Page ID
- 149962
Responde a la pregunta de cómo encontrar el comando R si solo sabes lo que debe hacer (por ejemplo, “anova”). Para encontrar esto desde dentro de R, puede ir de varias maneras. Primero es usar doble signo de interrogación comando?? :
Código\(\PageIndex{1}\) (R):
(La salida puede ser larga porque incluye todos los paquetes instalados. Preste atención a las filas que comenzaron con “base” y “estadísticas”.)
Se podría lograr un resultado similar si inicia la ayuda interactiva (basada en navegador web) con help.start () y luego ingresa “anova” en el cuadro de búsqueda.
En segundo lugar, una manera aún más sencilla, es usar apropos ():
Código\(\PageIndex{2}\) (R):
A veces, nada ayuda:
Código\(\PageIndex{3}\) (R):
Entonces empieza a buscar en Internet. Podría hacerse desde dentro de R:
Código\(\PageIndex{4}\) (R):
En el navegador web, debería ver la nueva pestaña (o ventana) con los resultados de la consulta.
Si nada ayuda, como la comunidad R. Comando help.request () le guiará a través de la secuencia de publicación.
Respuesta a la pregunta de la trama (Figura 2.8.4):
Código\(\PageIndex{5}\) (R):
(Aquí se usaron xlab e ylab vacíos para eliminar etiquetas de ejes. Tenga en cuenta que pch=0 es el rectángulo.)
En lugar de col="green”, se puede usar col=3. Consulte a continuación el comando palette () para entender cómo funciona. Para conocer todos los nombres de color, escriba colores (). El argumento col también podría tener múltiples valores. Comprueba qué pasa si supones, diciendo, col= 1:3 (presta atención a los últimos puntos).
Para conocer los tipos de puntos disponibles, ejecute ejemplo (puntos) y omita varias gráficas para ver la tabla de puntos; o simplemente mire la Figura A.1.1 de este libro (y lea los comentarios de cómo se hizo).
Respuesta a la pregunta sobre los huevos. Primero, carguemos el archivo de datos. Para usar el comando read.table (), necesitamos conocer la estructura del archivo. Para conocer la estructura, (1) necesitamos buscar en este archivo desde R con url.show () (o sin R, en el navegador de Internet), y también (2) buscar en el archivo complementario, eggs_c.txt.
De (1) y (2), concluimos que el archivo tiene tres columnas sin nombre de las cuales necesitaremos primera y segunda (longitud de huevo y ancho en mm, respectivamente). Las columnas están separadas con un gran espacio, muy probablemente el símbolo Tab. Ahora podemos ejecutar read.table ():
Código\(\PageIndex{6}\) (R):
El siguiente paso es siempre verificar la estructura del nuevo objeto:
Código\(\PageIndex{7}\) (R):
También es buena idea mirar en las primeras filas de datos:
Código\(\PageIndex{8}\) (R):
Nuestras variables primera y segunda recibieron los nombres V1 (largo) y V2 (ancho). Ahora necesitamos trazar variables para ver posible relación. La mejor gráfica en ese caso es una gráfica de dispersión, y para hacer scatterplot en R, simplemente usamos el comando plot ():
Código\(\PageIndex{9}\) (R):
(Gráfica de comandos (y ~ x) utiliza la interfaz de fórmula R. Es casi lo mismo que plot (x, y)\(^{[1]}\); pero tenga en cuenta el orden diferente en los argumentos.)
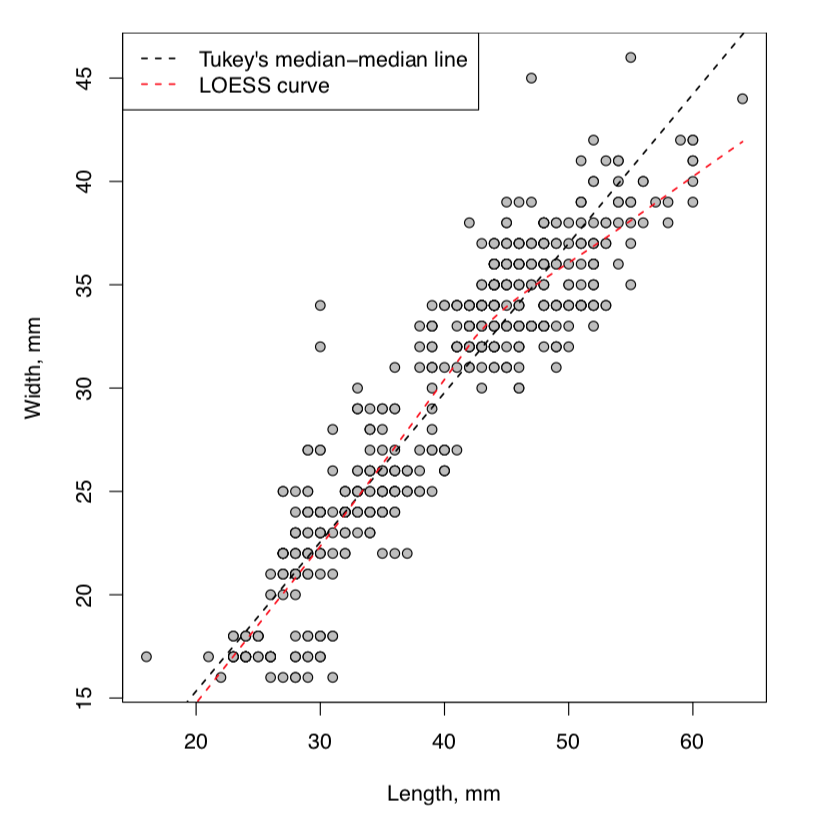
La “nube” resultante es definitivamente alargada e inclinada como debería ser en caso de dependencia. Lo que dejaría esto más claro, es algún tipo de línea “promedio” que muestra la dirección de la relación. Como es habitual, hay varias posibilidades en R (Figura\(\PageIndex{1}\)):
Código\(\PageIndex{10}\) (R):
(Tenga en cuenta el uso de line (), lines () y abline () —los tres son comandos realmente diferentes. lines () y abline () son comandos gráficos de bajo nivel que agregan línea (s) a la gráfica existente. Primero usa coordenadas mientras que el segundo usa coeficientes. line () y loess.smooth () no dibujan, calculan números para usar con comandos de dibujo. Para ver esto con más detalles, ejecute help () para cada comando.)
El enfoque de primera línea () utiliza el algoritmo de John Tukey basado en medianas (ver más abajo) mientras que loess.smooth () usa LOESS no lineal más complicado (LOCally Weighed Scatterplot Smoothing) que estima la forma general de la curva\(^{[2]}\). Ambos son aproximados pero robustos, exactamente lo que necesitamos para responder a la pregunta. Sí, existe una dependencia entre el ancho máximo del huevo y la longitud máxima del huevo.
Sin embargo, hay un problema. Mire en la Figura\(\PageIndex{1}\): muchos “huevos” se superponen con otros puntos que tienen exactamente la misma ubicación, y no es fácil ver cuántos datos pertenecen a un punto. Intentaremos acceder a esto en el próximo capítulo.
Respuesta a la pregunta del guión R. Basta con crear (con cualquier editor de texto) el archivo de texto y nombrarlo, por ejemplo, my_script1.r. En el interior, escriba lo siguiente:
pdf (” my_plot1.pdf “)
plot (1:20)
dev.off ()
Crea la prueba de subdirectorio y copia ahí tu script. Luego cierra R como de costumbre, ábrelo de nuevo, dirigirlo (a través del menú o con el comando setwd ()) para que el subdirectorio test sea el directorio de trabajo, y ejecuta:
Código\(\PageIndex{11}\) (R):
Si todo es correcto, entonces el archivo my_plot1.pdf aparecerá en el directorio test. Por favor, no olvide verificarlo: ábralo con su visor de PDF. Si algo salió mal, se recomienda eliminar la prueba de directorio junto con todo el contenido, modificar la copia maestra del script y repetir el ciclo nuevamente, hasta que los resultados sean satisfactorios.
Referencias
1. En el caso de nuestro marco de datos eggs, el comando del segundo estilo sería plot (eggs [, 1:2]) o plot (EGGs$v1, EGGs$v2), veremoreexplicationsinthenextchapter.
2. Otra variante es utilizar la función scatter.smooth () de alto nivel que reemplaza plot (). La tercera alternativa es un liso.spline () cúbico más suave que calcula los números para usar con líneas ().

