5.4: ¿Existe una asociación? Análisis de tablas
- Page ID
- 150020
Tablas de contingencia
¿Cómo se comparan muestras de datos categóricos? Estos frecuentemente son solo texto, no hay números, como en el clásico ejemplo de “Bebedor de té de Fisher”\(^{[1]}\). Una mujer británica afirmó poder distinguir si primero se le agregaba leche o té a la taza. Para probar, le dieron 8 tazas de té, en cuatro de las cuales se agregó primero la leche:
Código\(\PageIndex{1}\) (R):
La única manera es convertirlo a números, y la mejor manera de convertir es contar casos, hacer tabla de contingencia:
Código\(\PageIndex{2}\) (R):
La tabla de contingencia no es una matriz o marco de datos, es el tipo especial de objeto R llamado “tabla”.
En lenguaje de fórmulas R, las tablas de contingencia se describen con fórmula simple
~ factor (es)
Para usar este enfoque de fórmula, ejecute el comando xtabs ():
Código\(\PageIndex{3}\) (R):
(Hay que conectar más de un factor con el signo +.)
Si hay más de dos factores, R puede construir una tabla multidimensional e imprimirla como una serie de tablas bidimensionales. Por favor llame a los datos incrustados del Titanic para ver cómo se ve la tabla de contingencia tridimensional. Se puede construir una tabla de contingencia “plana” si todos los factores excepto uno se combinan en un factor multidimensional. Para ello, usa el comando ftable ():
Código\(\PageIndex{4}\) (R):
La tabla de funciones se puede utilizar simplemente para el cálculo de frecuencias (incluyendo datos faltantes, si es necesario):
Código\(\PageIndex{5}\) (R):
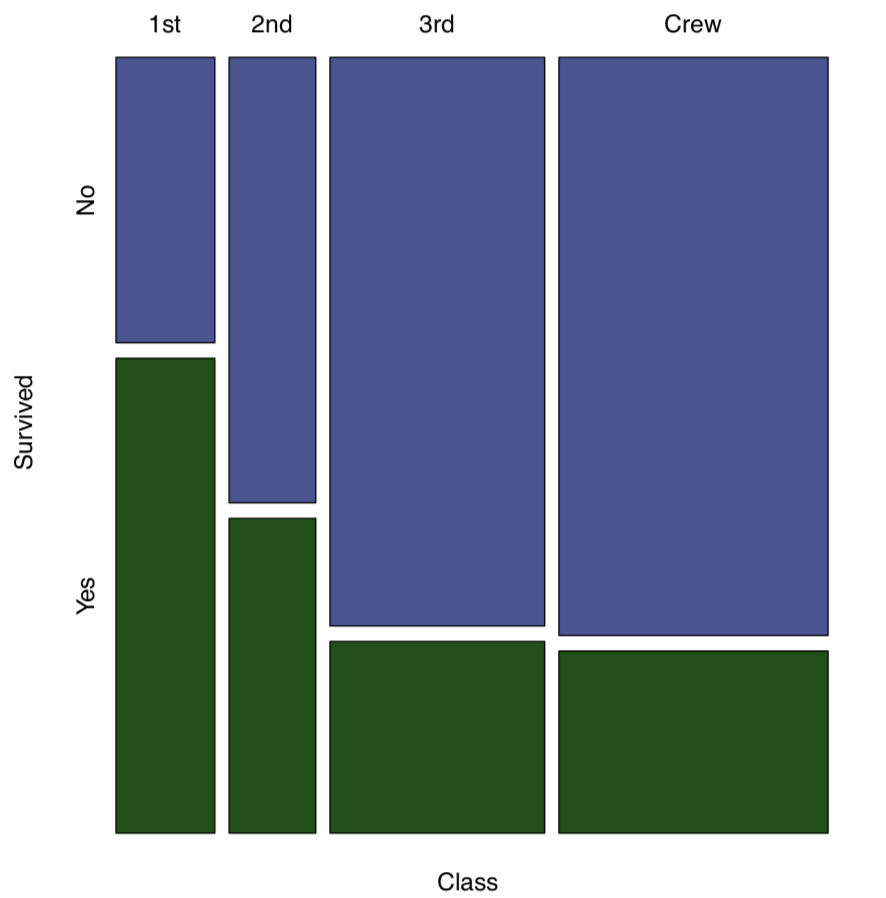
La función mosaicplot () crea una representación gráfica de una tabla de contingencia (Figura\(\PageIndex{1}\)):
Código\(\PageIndex{6}\) (R):
(Se utilizó el comando mosaicplot () porque apply () dio salida a una matriz. Si los datos son una “tabla” con más de una dimensión, el comando object, plot () generará la gráfica de mosaico por defecto.)
Las tablas de contingencia son bastante fáciles de hacer incluso a partir de datos numéricos. Supongamos que necesitamos buscar asociación entre mes y temperaturas cómodas en Nueva York. Si las temperaturas de 64 a 86°F (de 18 a 30°C) son temperaturas de confort, entonces:
Código\(\PageIndex{7}\) (R):
Ahora tenemos dos variables categóricas, confort y Airquality$month y podemos proceder a la tabla:
Código\(\PageIndex{8}\) (R):
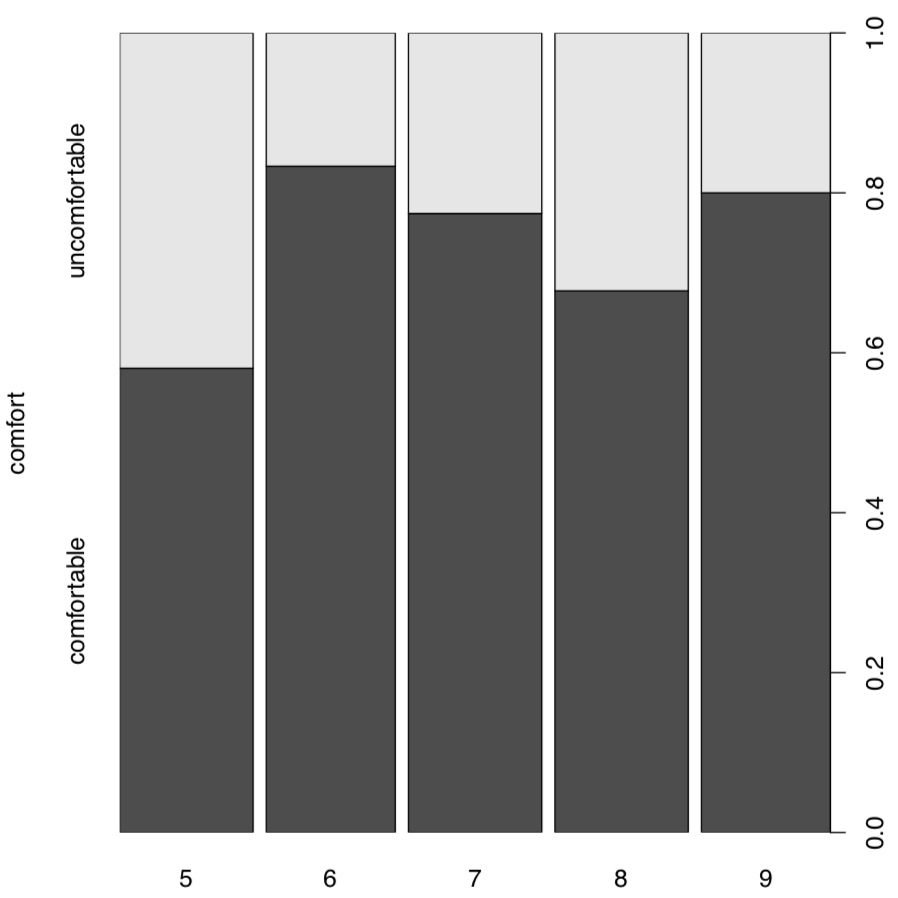
La gráfica de columna vertebral (Figura\(\PageIndex{2}\)) es buena para este tipo de tabla, parece un “híbrido” visualmente avanzado entre histograma, gráfico de barras y diagrama de mosaico:
Código\(\PageIndex{9}\) (R):
(Otra variante para trazar estas tablas bidimensionales es el dotchart (), por favor pruébalo tú mismo. Dotchart también es bueno para tablas unidimensionales, pero a veces es posible que necesite usar el reemplazo Dotchart1 () de asmisc.r —mantiene espacio para la etiqueta del eje y).
Pruebas de mesa
Para encontrar si hay una asociación en una tabla, se deben comparar dos frecuencias en cada celda: predicha (teórica) y observada. La seria diferencia es el signo de asociación. Los pares de hipótesis nulas y alternativas suelen ser:
- Nulo: distribución independiente de factores\(\approx\) sin patrón presente\(\approx\) ninguna asociación presente
- Alternativa: distribución concertada de factores\(\approx\) patrón presente\(\approx\) hay asociación
En términos de valores p:
La función chisq.test () ejecuta una prueba de chi-cuadrado, una de las dos pruebas más utilizadas para tablas de contingencia. La prueba de chi-cuadrado (o\(\chi^2\)) de dos muestras requiere ya sea tabla de contingencia o dos factores de la misma longitud (para calcular tabla a partir de ellos primero).
Ahora bien, ¿qué pasa con la tabla de confort de temperatura? assocplot (comf.month) muestra algunas desviaciones “sospechosas”. Para verificar si estos son estadísticamente significativos:
Código\(\PageIndex{10}\) (R):
No, no están asociados. Como antes, no hay nada misterioso en estos números. Todo se basa en diferencias entre valores esperados y observados:
Código\(\PageIndex{11}\) (R):
(Observe cómo se calculan los valores esperados y cómo se ven: esperados (nulos) son proporciones iguales entre ambas filas y columnas. Junio y septiembre tienen 30 días cada uno, de ahí ligeras diferencias en los valores, pero no en las proporciones esperadas).
Veamos ahora si el color del cabello y el color de ojos de los datos de HaireyeColor incrustados en tres dimensiones están asociados. Primero, podemos examinar las asociaciones gráficamente con assocplot () (Figura\(\PageIndex{3}\)):
Código\(\PageIndex{12}\) (R):
(En lugar de apply () utilizado en el ejemplo anterior, empleamos margin.table () que esencialmente hizo el mismo trabajo).
La gráfica de asociación muestra varias cosas: la altura de las barras refleja la contribución de cada celda al chi-cuadrado total, esto permite, por ejemplo, detectar valores atípicos. El cuadrado del rectángulo corresponde con la diferencia entre el valor observado y el esperado, así los rectángulos altos grandes indican más asociación (para entender esto mejor, compare esta parcela actual con assocplot (comf.month)). El color y la posición del rectángulo muestran el signo de la diferencia.
En general, es probable que haya una asociación. Ahora necesitamos verificar esta hipótesis con una prueba:
Código\(\PageIndex{13}\) (R):
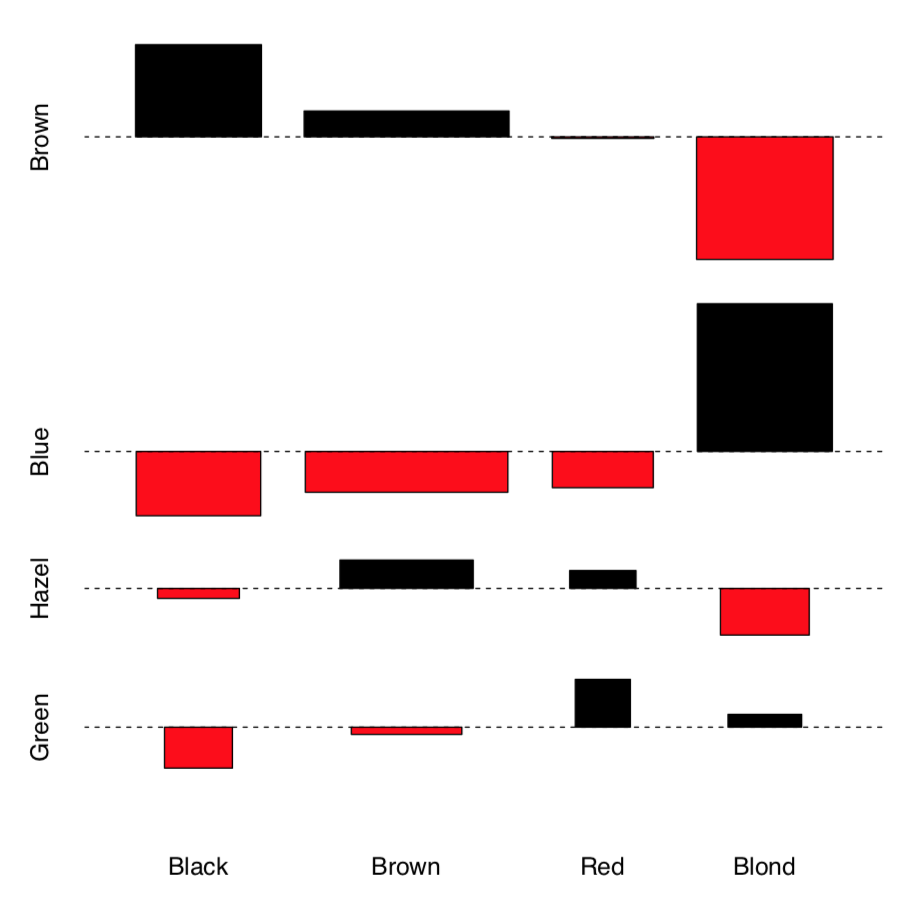
La prueba de chi-cuadrado toma como hipótesis nula “sin patrón”, “sin asociación”. Por lo tanto, en nuestro ejemplo, como rechazamos la hipótesis nula, encontramos que los factores están asociados.
¿Y qué pasa con la supervivencia en el “Titanic”?
Código\(\PageIndex{14}\) (R):
Sí (como recordará el lector de la famosa película), la supervivencia se asoció con estar en la clase particular.
La prueba general de chi-cuadrado muestra solo si la asimetría se presenta en cualquier parte de la tabla. Esto quiere decir que si es significativo, entonces al menos un grupo de pasajeros tiene la diferencia en la supervivencia. Al igual que ANOVA, la prueba no muestra cuál. Post hoc, o prueba de tabla por pares es capaz de mostrar esto:
Código\(\PageIndex{15}\) (R):
De la tabla de valores p, es evidente que los miembros de la tercera clase y de la tripulación no fueron diferentes por las tasas de supervivencia. Tenga en cuenta que las pruebas post hoc aplican el ajuste del valor p para comparaciones múltiples; prácticamente, significa que debido a que se realizaron 7 pruebas simultáneamente, los valores p se ampliaron con algún método (aquí, el método Benjamini & Hochberg es el predeterminado).
El archivo seedlings.txt contiene los resultados de un experimento que examina la germinación de semillas infectadas con diferentes tipos de hongos. En total, se probaron tres hongos, se probaron 20 semillas para cada hongo, y por lo tanto con los controles se probaron 80 semillas. ¿Difieren las tasas de germinación de las semillas infectadas?
Examinemos ahora el ejemplo más complicado. Un nutrido grupo de epidemiólogos se reunieron para una fiesta. A la mañana siguiente, muchos se despertaron con síntomas de intoxicación alimentaria. Debido a que eran epidemiólogos, decidieron recordar lo que cada uno de ellos comió en el banquete, y así determinar cuál fue la causa de la enfermedad. Los datos recopilados toman el siguiente formato:
Código\(\PageIndex{16}\) (R):
(Usamos head () aquí porque la mesa es muy larga.)
La primera variable (ILL) indica si el participante se enfermó o no (1 o 2 respectivamente); las variables restantes corresponden a diferentes alimentos.
Una simple mirada a los datos no revelará nada, ya que el banquete contó con 45 participantes y 13 comidas diferentes. Por lo tanto, se deben utilizar métodos estadísticos. Dado que los datos son nominales, utilizaremos tablas de contingencia:
Código\(\PageIndex{17}\) (R):
(Primero, ejecutamos la variable ILL contra cada columna e hicimos una lista de pequeñas tablas de contingencia. En segundo lugar, convertimos la lista en matriz tridimensional, al igual que lo son los datos del Titanic, y también hicimos nombres sensibles de dimensiones).
Ahora nuestros datos consisten en pequeñas tablas de contingencia que son elementos de matriz:
Código\(\PageIndex{18}\) (R):
(Tenga en cuenta dos comas que necesitaban decirle a R que queremos la tercera dimensión de la matriz.)
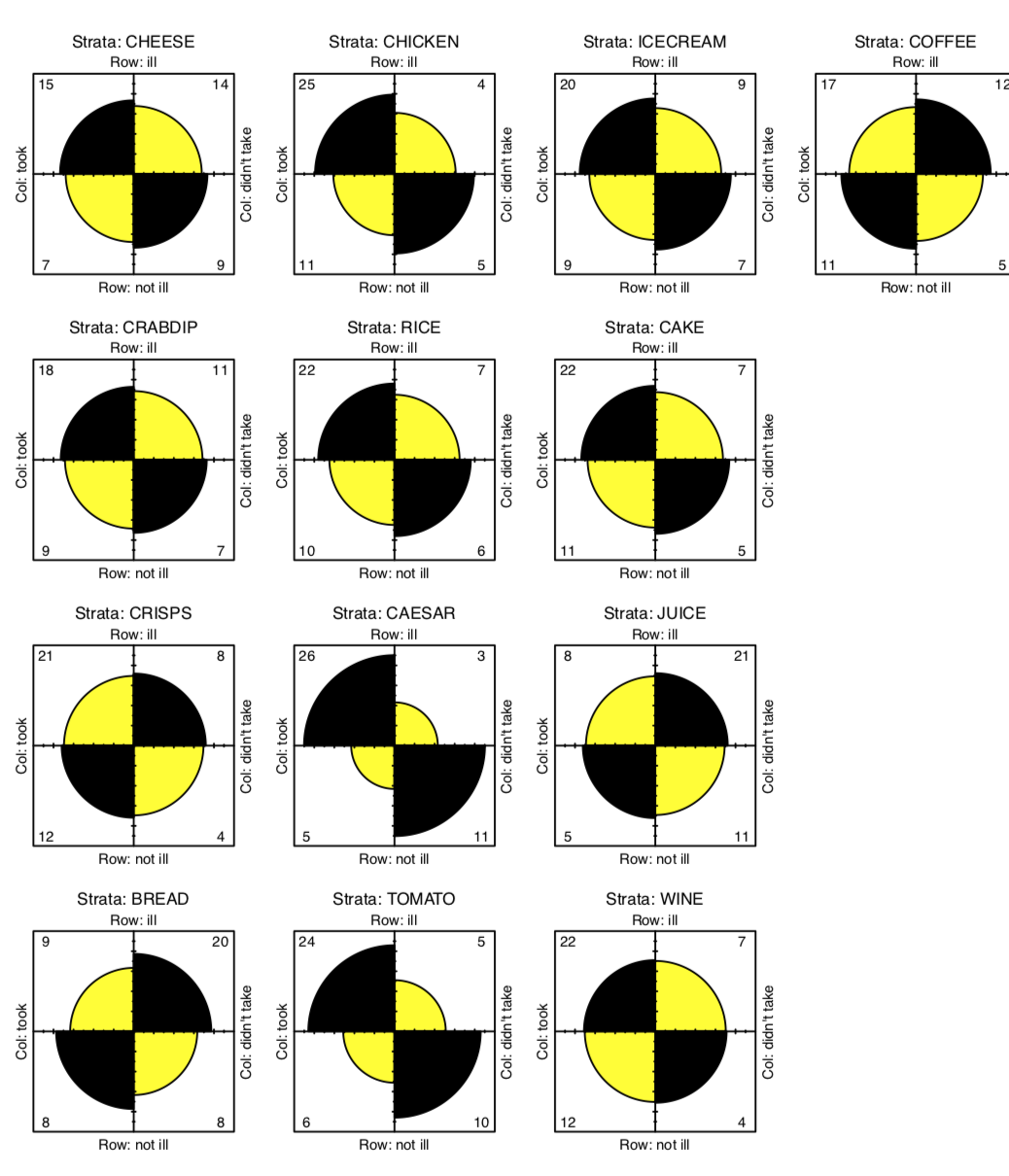
Ahora necesitamos una especie de análisis de tabla estratificado (con cada tipo de alimento). Dado que cada elemento en la tox.2 es\(2\times2\) tabla, la gráfica cuádruple visualizará bien estos datos (Figura\(\PageIndex{4}\)):
Código\(\PageIndex{19}\) (R):
(En parcelas cuádruple, la asociación corresponde con la diferencia entre dos pares de sectores diagonales. Dado que probamos varias veces, los anillos de confianza se suprimen).
Existen algunas diferencias aparentes, especialmente para CESAR, PAN y TOMATE. Para verificar su significado, primero aplicaremos la prueba de chi-cuadrado varias veces y verificaremos los valores p:
Código\(\PageIndex{20}\) (R):
(Un apply () nos permite no escribir el código para la prueba 13 veces. Puede omitir cbind () ya que solo se usaba para hacer que la salida fuera más bonita. Hubo múltiples advertencias, y pronto volveremos a ellas.)
El resultado es que dos alimentos presentan asociaciones significativas con la enfermedad: la ensalada César y el tomate. ¡Se identifica al culpable! Casi. Después de todo, es poco probable que ambos platillos estuvieran contaminados. Ahora debemos tratar de determinar cuál fue la principal causa de la intoxicación alimentaria. Volveremos a este tema más adelante.
Hablemos de un detalle más. Arriba, aplicamos la prueba de chi-cuadrado simultáneamente varias veces. Para dar cuenta de comparaciones múltiples, debemos ajustar los valores p, ampliarlos de acuerdo con la regla particular, por ejemplo, con la regla de corrección Bonferroni ampliamente conocida, o con la regla de corrección (más confiable) de Benjamini y Hochberg como en el siguiente ejemplo:
Código\(\PageIndex{21}\) (R):
Ahora ya sabes cómo aplicar correcciones de valor p para comparaciones múltiples. Trate de hacer esto para nuestros datos de toxicidad. A lo mejor, ¿ayudará a identificar al culpable?
El caso especial de la prueba de chi-cuadrado es la prueba de bondad de ajuste, o prueba G. Lo aplicaremos a los famosos datos, resultados del primer experimento de Gregor Mendel. En este experimento, cruzó plantas de guisante que crecieron a partir de semillas redondas y anguladas. Al contar semillas de la primera generación de híbridos, encontró que entre 7 mil 324 semillas, 5 mil 474 fueron redondas y 1850 en ángulo. Mendel supuso que la verdadera proporción en este y otros seis experimentos es 3:1\(^{[2]}\):
Código\(\PageIndex{22}\) (R):
La prueba de bondad de ajuste utiliza el nulo de que las frecuencias en el primer argumento (interpretada como tabla de contingencia unidimensional) no son diferentes de las probabilidades en el segundo argumento. Por lo tanto, se apoya estadísticamente la relación 3:1. Como podría notar, no es radicalmente diferente de la prueba de proporción explicada en el capítulo anterior.
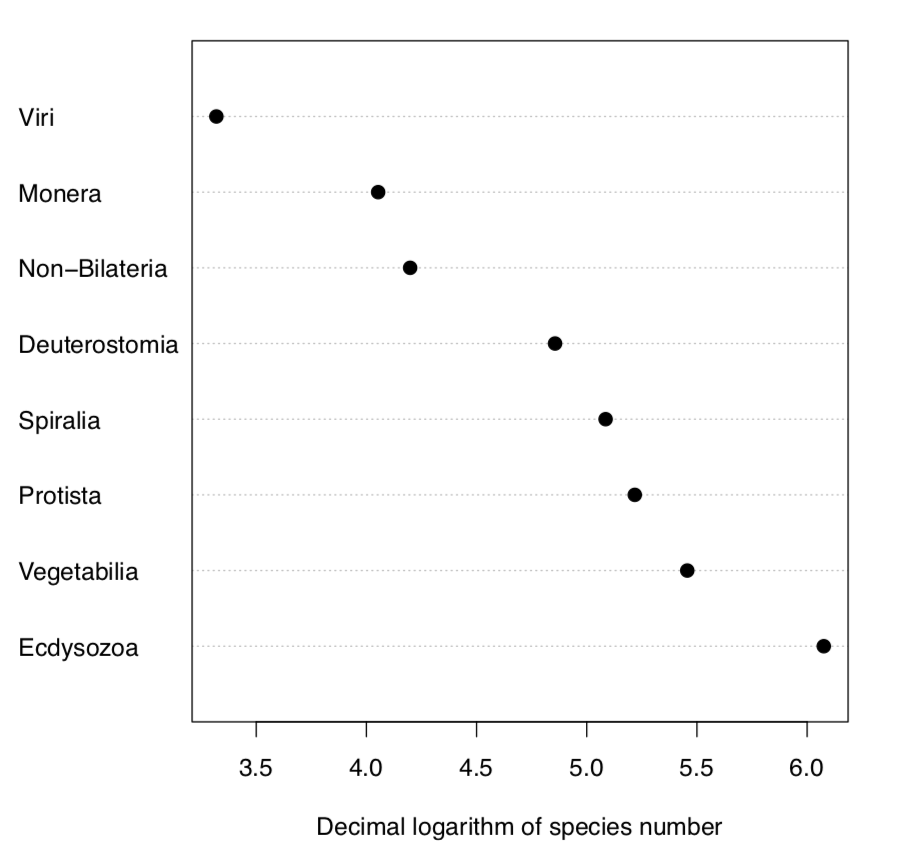
Sin el parámetro p, la prueba G simplemente verifica si las probabilidades son iguales. Comprobemos, por ejemplo, si el número de especies en supergrupos de organismos vivos en la Tierra es igual:
Código\(\PageIndex{23}\) (R):
Naturalmente, el número de especies no es igual entre supergrupos. Algunos de ellos como bacterias (supergrupo Monera) tienen sorprendentemente bajo número de especies, otros como insectos (supergrupo Ecdysozoa) —número realmente grande (Figura\(\PageIndex{5}\)).
La prueba de chi-cuadrado funciona bien cuando el número de casos por célula es más de 5. Si hay menos casos, R da al menos tres soluciones alternativas.
Primero, en lugar del valor p estimado a partir de la distribución teórica, hay una manera de calcularlo directamente, con la prueba exacta de Fisher. La mesa bebedero de té contiene menos de 5 cajas por celda por lo que es un buen ejemplo:
Código\(\PageIndex{24}\) (R):
La prueba de Fisher comprueba el nulo si la razón de probabilidades es solo una. Si bien en este caso, el cálculo da odds ratio\((3\mathbin{:}1)/(1\mathbin{:}3)=9\), solo hay 8 observaciones, y el intervalo de confianza aún incluye una. Por lo tanto, contrariamente a la primera impresión, la prueba no apoya la idea de que la mujer antes mencionada sea una buena adivinadora.
La parcela cuádruple (por favor compruébelo usted mismo) da el resultado similar:
Código\(\PageIndex{25}\) (R):
Si bien existe una diferencia aparente entre las diagonales, los anillos de confianza se cruzan significativamente.
La prueba de Fisher es computacionalmente intensiva por lo que no se recomienda su uso para un gran número de casos.
La segunda solución es la corrección de continuidad de Yates que en R es predeterminada para la prueba de chi-cuadrado en 22 tablas. Ahora utilizamos datos de la\(^{[3]}\) publicación original de Yates (1934), los datos se toman del estudio de la influencia de la lactancia materna y artificial en la formación de dientes:
Código\(\PageIndex{26}\) (R):
(Tenga en cuenta la advertencia al final.)
La corrección de Yates no es un valor predeterminado para la función summary.table ():
Código\(\PageIndex{27}\) (R):
(Nota diferente valor p: este es un efecto de no corrección. Para todos los demás tipos de tablas (e.g., non\(2\times2\)), los resultados de chisq.test () y summary.table () deben ser similares.)
La tercera forma es simular el valor p de prueba chi-cuadrado con replicación:
Código\(\PageIndex{28}\) (R):
(Tenga en cuenta que dado que este algoritmo se basa en un procedimiento aleatorio, los valores p pueden diferir).
¿Cómo calcular un tamaño de efecto para la asociación de variables categóricas? Uno de ellos es el ratio de probabilidades de la prueba de Fisher (ver arriba). También hay varias medidas de tamaño de efecto diferentes que cambian de 0 (sin asociación) a (teóricamente) 1 (que es una asociación extremadamente fuerte). Si no desea utilizar paquetes externos, uno de ellos,\(\phi\) coeficiente es fácil de calcular a partir de la estadística\(\chi\) -cuadrado.
Código\(\PageIndex{29}\) (R):
\(\Phi\)coeficiente funciona sólo para dos variables binarias. Si las variables no son binarias, hay coeficientes T de Tschuprow y V de Cramer. Ahora es mejor usar el código externo del asmisc.r distribuyendo con este libro:
Código\(\PageIndex{30}\) (R):
R paquete vcd tiene función assocstats () que calcula odds ratio,\(\phi\), V de Cramer y varias otras medidas de efecto.
En el repositorio abierto, el archivo cochlearia.txt contiene mediciones de caracteres morfológicos en varias poblaciones (localizaciones) de escorbuto-pasto, Cochlearia. Uno de los caracteres, binario IS.CREEPING refleja la forma de vida vegetal: rastrero o tallo erguido. Verifique si el número de plantas rastreras es diferente entre ubicaciones, proporcione tamaños de efecto y valores p.
Hay muchas pruebas de mesa. Por ejemplo, la prueba de proporciones del capítulo anterior podría extenderse fácilmente para dos muestras y por lo tanto podría usarse como prueba de tabla. También existe mcnemar.test () que se utiliza para comparar proporciones cuando pertenecen a los mismos objetos (proporciones emparejadas). Es posible que desee consultar la ayuda (y especialmente los ejemplos) para entender cómo funcionan.
En los datos de betula (ver arriba), hay dos caracteres binarios: LÓBULOS (posición de los lóbulos en la bráctea de la flor) y ALAS (el tamaño relativo de las alas del fruto). Por favor, encuentre si las proporciones de plantas con valores 0 y 1 de LOBOS son diferentes entre la ubicación 1 y la ubicación 2.
¿Las proporciones de LOBES y valores WING son diferentes en todo el conjunto de datos?
La secuencia típica de procedimientos relacionados con el análisis de tablas se enumera a continuación:
- Comprobar el fenómeno de asociación: table (), xtabs ()
- Trazar primero: mosaicplot (), spineplot (), assocplot ()
- Decidir es la asociación es estadísticamente significativa: chisq.test (), fisher.test ()
- Mida qué tan fuerte es una asociación: VTCoeffs ()
- Opcionalmente, si hay más de dos grupos por caso involucrado, ejecute pruebas de pairise post hoc con la corrección apropiada: Pares.Table2.test ()
Para concluir este capítulo de “diferencias”, aquí está la Tabla\(\PageIndex{1}\) que guiará al lector a través de los tipos de análisis más utilizados. Tenga en cuenta también el cuadro mucho más detallado 6.1.1 del apéndice.
| Normal | No normal | ||
|---|---|---|---|
| medición o clasificado | nominal | ||
| \(\mathbf{=2}\)muestras | Prueba de estudiante | Prueba de Wilcoxon | Prueba Chi-cuadrado (+ prueba post-hoc) |
| \(\mathbf{>2}\)muestras |
ANOVA o unidireccional + alguna prueba post hoc |
Kruskall-Wallis + alguna prueba post hoc |
|
Cuadro\(\PageIndex{1}\): Métodos utilizados con mayor frecuencia para analizar diferencias y patrones. Esta es la variante simplificada del Cuadro 6.1.1.
Referencias
1. Fisher R.A. 1971. El diseño de experimentos. 9ª ed. P. 11.
2. Mendel G. 1866. Versuche über Pflanzen-Hybriden. Verhandlungen des naturforschenden Vereines en Brünn. Bd. 4, Abhandlungen: 12. http://biodiversitylibrary.org/page/40164750
3. Yates F. 1934. Tablas de contingencia que involucran números pequeños y la\(x^2\) prueba. Revista de la Real Sociedad Estadística. 1 (2): 217—235.


