5.5: Respuestas a ejercicios
- Page ID
- 150027
Dos pruebas de muestra, tamaños de efecto
Respuesta a la pregunta de prueba de signo. Basta con escribir:
Código\(\PageIndex{1}\) (R):
Aquí la prueba de signos no logró encontrar diferencias obvias porque (como la prueba t y la prueba de Wilcoxon) considera solo valores centrales.
Respuesta a la pregunta del ozono. Para saber si nuestros datos se distribuyen normalmente, podemos aplicar la función Normality ():
Código\(\PageIndex{2}\) (R):
(Aquí aplicamos la función unstack () que segregó nuestros datos por meses.)
Respuesta a la pregunta del argón. Primero, tenemos que verificar los supuestos:
Código\(\PageIndex{3}\) (R):
Es claro que en este caso, la prueba no paramétrica funcionará mejor:
Código\(\PageIndex{4}\) (R):
(Utilizamos jitter () para romper lazos. Sin embargo, tenga cuidado e intente verificar si este ruido aleatorio no influye en el valor p. Aquí, no lo hace.)
Y sí, las gráficas de caja (Figura 5.2.4) decían la verdad: hay una diferencia estadística entre dos conjuntos de números.
Respuesta a la pregunta de los cajeros. Verifique primero la normalidad:
Código\(\PageIndex{5}\) (R):
Ahora, podemos comparar medios:
Código\(\PageIndex{6}\) (R):
Es probable que el primer cajero tenga líneas generalmente más grandes:
Código\(\PageIndex{7}\) (R):
La diferencia no es significativa.
Respuesta a la pregunta de calificaciones. Primero, comprobar la normalidad:
Código\(\PageIndex{8}\) (R):
(Function split () creó tres nuevas variables de acuerdo con el factor de agrupación; es similar a unstack () de la respuesta anterior pero puede aceptar grupos de tamaño desigual.)
Datos de verificación (también es posible trazar parcelas de caja):
Código\(\PageIndex{9}\) (R):
Es probable que la primera clase tenga resultados similares entre exámenes pero en el primer examen, el segundo grupo podría tener mejores calificaciones. Como los datos no son normales, usaremos métodos no paramétricos:
Código\(\PageIndex{10}\) (R):
Para la primera clase, aplicamos la prueba pareada ya que las calificaciones en primer y segundo exámenes pertenecen a las mismas personas. Para ver si existen diferencias entre diferentes clases, utilizamos hipótesis alternativas unilaterales porque necesitábamos entender no si la segunda clase es diferente, sino si es mejor.
En consecuencia, las calificaciones de la primera clase no son significativamente diferentes entre exámenes, pero la segunda clase se desempeñó significativamente mejor que la primera. El primer intervalo de confianza incluye cero (como debería ser en el caso de que no haya diferencia), y el segundo no es de mucha utilidad.
Ahora tamaños de efecto con Delta de Cliff no paramétrico adecuado:
Código\(\PageIndex{11}\) (R):
Por lo tanto, los resultados de la segunda clase son solo ligeramente mejores, lo que incluso podría ser insignificante ya que el intervalo de confianza incluye 0.
Respuesta a la pregunta sobre las hojas de saúco molido (Figura\(\PageIndex{1}\)).
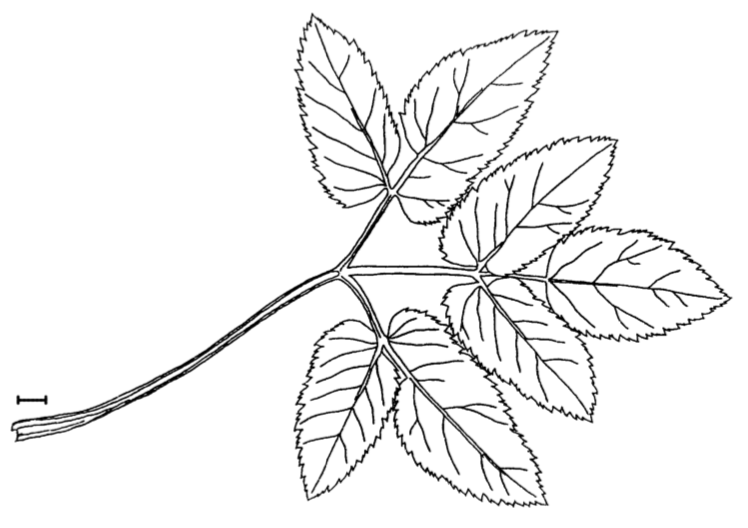
Primero, verificar los datos, cargarlos y verificar el objeto:
Código\(\PageIndex{12}\) (R):
(También convertimos la variable SUN en factor y suministramos las etiquetas adecuadas).
Comprobemos los datos para la normalidad y para el carácter más diferente (Figura\(\PageIndex{2}\)):
Código\(\PageIndex{13}\) (R):
TERM.L (longitud de la valva terminal, es la más a la derecha en la Figura\(\PageIndex{1}\)), es probablemente más diferente entre sol y sombra. Dado que este carácter es normal, realizaremos una prueba paramétrica más precisa:
Código\(\PageIndex{14}\) (R):
Para reportar el resultado de la prueba t, es necesario proporcionar grados de libertad, estadística y valor p, por ejemplo, como “en una prueba de Welch, el estadístico t es 14.85 sobre 63.69 grados de libertad, el valor p es cercano a cero, así rechazamos la hipótesis nula”.
Los tamaños de los efectos suelen estar concertados con valores p pero proporcionan información útil adicional sobre la magnitud de las diferencias:
Código\(\PageIndex{15}\) (R):
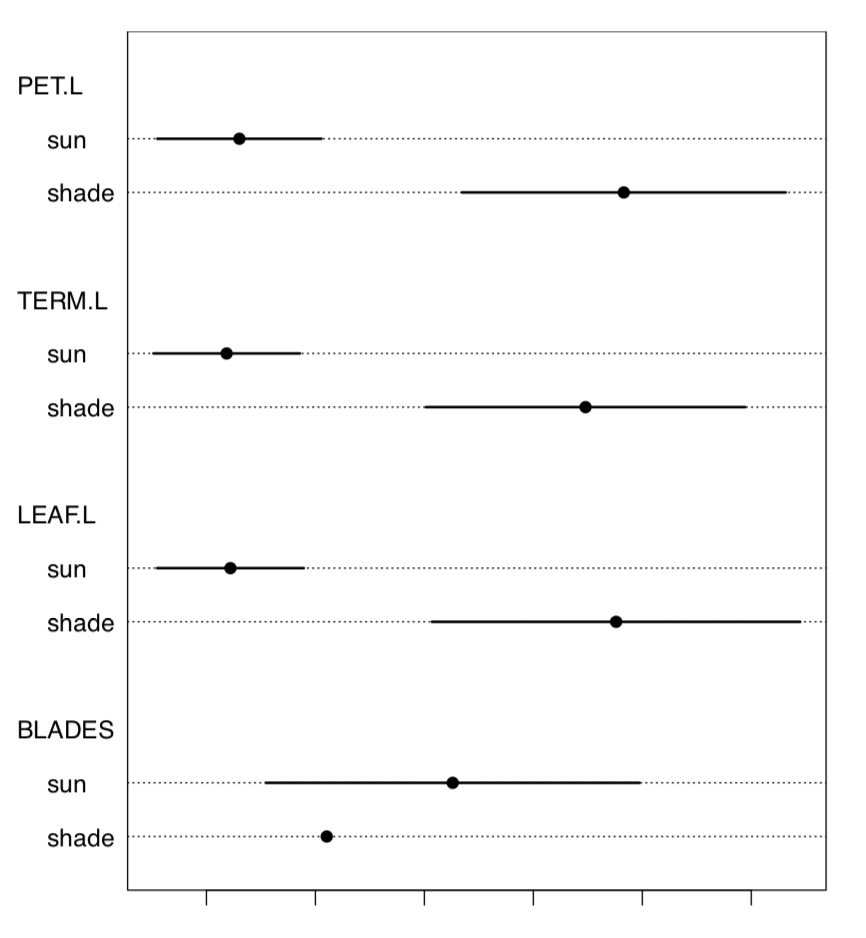
Tanto la d de Cohen como la K de Lyubishchev (coeficiente de divergencia) son grandes.
ANOVA
Responder a las preguntas de altura y color. Sí en ambas preguntas:
Código\(\PageIndex{16}\) (R):
Existen diferencias significativas entre los tres grupos.
Respuesta a la pregunta sobre las diferencias entre trigos de vacuno (Figura\(\PageIndex{3}\)) de siete ubicaciones.
Cargue los datos y compruebe su estructura:
Código\(\PageIndex{17}\) (R):
Trazar primero (Figura\(\PageIndex{4}\)):
Código\(\PageIndex{18}\) (R):
Comprobar suposiciones:
Código\(\PageIndex{19}\) (R):
En consecuencia, la longitud de la hoja debe analizarse con procedimiento no paramétrico, y la altura de la planta, con paramétrica que no asume homogeneidad de varianza (prueba unidireccional):
Código\(\PageIndex{20}\) (R):
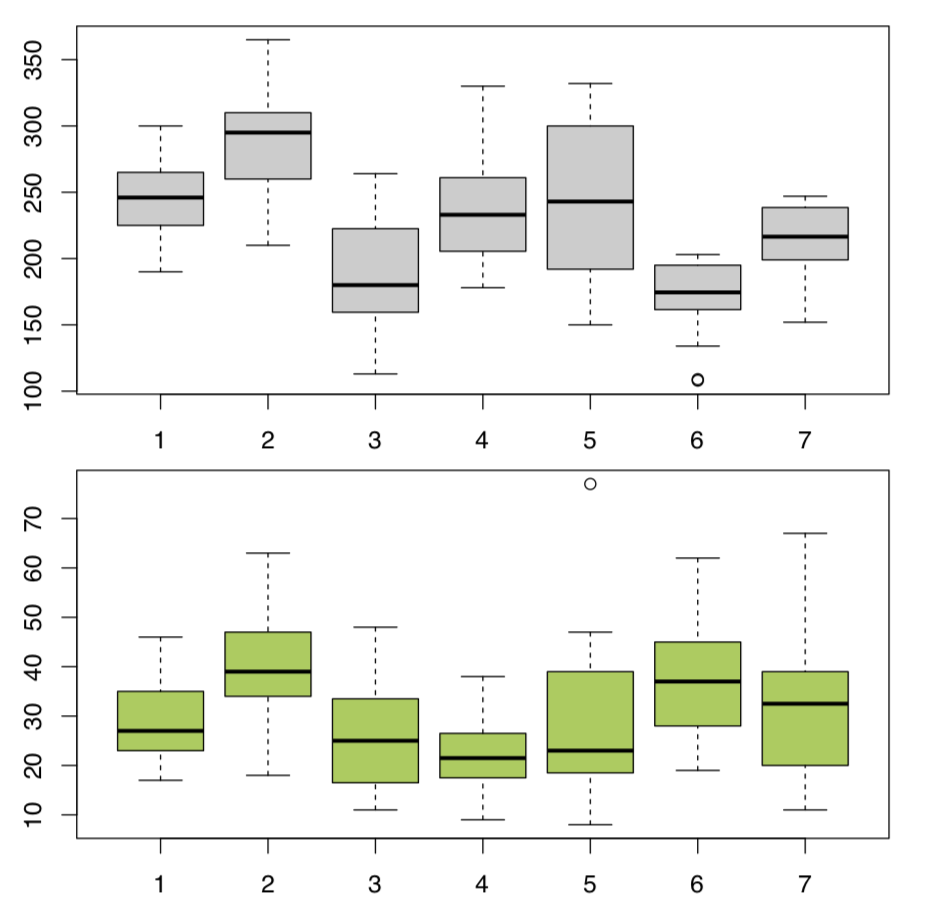
Ahora la longitud de la hoja:
Código\(\PageIndex{21}\) (R):
En general, los pares de ubicación 2—4 y 4—6 son divergentes estadísticamente en ambos casos. Esto es visible también en las gráficas de caja (Figura\(\PageIndex{5}\)). Hay diferencias más significativas en las alturas de las plantas, la ubicación #6, en particular, es bastante sobresaliente.
Tablas de contingencia
Respuesta a la pregunta de las plántulas. Cargar datos y verificar su estructura:
Código\(\PageIndex{22}\) (R):
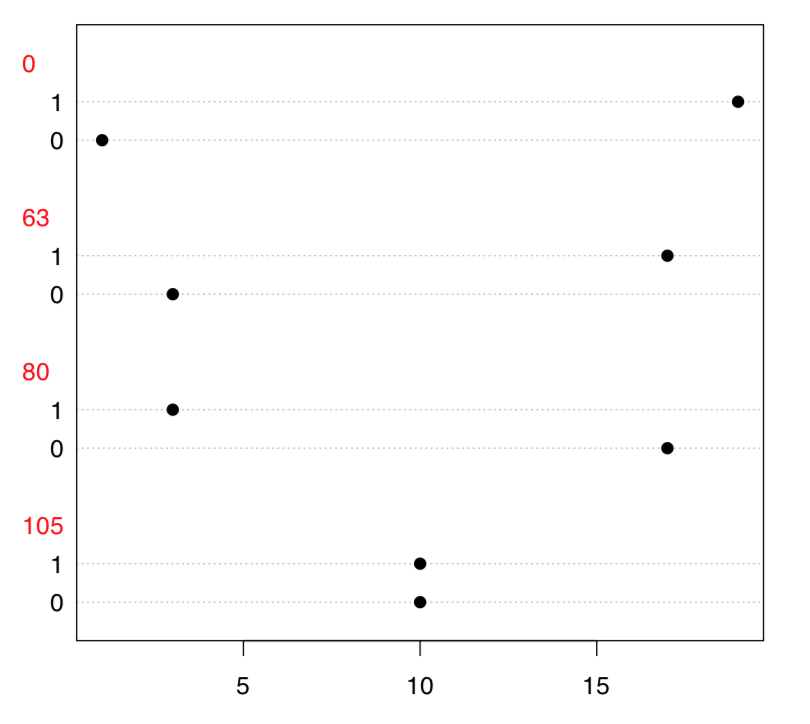
Ahora bien, lo que necesitamos es examinar la tabla porque ambas variables sólo parecen números; de hecho, son categóricas. Dotchart (Figura\(\PageIndex{5}\)) es una buena manera de explorar la tabla bidimensional:
Código\(\PageIndex{23}\) (R):
Para explorar posibles asociaciones visualmente, empleamos el paquete vcd:
Código\(\PageIndex{24}\) (R):
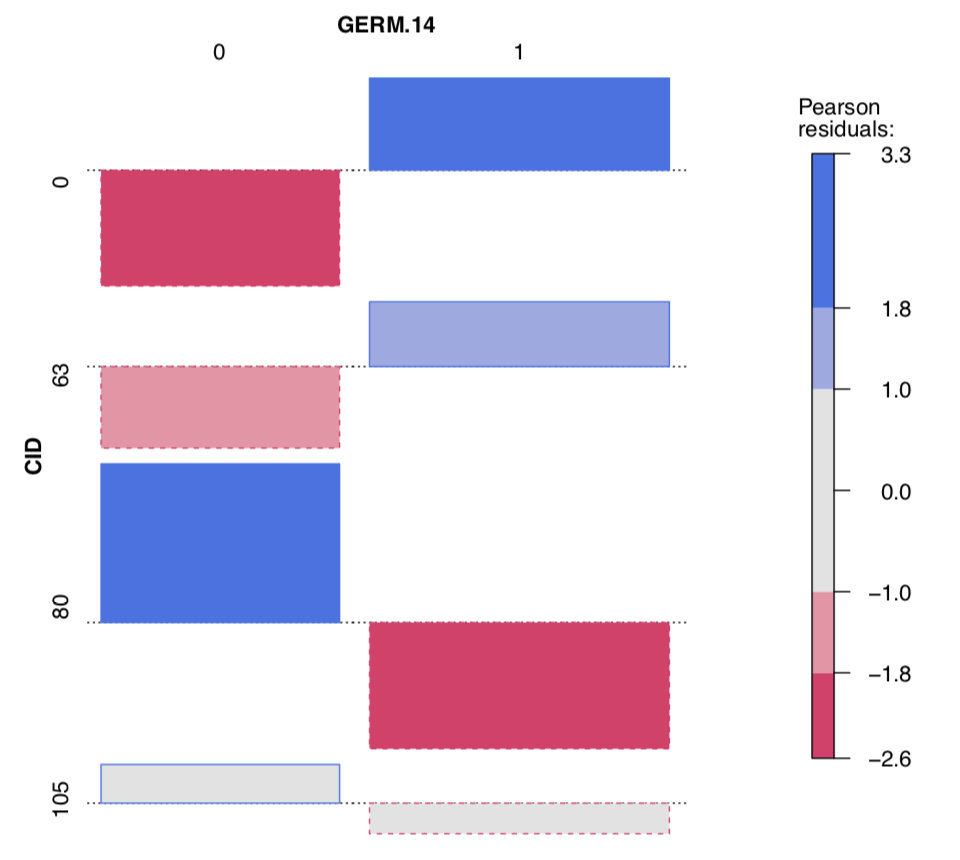
Tanto la salida de la tabla como la gráfica de asociación vcd (Figura\(\PageIndex{6}\)) sugieren cierta asimetría (especialmente para CID80) que es un signo de posible asociación. Comprobemos numéricamente, con la prueba de chi-cuadrado:
Código\(\PageIndex{25}\) (R):
Sí, existe una asociación entre el hongo (o su ausencia) y la germinación. ¿Cómo conocer las diferencias entre muestras particulares? Aquí necesitamos una prueba post hoc:
Código\(\PageIndex{26}\) (R):
(Se utilizó la prueba exacta de Fisher porque algunos recuentos fueron realmente pequeños).
Ahora está claro que los patrones de germinación de dos infecciones fúngicas, CID80 y CID105, son significativamente diferentes de la germinación en el testigo (CID0). Además, se encontró asociación significativa en cada comparación entre tres infecciones; esto significa que los tres patrones de germinación son estadísticamente diferentes. Finalmente, un hongo, CID63 produce un patrón de germinación que no es estadísticamente diferente del testigo.
Respuesta a la pregunta sobre múltiples comparaciones de toxicidad. Aquí vamos a ir por el camino un poco diferente. En lugar de usar array, extraeremos los valores p directamente de los datos originales, y evitaremos advertencias con la prueba exacta:
Código\(\PageIndex{27}\) (R):
(No podemos usar PairWise.table2.test () de la respuesta anterior ya que nuestras comparaciones tienen una estructura diferente. Pero usamos prueba exacta para evitar advertencias relacionadas con pequeños números de recuentos).
Ahora podemos ajustar los valores p:
Código\(\PageIndex{28}\) (R):
Bueno, ahora podemos decir que la ensalada César y los tomates son apoyados estadísticamente como culpables. Pero, ¿por qué las pruebas de mesa siempre nos muestran dos factores? Esto podría deberse a la interacción: en palabras simples, significa que las personas que tomaron la ensalada, frecuentemente llevaban tomates con ella.
Respuesta a la pregunta del escorbuto-pasto. Verifique el archivo de datos, cargue y verifique el resultado:
Código\(\PageIndex{29}\) (R):
(Además, convertimos LOC e IS.CREEPER a factores y proporcionamos etiquetas de nuevo nivel).
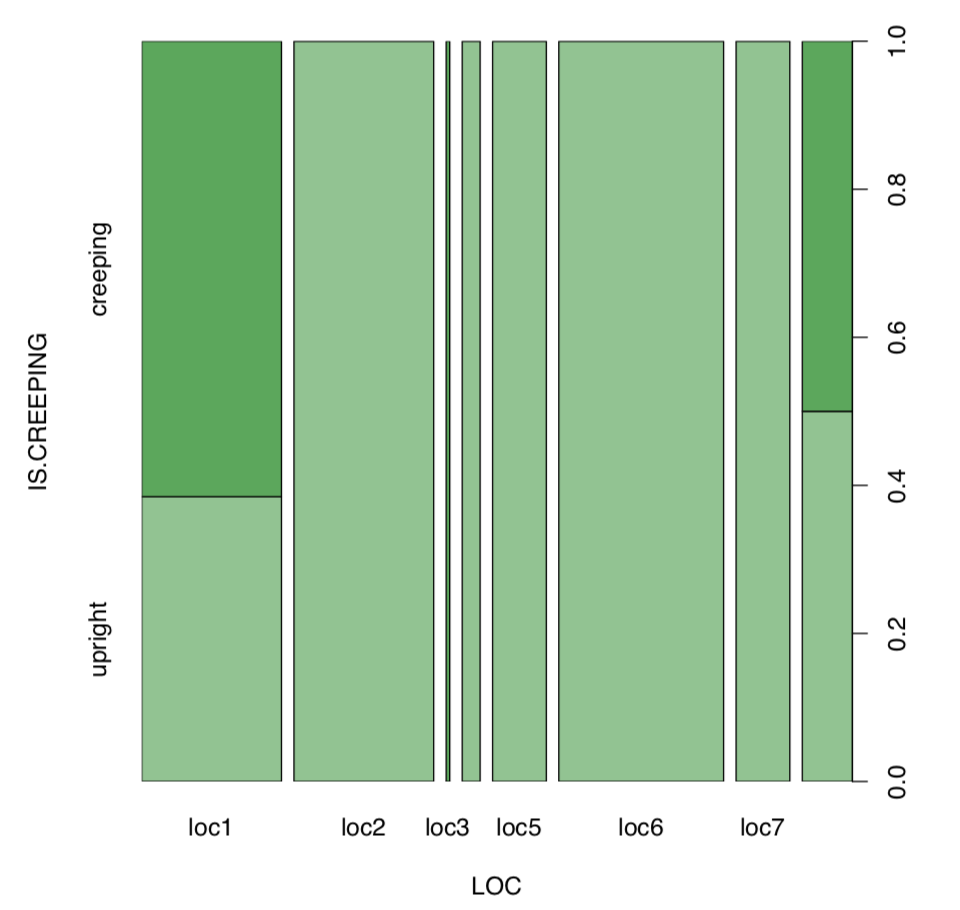
El siguiente paso es el análisis visual (Figura\(\PageIndex{7}\)):
Código\(\PageIndex{30}\) (R):
Algunas ubicaciones se ven diferentes. Para analizar, necesitamos tabla de contingencia:
Código\(\PageIndex{31}\) (R):
Ahora la prueba y el tamaño del efecto:
Código\(\PageIndex{32}\) (R):
(Ejecute Pairwise.table2.test (cc.lc) usted mismo para comprender las diferencias en los detalles.)
Sí, existe una asociación grande y estadísticamente significativa entre la localidad y la forma de vida del pasto escorbuto.
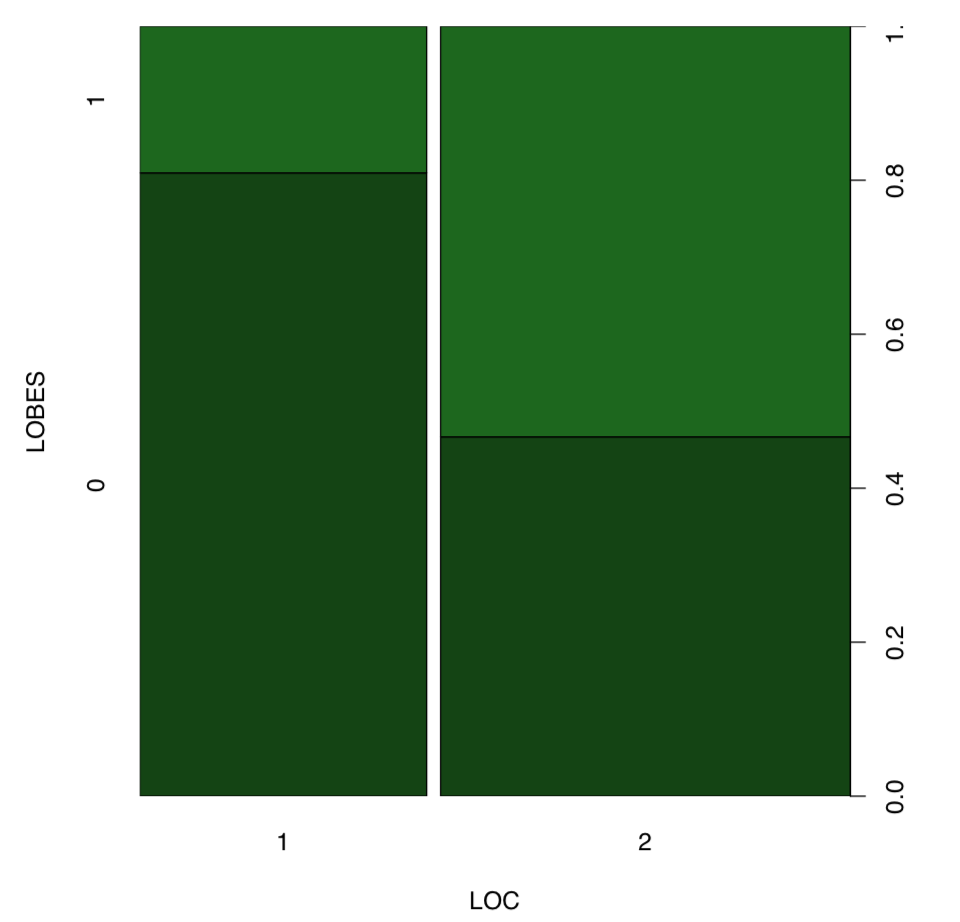
Respuesta a la pregunta sobre la igualdad de proporciones de carácter LOBES en dos localidades de abedul. Primero, debemos seleccionar estas dos localidades (1 y 2) y contar allí las proporciones. La forma más corta es usar la función table ():
Código\(\PageIndex{33}\) (R):
La gráfica de columna vertebral (Figura\(\PageIndex{8}\)) ayuda a hacer que las diferencias en la tabla sean aún más evidentes:
Código\(\PageIndex{34}\) (R):
(Tenga en cuenta también cómo crear dos colores intermedios entre negro y verde oscuro).
La elección más natural es prop.test () que es aplicable directamente a la salida de table ():
Código\(\PageIndex{35}\) (R):
En lugar de la prueba de proporción, podemos usar Fisher exacto:
Código\(\PageIndex{36}\) (R):
... o chi-cuadrado con simulación (tenga en cuenta que una celda tiene solo 4 casos), o con corrección por defecto de Yates:
Código\(\PageIndex{37}\) (R):
Con todo, sí, las proporciones de plantas con diferente posición de lóbulos son diferentes entre la ubicación 1 y 2.
¿Y qué pasa con el tamaño del efecto de esta asociación?
Código\(\PageIndex{38}\) (R):
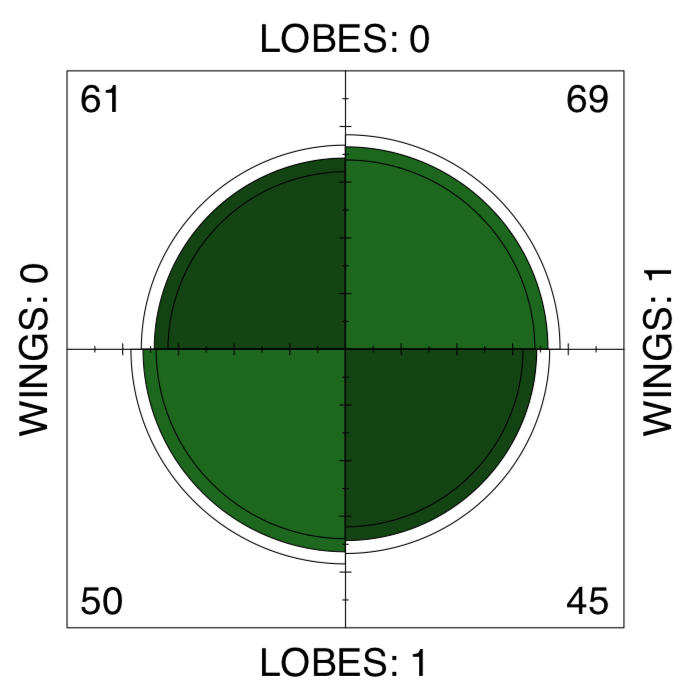
Respuesta a la pregunta sobre la igualdad de proporciones en todo el conjunto de datos de betula. Primero, haz mesa:
Código\(\PageIndex{39}\) (R):
No hay asimetría aparente. Dado que betula.lw es\(2\times2\) tabla, podemos aplicar parcela cuádruple. Muestra diferencias no solo como diferentes tamaños de sectores, sino que también permite verificar el intervalo de confianza del 95% con anillos marginales (Figura\(\PageIndex{9}\)):
Código\(\PageIndex{40}\) (R):
Tampoco sugerente... Por último, tenemos que poner a prueba la asociación, si la hay. Noe que las muestras están relacionadas. Esto se debe a que se midieron los Lóbulos y ALAS en las mismas plantas. Por lo tanto, en lugar de la prueba de chi-cuadrado o proporción deberíamos ejecutar la prueba de McNemar:
Código\(\PageIndex{41}\) (R):
Se concluye que las proporciones de dos estados de carácter en cada uno de los caracteres no son estadísticamente diferentes.