10.3: R y Bootstrapping
- Page ID
- 149951
Todas las generalidades como desviación estándar y media se toman normalmente de la muestra pero pretenden representar a toda la población estadística. Por lo tanto, es posible que estas estimaciones puedan estar seriamente equivocadas. Se diseñaron técnicas estadísticas como bootstrapping para minimizar el riesgo de estos errores. Bootstrap se basa únicamente en la muestra dada pero trata de estimar toda la población.
La idea de bootstrap se inspiró en Buerger y Raspe “Las aventuras milagrosas del barón Munchausen”, donde el personaje principal se saca (junto con su caballo) de un pantano por su cabello (Figura\(\PageIndex{1}\)). El bootstrap estadístico fue promovido activamente por Bradley Efron desde la década de 1970, pero no se utilizó con frecuencia hasta la década de 2000 porque es computacionalmente intensivo. En esencia, bootstrap es la estrategia de re-muestreo que reemplaza parte de la muestra con la submuestra propia. En R, podemos simplemente muestrear () nuestros datos con el reemplazo.

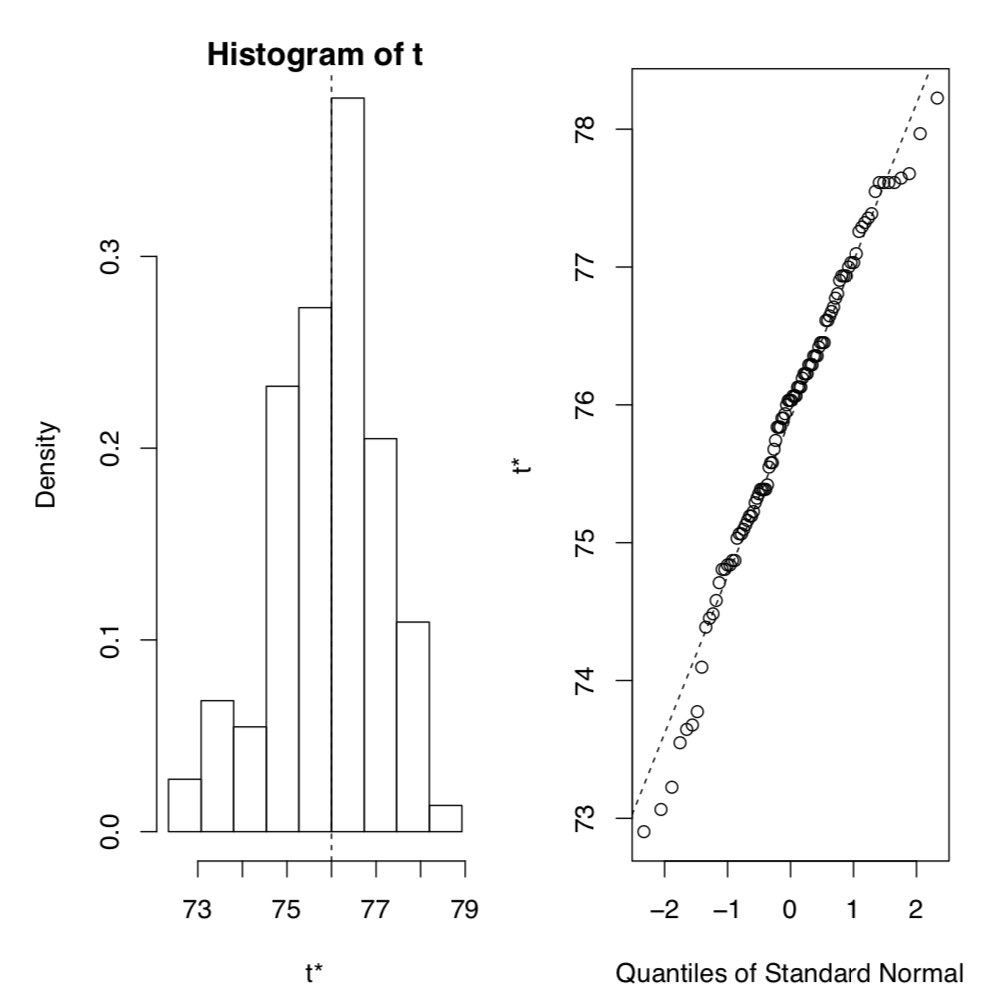
Primero, arrancaremos la media (Figura\(\PageIndex{2}\)) usando el paquete de arranque avanzado:
Código\(\PageIndex{1}\) (R):
(Tenga en cuenta que aquí y en muchos otros lugares en este libro el número de réplicas es de 100. Para los fines de trabajo, sin embargo, recomendamos que sea de al menos 1,000.)
Código\(\PageIndex{2}\) (R):
El arranque del paquete permite calcular el intervalo de confianza del 95%:
Código\(\PageIndex{3}\) (R):
Paquete bootstrap más básico bootstraps de una manera más sencilla. Para demostrarlo, utilizaremos el archivo de datos spur.txt. Estos datos son el resultado de mediciones de longitud de espolón en 1511 flores de orquídea Dactylorhiza. La longitud del espolón es importante porque solo los polinizadores con partes de la boca comparables a la longitud del espolón pueden polinizar con éxito estas flores.
Código\(\PageIndex{4}\) (R):
Jackknife es similar al bootstrap pero en ese caso las observaciones estarán sacando de la muestra una por una sin reemplazo:
Código\(\PageIndex{5}\) (R):
Esto es posible arrancar la desviación estándar y la media de estos datos incluso sin ningún paquete adicional, con para ciclo y muestra ():
Código\(\PageIndex{6}\) (R):
(Alternativamente, tt podría ser un marco de datos vacío, pero de esta manera toma más tiempo de computadora, lo cual es importante para bootstrap. Lo que hicimos anteriormente, es la pre-asignación, forma útil de ahorrar tiempo y memoria.)
En realidad, la distribución de longitud de espolón no sigue la ley normal (compruébelo usted mismo) Es mejor entonces estimar la mediana y mediana de la desviación absoluta (en lugar de la media y la desviación estándar), o la mediana y el rango del 95%:
Código\(\PageIndex{7}\) (R):
(Tenga en cuenta el uso de la función replicate (), este es otro miembro de la familia apply ().)
Este enfoque permite también arrancar casi cualquier medida. Permítanos, por ejemplo, arrancar el intervalo de confianza del 95% para K de Lyubishchev:
Código\(\PageIndex{8}\) (R):
Bootstrap y jackknife están relacionados con numerosas técnicas de remuestreo. Hay múltiples paquetes R (como monedas) que proporcionan pruebas de remuestreo y procedimientos relacionados:
Código\(\PageIndex{9}\) (R):
Bootstrap también es ampliamente utilizado en el aprendizaje automático. Arriba había un ejemplo de la función Jclust () del conjunto asmisc.r. También hay BootA (), Bootrf () y BootKnN () para boorstrap resultados no supervisados y supervisados.
) plantas. Utilice los métodos bootstrap y remuestreo.


