1.1: Introducción y Objetivos
- Page ID
- 54868
curso de biología computacional
Estas notas de clase tienen como objetivo ser impartidas como un curso de término sobre biología computacional, cada conferencia de 1.5 horas cubriendo un capítulo, junto con tareas quincenales y sesiones de tutoría para ayudar a los estudiantes a lograr sus propios proyectos de investigación independientes. Las notas surgieron del curso del MIT 6.047/6.878, y reflejan muy de cerca la estructura de las conferencias correspondientes.
Dualidad de Metas: Fundamentos y Fronteras
Hay dos metas para este curso. El primer objetivo es introducirte en las bases del campo de la biología computacional. A saber, introducir los problemas biológicos fundamentales del campo, y aprender las técnicas algorítmicas y de aprendizaje automático necesarias para abordarlos. Esto va más allá de aprender a usar los programas y herramientas en línea que son populares en cualquier año dado. En cambio, el objetivo es que entiendas los principios subyacentes de las técnicas más exitosas que están actualmente en uso, y brindarte la capacidad de diseñar e implementar la próxima generación de herramientas. Esa es la razón por la que una clase de algoritmos introductorios se establece como pre-req; la mejor manera de obtener una comprensión más profunda de los algoritmos presentados es implementarlos usted mismo.
El segundo objetivo del curso es abordar las fronteras de investigación de la biología computacional, y de eso se tratan realmente todos los temas avanzados y tareas prácticas. De hecho, nos gustaría darte una idea de cómo funciona la investigación, exponerte a las direcciones de investigación actuales, guiarte para encontrar los problemas más interesantes para ti y ayudarte a convertirte en un practicante activo en el campo. Esto se logra a través de conferencias invitadas, conjuntos de problemas, laboratorios y, lo más importante, un proyecto de investigación independiente a largo plazo, donde realiza su investigación independiente.
Los módulos del curso siguen ese patrón, cada uno consistente en conferencias que cubren los fundamentos y las fronteras de cada tema. Las conferencias fundacionales introducen los problemas clásicos en el campo. Estos problemas se entienden muy bien y ya se han encontrado soluciones elegantes; algunos incluso se han enseñado desde hace más de una década. La parte de fronteras del módulo abarca temas avanzados, generalmente abordando cuestiones centrales que aún permanecen abiertas en el campo. Estos capítulos suelen incluir conferencias invitadas de algunos de los pioneros en cada área que hablan tanto sobre el estado general del campo como de la investigación de su propio laboratorio.
Las asignaciones para el curso siguen el mismo patrón de fundación/fronteras. La mitad de las tareas van a consistir en elaborar los métodos con lápiz sobre papel y profundizar en las nociones algorítmicas y de aprendizaje automático de los problemas. La otra mitad en realidad van a ser preguntas prácticas consistentes en asignaciones de programación, donde se proporcionan conjuntos de datos reales. Analizarás estos datos usando las técnicas que has aprendido e interpretarás tus resultados, dándote una experiencia real. Las tareas se construyen hasta el proyecto final, donde propondrás y llevarás a cabo un proyecto de investigación original, y presentarás tus hallazgos en formato conferencia. En general, las asignaciones están diseñadas para darle la oportunidad de aplicar métodos de biología computacional a problemas reales en biología.
Dualidad de disciplinas: Computación y Biología
Además de apuntar a cubrir tanto fundaciones como fronteras, la otra dualidad importante de este curso es entre cómputos y biología.
Desde la perspectiva biológica del curso, nuestro objetivo es enseñar temas que son fundamentales para nuestra comprensión de la biología, la medicina y la salud humana. Por lo tanto, rehuimos cualquier problema computacionalmente interesante que sea de inspiración biológica, pero que no sea relevante para la biología. No solo vamos a ver algo en biología, a inspirarnos, y luego ir a la informática y hacer muchas cosas que a la biología nunca le importarán. En cambio, nuestro objetivo es trabajar en problemas que puedan hacer un cambio significativo en el campo de la biología. Nos gustaría que publicaran artículos que realmente importan a la comunidad biológica y que tengan un impacto biológico real. Por lo tanto, este objetivo ha guiado la selección de temas para el curso, y cada capítulo se centra en un problema biológico fundamental.
Desde la perspectiva computacional del curso, siendo después de todo una clase de informática, nos enfocamos en explorar técnicas y principios generales que sin duda son importantes en la biología computacional, pero que no obstante pueden aplicarse en cualquier otro campo que requiera análisis e interpretación de datos. De ahí que si lo que quieres es adentrarte en cosmología, meteorología, geología, o algo así, esta clase ofrece técnicas computacionales que probablemente serán útiles cuando se trata de conjuntos de datos del mundo real relacionados con esos campos.
¿Por qué Biología Computacional?
lecture1_transcript.html #Motivations
Son muchas las razones por las que la Biología Computacional ha surgido como una disciplina importante en los últimos años, y quizás algunas de estas te llevan a recoger este libro o registrarte en esta clase. A pesar de que tenemos nuestra propia opinión sobre cuáles son estas razones, año con año hemos pedido a los estudiantes su propia visión sobre lo que ha permitido que el campo de la Biología Computacional se expanda tan rápidamente en los últimos años. Sus respuestas caen en varios temas amplios, que resumimos aquí.
- Quizás la razón más fundamental por la que los enfoques computacionales son tan adecuados para el estudio de los datos biológicos es que en su núcleo, los sistemas biológicos son fundamentalmente de naturaleza digital. Para ser contundentes, los humanos no son los primeros en construir una computadora digital; nuestros antepasados son la primera computadora digital, ya que las primeras formas de vida basadas en ADN ya estaban almacenando, copiando y procesando información digital codificada en las letras A, C, G y T. La mayor ventaja evolutiva de un medio digital para almacenar información genética es que puede persistir a lo largo de miles de generaciones, mientras que las señales analógicas se diluirían de generación en generación a partir de la difusión química básica.
- Además del ADN, muchos otros aspectos de la biología son digitales, como los interruptores biológicos, que aseguran que solo se logren dos estados discretos posibles mediante bucles de retroalimentación y procesos metaestables, aunque estos sean implementados por niveles de moléculas. Los extensos circuitos de retroalimentación y otros circuitos regulatorios diversos implementan decisiones discretas a través de componentes que de otro modo serían inestables, nuevamente con principios de diseño similares a la práctica de ingeniería, haciendo que nuestra búsqueda por comprender los sistemas biológicos desde una perspectiva de ingeniería sea
- Las ciencias que se benefician mucho del procesamiento de datos, como la Biología Computacional, siguen un ciclo virtuoso que involucra los datos disponibles para su procesamiento. Cuanto más se pueda hacer procesando y analizando los datos disponibles, más financiamiento se destinará al desarrollo de tecnologías para obtener, procesar y analizar aún más datos. Las nuevas tecnologías, como la secuenciación y las técnicas experimentales de alto rendimiento, como los ensayos de micromatrices, dos híbridos de levadura y Chip-chip, están creando cantidades enormes y crecientes de datos que pueden analizarse y procesarse usando técnicas computacionales. Los proyectos genómicos de $1000 y $100 son evidencia de este ciclo. Hace más de diez años, cuando comenzaron estos proyectos, hubiera sido absurdo incluso imaginar procesar cantidades tan masivas de datos. Sin embargo, a medida que se idearon más ventajas potenciales a partir del procesamiento de estos datos, se dedicó más financiamiento al desarrollo de tecnologías que harían factibles estos proyectos.
- La capacidad de procesar datos ha mejorado mucho en los últimos años, debido a: 1) el enorme poder compu- tacional disponible en la actualidad (debido a la ley de Moore, entre otras cosas), y 2) los avances en las técnicas algorítmicas en cuestión.
- Los enfoques de optimización se pueden utilizar para resolver, a través de técnicas computacionales, que de otra manera son problemas in- tratables.
- Las consideraciones de tiempo de ejecución y memoria son críticas cuando se trata de enormes conjuntos de datos. Un algoritmo que funcione bien en un genoma pequeño (por ejemplo, una bacteria) podría ser demasiado ineficiente en el tiempo o en el espacio para ser aplicado a 1000 genomas de mamíferos. Además, las preguntas combinatorias aumentan drásticamente la complejidad algorítmica.
- Los conjuntos de datos biológicos pueden ser ruidosos y filtrar la señal del ruido es un problema computacional.
- Los enfoques de aprendizaje automático son útiles para hacer inferencias, clasificar características biológicas e identificar
señales robustas.
- A medida que se profundiza nuestra comprensión de los sistemas biológicos, hemos comenzado a darnos cuenta de que tales sistemas no pueden analizarse aisladamente. Estos sistemas han demostrado estar entrelazados de formas antes inauditas, y hemos comenzado a cambiar nuestros análisis a técnicas que los consideran todos como un todo.
- Es posible utilizar enfoques computacionales para encontrar correlaciones de manera imparcial, y llegar a conclusiones que transformen el conocimiento biológico y faciliten el aprendizaje activo. Este enfoque se llama descubrimiento basado en datos.
- Los estudios computacionales pueden predecir hipótesis, mecanismos y teorías para explicar observaciones experimentales. Estas hipótesis falsificables pueden entonces ser probadas experimentalmente.
- Los enfoques computacionales pueden ser utilizados no solo para analizar datos existentes sino también para motivar la recolección de datos y sugerir experimentos útiles. Además, el filtrado computacional puede estrechar el espacio de búsqueda experimental para permitir diseños experimentales más enfocados y eficientes.
- La biología tiene reglas: La evolución está impulsada por dos reglas simples: 1) mutación aleatoria y 2) selección brutal. Los sistemas biológicos están limitados a estas reglas, y al analizar datos, buscamos encontrar e interpretar el comportamiento emergente que estas reglas generan.
- Los conjuntos de datos se pueden combinar utilizando enfoques computacionales, de modo que la información recopilada a través de múltiples experimentos y el uso de diversos enfoques experimentales se pueda llevar a cabo en cuestiones de interés.
- Las visualizaciones efectivas de los datos biológicos pueden facilitar el descubrimiento.
- Los enfoques computacionales se pueden usar para simular y modelar datos biológicos.
- Los enfoques computacionales pueden ser más éticos. Por ejemplo, algunos experimentos biológicos pueden ser poco éticos para realizar en sujetos vivos pero podrían ser simulados por una computadora.
- A gran escala, los enfoques de ingeniería de sistemas se ven facilitados por la técnica computacional para obtener visiones globales sobre el organismo que son demasiado complejas para analizarlas de otra manera.
Encontrar elementos funcionales: una pregunta de biología computacional
lecture1_transcript.html #Codons
Varios problemas de biología computacional se refieren a encontrar señales biológicas en datos de ADN (por ejemplo, regiones codificantes, promotores, potenciadores, reguladores,...).
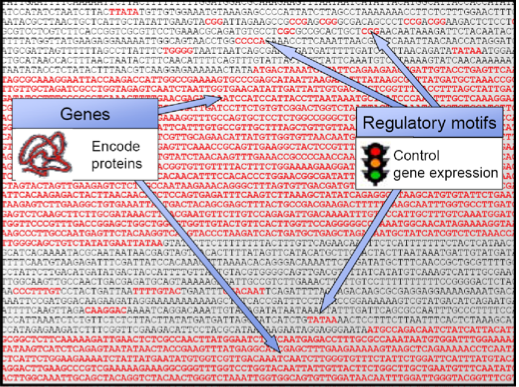
Figura 1.1: En este problema de biología computacional, se nos proporciona una secuencia de bases, y se desea localizar genes y motivos reguladores.
Luego discutimos una pregunta específica que la biología computacional puede ser utilizada para abordar: ¿cómo se pueden encontrar elementos funcionales en una secuencia genómica? La Figura 1.1 muestra parte de la secuencia del genoma de la levadura. Dada esta secuencia, podemos preguntar:
P: ¿Cuáles son los genes que codifican las proteínas?
R: Durante la traducción, el codón de inicio marca el primer aminoácido en una proteína, y el codón de parada indica el final de la proteína. Sin embargo, como se indica en el portaobjetos “Extracción de señal del ruido”, solo algunas de estas secuencias ATG en el ADN marcan realmente el inicio de un gen que se expresará como proteína. Los otros son “ruido”; por ejemplo, pueden haber formado parte de intrones (secuencias no codificantes que se empalman después de la transcripción).
P: ¿Cómo podemos encontrar características (genes, motivos reguladores y otros elementos funcionales) en la secuencia genómica?
R: Estas preguntas podrían abordarse ya sea experimentalmente o computacionalmente. Un enfoque experimental del problema sería crear un knockout, y ver si se ve afectada la aptitud del organismo. También podríamos abordar la cuestión computacionalmente viendo si la secuencia se conserva a través de los genomas de múltiples especies. Si la secuencia se conserva significativamente a lo largo del tiempo evolutivo, es probable que realice una función importante.
Hay advertencias en ambos enfoques. Quitar el elemento puede no revelar su función, incluso si no hay diferencia aparente con respecto al original, esto podría deberse simplemente a que no se han probado las condiciones adecuadas. Además, el simple hecho de que un elemento no se conserve no significa que no sea funcional. (También, tenga en cuenta que “elemento funcional” es un término ambiguo. Ciertamente, hay muchos tipos de elementos funcionales en el genoma que no son codificantes de proteínas. Curiosamente, 90-95% del genoma humano se transcribe (se usa como molde para hacer ARN). No se sabe cuál es la función de la mayoría de estas regiones transcritas, o de hecho si son funcionales).


