1.5: Introducción a algoritmos e inferencia probabilística
- Page ID
- 54853
1. Rápidamente revisaremos alguna probabilidad básica considerando una forma alternativa de representar motivos: una matriz de peso de posición (PWM). Nos gustaría modelar el hecho de que las proteínas pueden unirse a motivos que no están completamente especificados. Es decir, algunas posiciones pueden requerir un cierto nucleótido (por ejemplo, A), mientras que otras posiciones son libres para ser un subconjunto de los 4 nucleótidos (por ejemplo, A o C). Un PWM representa el conjunto de todas las secuencias de ADN que pertenecen al motivo mediante el uso de una matriz que almacena la probabilidad de encontrar cada uno de los 4 nucleótidos en cada posición en el motivo. Por ejemplo, considere el siguiente PWM para un motivo con longitud 4:
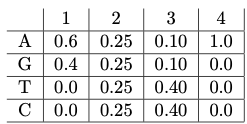
Decimos que este motivo puede generar secuencias de longitud 4. Los PWM suelen suponer que la distribución de una posición no está influenciada por la base de otra posición. Observe que cada posición está asociada con una distribución de probabilidad sobre los nucleótidos (suman a 1 y no son negativos).
2. También podemos modelar la distribución de fondo de los nucleótidos (la distribución que se encuentra a través del genoma):
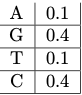
Observe cómo las probabilidades para A y T son las mismas y las probabilidades de G y C son las mismas. Esto es consecuencia de la complementariedad del ADN que asegura que la composición global de A y T, G y C es la misma en general en el genoma.
3. Considera la secuencia\(S = GCAA.\)
- La probabilidad de que el motivo genere esta secuencia es\[P(S|M) = 0.4 × 0.25 × 0.1 × 1.0 = 0.01. \nonumber\]
- La probabilidad de que el fondo genere esta secuencia\[P (S|B) = 0.4 × 0.4 × 0.1 × 0.1 = 0.0016. \nonumber\]
4. Solo esto no es particularmente interesante. Sin embargo, dada la fracción de secuencias que son generadas por el motivo, por ejemplo P (M) = 0.1, y suponiendo que todas las demás secuencias son generadas por el fondo (P (B) = 0.9) podemos calcular la probabilidad de que el motivo genere la secuencia usando la Regla de Bayes:
\[\begin{align*} P(M|S) &= \frac{P(S|M)P(M)}{P(S)} \\[4pt] &= \frac{P(S|M)P(M)}{P(S|B)P(B)+P(S|M)P(M)} \\[4pt] &= \frac{0.01 \times 0.1}{0.0016 \times 0.9 + 0.01 \times 0.1} = 0.40984 \end{align*}\]


