21.4: Aplicación de Redes
- Page ID
- 54992
Utilizando árboles de regresión lineal y regresión, intentaremos predecir la expresión a partir de redes. Mediante la clasificación colectiva y el etiquetado de relajación, intentaremos asignar función a elementos desconocidos de la red.
Nos gustaría utilizar redes para:
- predecir la expresión de genes a partir de reguladores.
En la predicción de la expresión, el objetivo es parametrizar una relación dando niveles de expresión génica a partir de los niveles de expresión del regulador. Se puede resolver de diversas maneras incluyendo regresión y se relaciona con el problema de encontrar redes funcionales.
- predecir funciones para genes desconocidos.
Descripción general de los modelos funcionales
Un modelo de predicción es un gaussiano condicional: un modelo simple entrenado por regresión lineal. Un modelo de predicción más complejo es un árbol de regresión entrenado por regresión no lineal.
Modelos gaussianos condicionales
Los modelos gaussianos condicionales predicen sobre un espacio continuo y son entrenados por una regresión lineal simple para maximizar la probabilidad de datos. Predicen dianas cuyos niveles de expresión son medios de gaussianos sobre reguladores.
El aprendizaje gaussiano condicional toma una red estructurada y dirigida con objetivos y factores de transcripción reguladores. Puede estimar los parámetros gaussianos, μ, a partir de los datos encontrando parámetros que maximizan la probabilidad; después de una derivada, el enfoque ML se reduce a resolver una ecuación lineal.
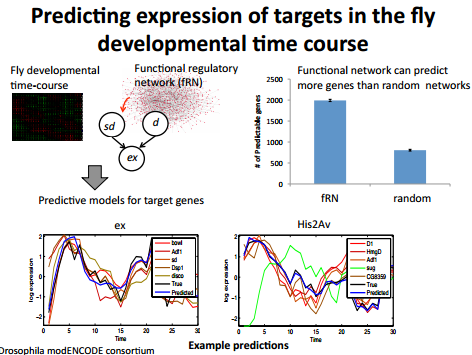
A partir de una red reguladora funcional derivada de múltiples fuentes de datos 6, el Dr. Roy entrenó un modelo gaussiano para la predicción utilizando datos de expresión de curso de tiempo y lo probó en un conjunto de pruebas de suspensión. En comparaciones con predicciones por un modelo entrenado a partir de una red aleatoria, se encontró que la red predijo sustancialmente mejor que al azar.
El modelo lineal utilizado hace una fuerte suposición sobre la linealidad de la interacción. Probablemente esta no sea una suposición muy precisa pero parece funcionar hasta cierto punto con el conjunto de datos probado.
Modelos de árbol de regresión
Los modelos de árbol de regresión permiten al modelador utilizar una distribución multimodal incorporando dependencias no lineales entre el regulador y la expresión del gen diana. La estructura final de un árbol de regresión describe la gramática de expresión en términos de una serie de elecciones realizadas en los nodos del árbol de regresión. Debido a que los objetivos pueden compartir programas regulatorios, se pueden incorporar nociones de motivos recurrentes. Los árboles de regresión son modelos ricos pero difíciles de aprender. Árboles de regresión en la predicción de expresión
En la práctica, la predicción se abre paso por un árbol de regresión dados los niveles de expresión del regulador. Al llegar a los nodos foliares del árbol de regresión, se realiza una predicción para la expresión génica.
Predicción funcional para nodos no anotados
Dada una red con un conjunto incompleto de etiquetas, el objetivo de la anotación de funciones es predecir etiquetas para genes desconocidos. Utilizaremos métodos que caen dentro de la amplia categoría de culpabilidad por asociación. Si no sabemos nada de un nodo sino que sus vecinos están involucrados en una función, asigne esa función al nodo desconocido.
La asociación puede incluir cualquier noción de relación de red discutida anteriormente, tal como coexpresión, interacciones proteína-proteína y corregulación. Muchos métodos funcionan, dos serán discutidos: clasificación colectiva y clasificación de relajación; ambos funcionan para redes regulatorias codificadas como gráficas no dirigidas.
Clasificación Colectiva
Ver la predicción funcional como un problema de clasificación: Dado un nodo, ¿cuál es su clase reguladora?.
fuente desconocida. Todos los derechos reservados. Este contenido está excluido de nuestro Creativo
Licencia Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
Para utilizar la estructura gráfica en el problema de predicción, capturamos propiedades de la vecindad de un gen en atributo relacional. Dado que todos los puntos están conectados en una red, los puntos de datos ya no están distribuidos inde- penalmente - el problema de predicción se vuelve sustancialmente más difícil que un problema de clasificación estándar.
La clasificación iterativa es un método sencillo con el que resolver el problema de clasificación. Comenzando con una suposición inicial para genes no etiquetados, infiere etiquetas iterativamente, permitiendo que las etiquetas cambiadas influyan en las predicciones de etiquetas de nodo de una manera similar al muestreo de gibbs 7
El etiquetado de relajación es otro enfoque originalmente desarrollado para trac redes terroristas. El modelo utiliza un puntaje de sospecha donde los nodos son etiquetados con una desconfianza de acuerdo con la desconfianza de sus vecinos. El método se llama etiquetado de relajación porque gradualmente se asienta en una solución de acuerdo con un parámetro de aprendizaje. Es otra instancia de aprendizaje iterativo donde a los genes se les asignan probabilidades de tener una función dada.
Redes reglamentarias para la predicción de funciones
Para pares de nodos, calcule una similitud regulatoria —la cantidad de interacción— igual al tamaño de la intersección de sus reguladores dividido por el tamaño de su unión. Al tener esta similitud de interacción en forma de gráfica no dirigida sobre objetivos de red, se pueden utilizar clústeres derivados de una red en la clasificación funcional final.
El modelo tiene éxito en la predicción del desarrollo del disco invaginal y del sistema neural. La línea azul de la Fig. 21.2a muestra la puntuación de cada gen que predice su participación en el desarrollo del sistema neural.
La coexpresión y corregulación se puede utilizar lado a lado para aumentar el conjunto de genes conocidos por participar en el desarrollo del sistema neural.
6 fuentes de datos incluyeron cromatina, unión física, expresión, motivo
7 ver la conferencia anterior de Manolis describiendo el descubrimiento de motivos


