27.5: Inferir ortológicos/Parálogos, Duplicación y Pérdida Genética
- Page ID
- 54463
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Hay dos árboles de uso común, Árbol de especies y Árbol de genes. En esta sección se explica cómo se pueden usar estos árboles y cómo encajar un árbol genético dentro de un árbol de especies (reconciliación).
Especies Árbol
Especies arbóreas que muestran cómo diferentes especies evolucionaron entre sí. Estos árboles se crean usando caracteres morfológicos, evidencia fósil, etc. Las hojas de cada árbol están etiquetadas como especies y el resto del árbol muestra cómo se relacionan estas especies. Un ejemplo de árbol especie se muestra en la Figura 27.1. Nota: en conferencia se menciona que una especie puede ser pensada como una” bolsa de genes”, es decir, el grupo de genes comunes entre los miembros de una especie.
Árbol de genes

Los árboles génicos son árboles que miran genes específicos en diferentes especies. Las hojas de los árboles génicos están marcadas con secuencias génicas o identificadores de genes asociados con secuencias específicas. La Figura 27.2 muestra un ejemplo de un árbol génico que tiene 4 genes (hojas). Las secuencias asociadas a cada gen se presentan en el lado derecho de la Figura 27.2.
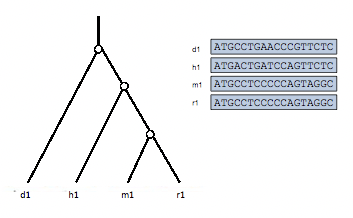
Evolución de la Familia Génica
Los árboles genéticos evolucionan dentro de un árbol de especies. Un ejemplo de un árbol génico contenido en un árbol de especie se muestra en la Figura 27.3 a continuación.

En la siguiente subsección se explica cómo podemos encajar árboles genéticos dentro de árboles de una especie usando Reconciliation.
Reconciliación
La reconciliación es un algoritmo que ayuda a comparar árboles genéticos con árboles genómicos ajustando un árbol genético dentro de un árbol de especies. Esto se hace mapeando los vértices en el árbol de genes a vértices en el árbol de la especie. Esta subsección se centrará en la Reconciliación, definiciones relacionadas, algoritmos (Conciliación de Parsimonia Máxima y SPIDIR) y ejemplos.
Definiciones
Dos genes son ortólogos si su ancestro común más reciente (MRCA) es una especiación (dividiéndose en diferentes especies).
Los parálogos son genes cuyo MRCA es una duplicación.
La Figura 27.4 a continuación ilustra cómo se pueden representar estos tipos de genes en un árbol génico. El árbol de abajo tiene 4 nodos de especiación, una duplicación y una pérdida.

Un diagrama de mapeo es un diagrama que muestra el mapeo de nodos desde el árbol génico hasta el árbol de especies. La Figura 27.5 muestra un ejemplo de un diagrama de mapeo.
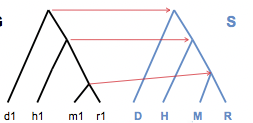
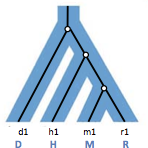
Un diagrama de anidación muestra cómo se puede anidar el árbol genético dentro del árbol de la especie. Para cada diagrama de mapeo hay un diagrama de anidamiento. La Figura 27.6 muestra un ejemplo de un posible diagrama de anidamiento para el diagrama de mapeo de la Figura 27.5.
Algoritmo de reconciliación máxima de parsimonia (MPR)
MPR es un algoritmo que ajusta un árbol génico en un árbol de especies mientras minimiza el número de duplicaciones y deleciones.

Dado un árbol genético y un árbol de especies, el algoritmo encuentra la reconciliación que minimiza el número de duplicaciones y deleciones. La Figura 27.7 anterior muestra un ejemplo de un posible mapeo de un árbol génico a un árbol de especies. La Figura 27.8 presenta el pseudocódigo para el algoritmo MPR. El caso base consiste en emparejar las hojas del árbol génico con las hojas del árbol de la especie; el algoritmo luego progresa por los vértices del árbol génico, dibujando una relación entre el MRCA de todas las hojas dentro del subárbol de un vértice dado y el vértice MRCA correspondiente en el árbol de la especie. En el pseudocódigo, I (G) representa el árbol de especies y L (G) representa el árbol génico.

Mapeamos las flechas lo más bajo posible, ya que un mapeo más bajo generalmente da como resultado menos eventos. Sin embargo, no podemos mapear demasiado bajo. Mapear demasiado bajo significa que estamos violando la restricción de que el MRCA de un nodo dado es al menos tan alto como el MRCA de sus hijos. Mapeamos lo más bajo posible sin violar las relaciones descendentes-ancestros. El algoritmo va recursivamente de abajo hacia arriba, comenzando desde las hojas. Dado que tomamos muestras de genes de especies conocidas para construir el árbol genético, existe un mapeo directo entre las hojas del árbol genético y las hojas del árbol de la especie. Para mapear a los antepasados, para cada nodo (subiendo recursivamente por el árbol) observamos al niño derecho y al niño izquierdo y tomamos el ancestro menos común (LCA) de las especies a las que mapean. Si un nodo se mapea a su hijo derecho o izquierdo, sabemos que hay una duplicación. Una rama esperada que no existe indica una pérdida.
Ejemplos de reconciliación
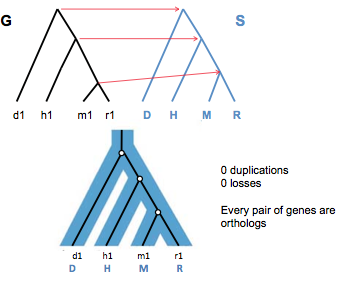

En la Figura 27.10, vemos una reconciliación parsimoniosa (número mínimo de pérdidas y duplicaciones) para un caso en el que los nodos del árbol de genes no pueden mapearse directamente a través. Esto es el resultado de las ubicaciones intercambiadas de h1 y d1 en el árbol génico; el ancestro menos común para d1, m1 y r1 es ahora el vértice raíz del árbol de la especie.
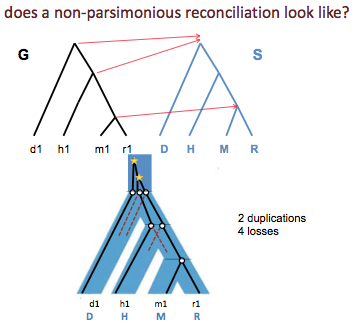
La Figura 27.11 muestra una reconciliación no parsimoniosa. El mapeo parsimonioso para los mismos árboles se muestra en la Figura 27.9.
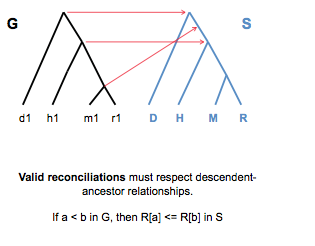
La Figura 27.12 muestra una reconciliación no válida. Esta reconciliación no es válida ya que no respeta las relaciones descendentes-ancestros. Para que esta reconciliación sea posible, el descendiente tendría que viajar atrás en el tiempo y ser creado ante su antepasado. Claramente, tal escenario sería imposible. Una conciliación válida debe satisfacer lo siguiente: Si a < b en G, entonces R [a]\(\leq\) R [b] en S.
Interpretación de ejemplos de reconciliación
Los árboles genéticos, cuando se reconcilian con árboles de especies, ofrecen una visión significativa de los eventos evolutivos (es decir, duplicaciones y pérdidas). Las duplicaciones describen que el mismo gen se encuentra en loci separados -m2 o r2, en esta situación- y es un mecanismo importante para crear nuevos genes y funciones. Estas consecuencias evolutivas se dividen en tres categorías: no funcionalización, neofuncionalización y subfuncionalización. La no funcionalización es bastante común y hace que una de las copias, como era de esperar, simplemente no funcione. La neofuncionalización es cuando una de las copias desarrolla una función completamente nueva. La subfuncionalización es cuando las copias conservan diferentes partes (dividiendo la mano de obra, de alguna manera), y juntas, realizan la misma función.
En la Figura 4, vemos que se produjo un evento de duplicación antes de la divergencia de ratones y ratas como especie. Es por ello que vemos genes similares tanto en m1 como en m2, que representan dos loci separados. d2 y h2 no están incluidos en la gráfica porque en el gen que se está considerando no está presente en esos loci (ya que no se produjo ningún evento de duplicación), mientras que es tanto en m2 como r2.
Si el evento de duplicación hubiera ocurrido un nivel superior en la Figura 4, sin ver un h2 correspondiente en el árbol génico, esto implicaría una pérdida dentro de la rama h del árbol de la especie.

