27.6: Reconstrucción
- Page ID
- 54434
En la sección anterior aprendimos a comparar y combinar árboles genéticos y árboles de especies. En esta sección, utilizaremos esta información para reconstruir árboles genéticos y árboles de especies.
Reconstrucción de árboles de especies
En el pasado, era muy difícil identificar un gen marcador que diera una idea de la diferenciación para una especie específica. A medida que mejoraba la secuenciación, comenzamos a tener muchos datos de secuenciación en varios genes. A partir de diferentes conjuntos de loci, las personas construyeron diferentes árboles, los cuales dependían en gran medida del conjunto de loci elegidos. Las posibles razones por las que los árboles difieren incluyen ruido (de errores de estimación estadística y ruido), duplicaciones y pérdidas ocultas, y clasificación de alelos en una población.
Problema de reconstrucción de árboles de especies
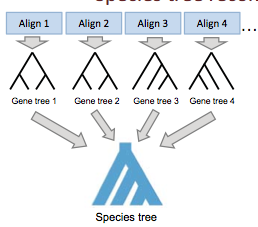
Dados muchos árboles genéticos diferentes que no están de acuerdo, nuestro objetivo es convertirlos en un árbol de una especie (como se muestra en la Figura 27.13. Hay muchos algoritmos diferentes que reconstruyen árboles de especies. Estos algoritmos incluyen métodos Supermatrix (Rokas 2003, Ciccareli 2006), métodos Supertree (Creevey & McInerney 2005), Minimizando la coalescencia profunda (Maddison & Knowles 2006) y Modelado de coalescencia (Liu & Pearl 2007).
Una forma de hacer esto, que en su mayoría es efectiva para datos ruidosos, es reunir más datos para aumentar la precisión. Esto se hace concatenando alineaciones de genes en una supermatriz.
Otro método consiste en construir un árbol para cada uno y usar un método de consenso para resumir estos árboles. Luego identificamos ramas análogas a través de la gran cantidad de árboles y construimos una especie de árbol que tiene las ramas que ocurren con mayor frecuencia.
Hay otra forma de reconstruir un árbol de especies, que es efectiva en caso de que los árboles genéticos no estén de acuerdo por duplicaciones y pérdidas. El objetivo es encontrar la especie arbórea que aplique la menor cantidad de duplicaciones. Construimos todos los árboles genéticos y luego proponemos un árbol de especies. A continuación, utilizamos la reconciliación para determinar el número de eventos que implica cada árbol génico combinado con la especie propuesta. Luego, proponemos árboles de otras especies y movemos ramas alrededor. Los árboles de especies equivocadas tienden a tener muchos eventos que no sucedieron. El árbol correcto debe tener el menor número de eventos.
Mejorando la reconstrucción de árboles genéticos y el aprendizaje entre árboles genéticos
Podemos usar métodos similares a los descritos anteriormente para construir mejores árboles genéticos. Esto se puede hacer usando información de un árbol de especies para estudiar un árbol genético de interés. Por ejemplo, los árboles de especies pueden ser utilizados para determinar cuándo ocurrieron pérdidas y duplicaciones. La idea es que podamos usar el hecho de que las especies arbóreas a menudo se construyen a partir de todo el genoma, para obtener más información sobre árboles genéticos relacionados. Podemos usar tanto la longitud de la sucursal como el número de eventos para hacer esto.
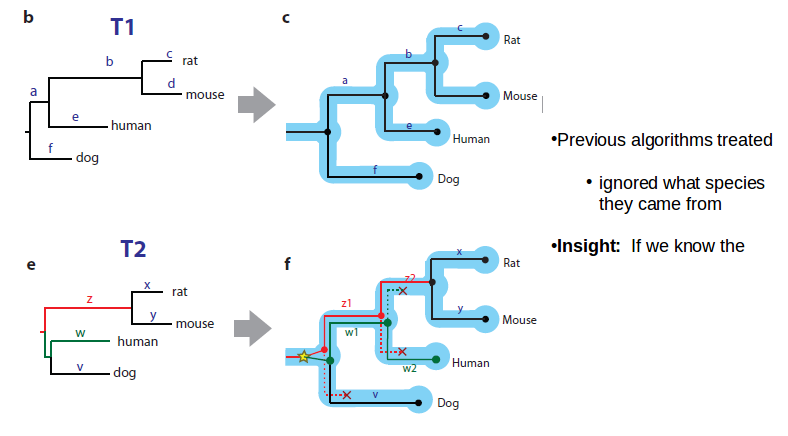
Si conocemos la especie arbórea, podemos desarrollar un modelo para qué tipo de longitudes de rama podemos esperar. Podemos usar el orden de genes conservados para contar ortólogos y construir árboles.
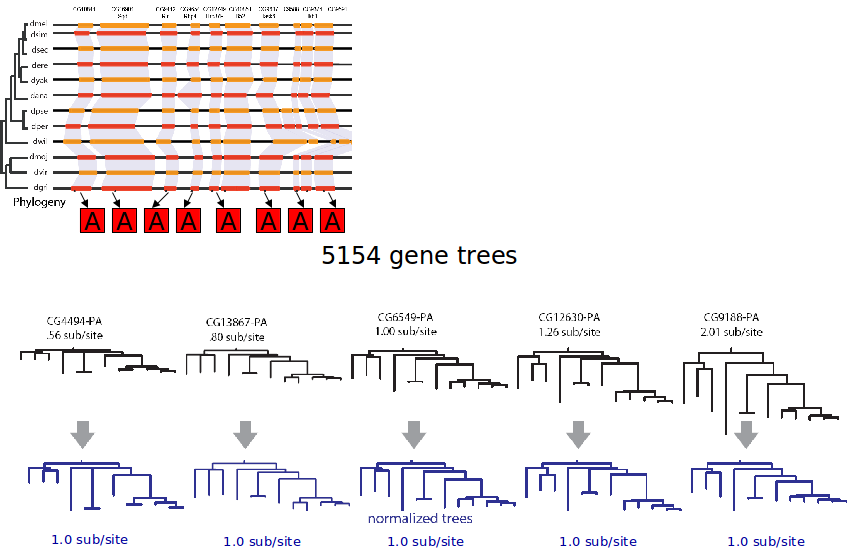
Cuando un gen está evolucionando rápidamente en una especie, está evolucionando rápidamente en todas las especies. Podemos modelar una longitud de rama como dos componentes de velocidad diferentes. Uno es específico de gen (presente en todas las especies) y el otro es específico de especie, que se personaliza a una especie específica.
%253D613.190.png)
Este método mejora enormemente la precisión de la reconstrucción.

