8.8: Locus RII de T4
- Page ID
- 57268
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Mapeo dentro de un gen: el locus RII
T2 y su pariente cercano T4 son virus que infectan a la bacteria E. coli. La infección termina con la destrucción (lisis) de la célula bacteriana por lo que estos virus son ejemplos de bacteriófagos (“comedores de bacterias”). Han sido enormemente útiles en estudios genéticos porque:
- Los virus de dos (o más) genotipos diferentes pueden infectar simultáneamente una sola bacteria.
- Las moléculas de ADN de uno de los virus infectantes pueden recombinarse con la de otro formando moléculas recombinantes.
- La gran cantidad de virus liberados de una gran cantidad de hospedadores bacterianos permite detectar incluso raros eventos de recombinación.
Otra página —Bacteriophage Genetics— describe cómo se usa T2 para mapear el orden y el espaciado relativo de los genes en la molécula circular única de ADN que es el genoma del virus. Aquí veamos cómo se puede usar T4 para detectar mutaciones dentro de un solo gen y acelerar el proceso de mapeo de estas mutaciones puntuales mediante el uso de mutantes de deleción.
Detección de mutaciones dentro de un solo gen
En Bacteriophage Genetics, examinamos la mutación de un gen designado r, para “lisis rápida”. Resultó que en realidad hay tres loci génicos diferentes —rI, rII y rIII — mutaciones en cualquiera de los cuales produjeron un fenotipo de lisis rápida. Pero, además, se encontraron muchas mutaciones en cada una de estas. ¿Podría formarse el virus de tipo salvaje por recombinación entre mutaciones dentro del mismo gen? Seymour Benzer decidió averiguarlo.
Como vimos en Bacteriophage Genetics, la frecuencia de recombinación entre diferentes genes es baja (del orden de 10 -2). Uno esperaría que las frecuencias de recombinación entre mutaciones en un solo gen serían mucho menores (10 -4 o menos). Afortunadamente, Benzer podría explotar un fenómeno para permitirle detectar eventos tan raros: mutantes RII así como T4 de tipo silvestre pueden infectar y completar su ciclo de vida en una cepa de E. coli designada B. Sin embargo, mientras que los mutantes rII pueden infectar una cepa de E. coli designada K , no pueden completar su ciclo de vida en la cepa K. T4 de tipo silvestre can.
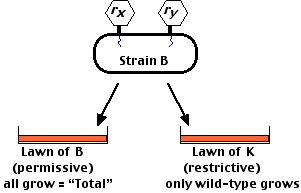
El procedimiento fue infectar la cepa B en cultivo líquido con dos mutantes a ensayar (designados aquí como r x y r y). Después de la incubación, estos se sembraron sobre un césped de:
- cepa B — que apoya el crecimiento de todos los virus dando así el número total de virus liberados.
- cepa K — en la que solo pueden crecer los virus de tipo silvestre.
La frecuencia de recombinación entre cualquier par de mutaciones se calcula como
\[\text{Recombination Frequency} = \dfrac{2 \times \text{number of wild-type plaques (strain K plaques)}}{\text{total number of plaques (on strain B)}}\]
Hay que duplicar el número que se encuentra en la cepa K porque solo se ve la mitad de los recombinantes; la otra mitad consiste en mutantes dobles. Usando esta técnica, Benzer finalmente encontró unas 2000 mutaciones diferentes en el gen rII. La frecuencia de recombinación entre algunos pares de estos fue tan baja como 0.02.
- El genoma T4 tiene 160,000 pares de bases de ADN que se extienden sobre ~1,600 centimorgans (cM).
- Así que 1 cM 100 pares de bases
- Entonces 0.02 cM representa un par de nucleótidos adyacentes.
- A partir de estos datos, Benzer concluyó que el
- unidad más pequeña de mutación y
- la unidad más pequeña de recombinación
En otras palabras, estas mutaciones representan un cambio en un solo par de bases —las llamamos mutaciones puntuales—. La recombinación entre dos moléculas de ADN puede ocurrir en cualquier par de nucleótidos.
Mapeo de mutaciones puntuales dentro de un gen
El orden relativo y espaciado de cualesquiera dos mutaciones puntuales en un solo gen como rII se puede hacer usando el procedimiento descrito en Bacteriophage Genetics. Pero con unas 2000 mutaciones diferentes para probar, el proceso llevaría muchísimo tiempo. (Incluso usando el procedimiento a describir ahora, Benzer pasó unos 10 años en el proyecto.) Benzer pudo acelerar el proceso de mapeo aprovechando el descubrimiento de que algunos de sus mutantes no tenían mutaciones puntuales sino deleciones. En contraste con las propiedades de los virus T4 con mutaciones puntuales, los virus T4 con deleciones en rII no mostraron recombinación con otros mutantes de RII o cualquier otro gen para el caso. Además, estas deleciones nunca volvieron a mutarse.
Mapeo de eliminación
Las deleciones pueden mapearse mediante el mismo procedimiento utilizado para las mutaciones puntuales. Simplemente cruce pares de mutantes de deleción y vea si producen progenie que puede crecer en la cepa K. de E. coli Aquí hay un ejemplo hipotético. Cada una de las 6 cepas de mutantes de deleción se cruzan con cada una de las otras.
| Cepas | 1 | 2 | 3 | 4 | 5 | 6 | |
| 1 | 0 | 0 | + | 0 | 0 | 0 | 1 y 3 no se superponen |
| 2 | 0 | + | + | 0 | 0 | debe cambiar 4 lejos de 2 | |
| 3 | 0 | + | + | 0 | 6 debe extenderse por debajo de 3 | ||
| 4 | 0 | + | + | extremo derecho de 4 debe ser removido de más de 6 | |||
| 5 | 0 | + | extremo izquierdo de 6 no debe superponerse 5 pero debe continuar solapándose 2. acortar el extremo derecho de 5 | ||||
| 6 | 0 |
A partir de los resultados, se puede dibujar un mapa que muestre el orden y el tamaño relativo de las eliminaciones.
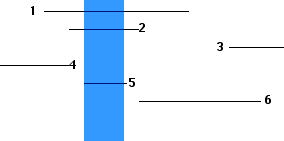
Con tal mapa de deleción, ahora se puede mapear rápidamente la ubicación de mutaciones puntuales al coinfectar cada una de las diferentes cepas de deleción (aquí 1—6) con la cepa mutante (“x”).
Ya no hay necesidad de contar las placas; simple ver si hay crecimiento o no.
| Coinfectar con cepa | 1 | 2 | 3 | 4 | 5 | 6 |
| y mutante “x” Resultados → | 0 | 0 | + | + | 0 | + |
A partir de estos resultados, se aprende que la mutación puntual “x” se localiza en el ADN de T4 dentro de la región mostrada anteriormente en azul.
Complementación
Como vimos anteriormente, se encontraron mutantes de lisis rápida (r) que mapearon a tres regiones diferentes del genoma T4: rI, rII y rIII. Esto significó que aquellos en diferentes regiones no eran alelos del mismo gen y más de un producto génico participó en la función de lisis. Incluso dentro de un “locus”, RII, resultó haber dos tramos diferentes de ADN, los cuales fueron necesarios intactos para la función de lisis. Esto fue revelado por la prueba de complementación que utilizó Benzer. En esta prueba,
- La cepa K de E. coli (que los mutantes rII pueden infectar pero no completar su ciclo de vida) —que crece en cultivo líquido— fue
- coinfectados con dos mutantes RII diferentes (mostrados en la figura como “1" y “2").
Obsérvese que este procedimiento difiere del anterior (recombinación) en que se utiliza la no permisiva E. coli K para la infección inicial (no la cepa B como antes). Ni la cepa rII “1" ni la cepa rII “2" son capaces de crecer en E. coli K. Pero si la función perdida en rII “1" NO es la misma que la función perdida en rII “2", entonces
- cada uno debe ser capaz de producir el producto génico que falta en el otro —complementación — y
- se producirán fagos vivos. (Nuevamente, no hay necesidad de contar las placas; simplemente vea si están formadas o no).
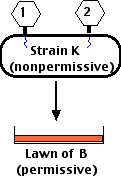
| Cepas mutantes | 1 | 2 | 3 | 4 | 5 |
| 1 | 0 | 0 | + | 0 | + |
| 2 | 0 | + | 0 | + | |
| 3 | 0 | + | 0 | ||
| 4 | 0 | + | |||
| 5 | 0 |
A partir de estos resultados, se puede deducir que estos 5 mutantes de RII se encuentran en dos grupos de complementación diferentes, que Benzer designó A (que contenía las cepas 1, 2 y 4) y B (que contenía las cepas 3 y 5). Trabajos posteriores mostraron que la función de RII dependía de los productos polipeptídicos codificados por dos regiones adyacentes (A y B) de RII (quizás actuando como heterodímero). En términos de función, entonces, tanto A como B califican como genes independientes. En coinfecciones por dos cepas mutantes,
- Si A o B está mutado en la misma molécula de ADN (” cis “), no hay función mientras
- si A está mutado en una molécula de ADN y B en la otra (“trans”), se restablece la función.
La complementación, entonces, es la capacidad de dos mutaciones diferentes para restaurar la función de tipo silvestre cuando están en el “trans” (en diferentes moléculas de ADN), pero no cuando están en “cis” (en la misma molécula de ADN). Benzer acuñó el término cistrón para estas unidades genéticas de función. Pero hoy, simplemente modificamos conceptos anteriores del “gen” para que se ajusten a esta definición operativa.


