8.7: Mapeo de los genes de T2
- Page ID
- 57247
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
T2 y su pariente cercano T4 son virus que infectan a la bacteria E. coli. La infección termina con la destrucción (lisis) de la célula bacteriana por lo que estos virus son ejemplos de bacteriófagos (“comedores de bacterias”). Cada partícula de virus (virión) consiste en:
- una cabeza de proteína (~0.1 µm) dentro de la cual hay una molécula circular única de ADN bicatenario que contiene 166,000 pares de bases.
- una cola de proteína de la que se extienden
- fibras finas de proteína
Ciclo de Vida
El virus se adhiere a la célula de E. coli (a). Esto requiere una interacción molecular precisa entre las fibras y la pared celular del huésped.
- La molécula de ADN se inyecta en la célula (b).
- En el plazo de 1 minuto, el ADN viral comienza a transcribirse y traducirse en algunas de las proteínas virales, y se detiene la síntesis de las proteínas del huésped.
- A los 5 minutos se producen enzimas virales necesarias para la síntesis de nuevas moléculas de ADN viral (c).
- A los 8 minutos, se sintetizan unas 40 proteínas estructurales diferentes para la cabeza y la cola virales.
- A los 13 minutos, comienza el ensamblaje de nuevas partículas virales (d).
- A los 25 minutos, la lisozima viral destruye la pared celular bacteriana y los virus estallan, listos para infectar nuevos huéspedes (e).
- Si las células bacterianas están creciendo en cultivo líquido, queda claro.
- Si las células bacterianas están creciendo en un “césped” en la superficie de una placa de agar, entonces aparecen agujeros, llamados placas, en el césped.
Ocasionalmente, aparecen nuevos fenotipos como un cambio en la apariencia de las placas o incluso una pérdida en la capacidad de infectar al huésped.
Ejemplos:
- h
- Algunas cepas de E. coli, por ejemplo una designada B/2, obtienen la capacidad de resistir la infección por T2 normal (“tipo silvestre”). La mutación ha provocado un cambio en la estructura de su pared celular para que las fibras de la cola de T2 ya no puedan unirse a ella. Sin embargo, T2 puede devolver el golpe. Aparecen mutantes T2 ocasionales que superan esta resistencia. El gen mutado, designado h (para “rango de hospedadores”), codifica un cambio en las fibras de la cola para que puedan unirse una vez más a la pared celular de la cepa B/2. El gen normal o “de tipo silvestre” se denomina h +.
- Cuando se coloca en placas sobre un césped que contiene tanto E. coli B como E. coli B/2,
- los virus mutantes (h) pueden lisar ambas cepas de E. coli, produciendo placas claras, mientras que
- los virus de tipo silvestre (h +) solo pueden lisar E. coli B produciendo placas moteadas o turbias.
- r
- Aparecen mutantes T2 ocasionales que salen de su célula hospedadora antes de lo normal.
- La mutación ocurre en un gen designado r (para “lisis rápida”). Se revela por las placas extragrandes que forma.
- El gen de tipo silvestre, que produce un tiempo normal de lisis, se denomina r +. Forma placas de tamaño normal.
Al igual que con tantos organismos, la ocurrencia de mutaciones proporciona las herramientas para aprender sobre cosas como
- la función del gen
- su ubicación en la molécula de ADN (mapeo)
Mapeo por frecuencias de recombinación
Como hemos visto, la cepa B de E. coli puede ser infectada tanto por cepas h + como h de T2. De hecho, una sola célula bacteriana puede ser infectada simultáneamente por ambas. Infectemos un cultivo líquido de E. coli B con dos virus T2 mutantes diferentes: h r + y h + r. cuando esto se hace en cultivo líquido, y luego sembrado en un césped mixto de E. coli B y B/2, aparecen cuatro tipos diferentes de placas.

| Genotipo | Fenotipo | Número de placas |
|---|---|---|
| hr + | transparente, pequeño | 460 |
| h + r | turbio, grande | 460 |
| h + r + | turbio, pequeño | 40 |
| hr | transparente, grande | 40 |
| Total = | 1000 |
Los más abundantes (460 cada uno) son los que representan los tipos parentales; es decir, los fenotipos son los esperados de las dos cepas infectantes. Sin embargo, aparecen pequeños números (40 cada uno) de dos nuevos fenotipos. Estos pueden explicarse por la recombinación genética que ha ocurrido ocasionalmente entre el ADN de cada tipo parental dentro de la célula bacteriana.

Al igual que en los organismos superiores, se supone que la frecuencia de los recombinantes es proporcional a la distancia entre los loci génicos. En este caso, 80 de cada 1000 placas fueron recombinantes, por lo que a la distancia entre los loci h y r se le asigna un valor de 8 unidades cartográficas o centimórganos (cM). Ahora coinfectar E. coli B con otras dos cepas de T2:
- hm + y
- h + m
| hm + | 470 |
| h + m | 470 |
| h + m + | 30 |
| hm | 30 |
| Total = | 1000 |
Nuevamente, se producen 4 tipos de placas: parentales (470 cada una) y recombinantes (30 cada una).
El menor número de recombinantes indica que estos dos loci génicos (h y m) están más cerca (6 cM) que h y r (8 cM). Pero el orden de los tres loci podría ser
- m —6— h —8— r
o - h —6— m -2- r
Para averiguar cuál es el orden correcto, realice un tercer apareamiento utilizando
- mr + y
- m + r

| mr + | 440 |
| m + r | 440 |
| m + r + | 60 |
| mr | 60 |
| Total = | 1000 |
Esto deja claro que el orden es m—h—r, no h—m—r.
Pero ¿por qué solo 12 cM entre los loci exteriores (m y r) en lugar de los 14 cM producidos al agregar las distancias del mapa que se encuentran en los dos primeros apareamientos?
Una cruz de tres puntos
La respuesta proviene de realizar un apareamiento entre virus T2 que difieren en los tres loci:
- hmr
y - h + m + r +
(Nota: esta vez uno de los padres tiene todos los mutantes; el otro todos los alelos de tipo salvaje — ¡no se confunda!)
| Grupo 1 | hmr | 435 |
| Grupo 2 | h + m + r + | 435 |
| Grupo 3 | h + mr + | 25 |
| Grupo 4 | hm + r | 25 |
| Grupo 5 | hmr + | 35 |
| Grupo 6 | h + m + r | 35 |
| Grupo 7 | hm + r + | 5 |
| Grupo 8 | h + mr | 5 |
| Total = | 1000 |
El resultado: se forman 8 tipos diferentes de placas.
- padres; es decir, no recombinantes en los Grupos 1 y 2;
- recombinantes — todos los demás
El análisis de estos datos muestra cómo el cruce de dos puntos entre m y r subestimó la verdadera distancia entre ellos.
Veamos primero los pares individuales de recombinantes como lo hicimos antes (ignorando así el tercer locus).
- Si miramos todos los recombinantes entre h y r pero ignoramos m (como en el primer experimento), encontramos que están contenidos en los Grupos 5, 6, 7 y 8 —dando el total de 80 que encontramos originalmente.
- Si miramos recombinantes entre h y m pero ignoramos r (como en el segundo experimento), encontramos que están contenidos en los Grupos 3, 4,7 y 8 —dando el mismo total de 60 que encontramos antes.
- Pero si nos enfocamos únicamente en m y r (como hicimos en el tercer experimento), encontramos que los recombinantes están contenidos en los Grupos 3, 4, 5 y 6 —dando el mismo total de 120 que antes mientras que los no recombinantes no están solo en los Grupos 1 y 2 sino también en los Grupos 7 y 8. El motivo: se produjo un doble cruce en estos casos, restaurando la configuración parental de los alelos m y r.
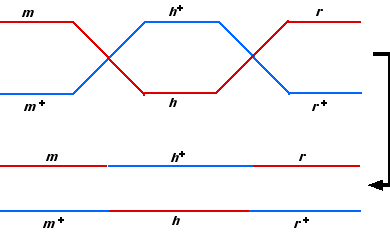
- Debido a que estos cruces dobles estaban ocultos en el tercer experimento, la distancia del mapa (12 cM) fue subestimada. Para obtener la verdadera distancia del mapa, agregamos su número a cada uno de los otros grupos recombinantes (Grupos 3,4,5, y 6) de manera que 25 + 5 +25 +5 +35 + 5 + 35 + 5 = 140, y la distancia verdadera del mapa entre m y r es el 14 cM que encontramos al agregar las distancias del mapa entre h y r (8 cM) y h y m (6 cM).
El cruce de tres puntos también es útil porque da el orden génico simplemente por inspección:
- Encuentra los genotipos más raros (aquí Grupos 7 y 8)
- El gen NO en la configuración parental (aquí h) es siempre el medio.


