4.2: Control de la Actividad Enzimática
- Page ID
- 52901
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Una versión imprimible de esta sección está aquí: BiochemFFA_4_2.pdf. Todo el libro de texto está disponible de forma gratuita de los autores en http://biochem.science.oregonstate.edu/content/biochemistry-free-and-easy
Regulación de la actividad enzimática
Aparte de su capacidad para acelerar en gran medida las tasas de reacciones químicas en las células, las enzimas tienen otra propiedad que las hace valiosas. Esta propiedad es que su actividad puede ser regulada, permitiendo que sean activadas e inactivadas, según sea necesario. Esto es tremendamente importante para mantener la homeostasis, permitiendo que las células respondan de manera controlada a los cambios en las condiciones tanto internas como externas.
La inhibición de enzimas específicas por fármacos también puede ser médicamente útil. Comprender los mecanismos que controlan la actividad enzimática es, por lo tanto, de considerable importancia.
Inhibición
Primero discutiremos cuatro tipos de inhibición enzimática: inhibición competitiva, no competitiva, no competitiva y suicida. De estos, los tres primeros tipos son reversibles. El último, la inhibición del suicidio, no lo es.
Inhibición competitiva
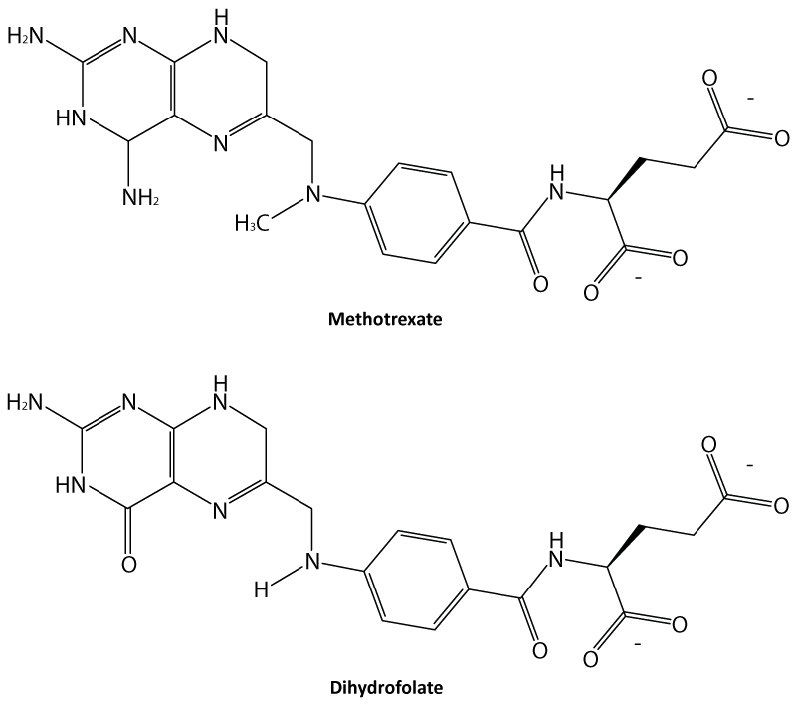
Probablemente el tipo de inhibición enzimática más fácil de entender es la inhibición competitiva y es la que más comúnmente se explota desde el punto de vista farmacéutico. Las moléculas que son inhibidores competitivos de enzimas se asemejan a uno de los sustratos normales de una enzima. Un ejemplo es el metotrexato, que se asemeja al sustrato de folato de la enzima dihidrofolato reductasa (DHFR). Esta enzima normalmente cataliza la reducción del folato, una reacción importante en el metabolismo de los nucleótidos.
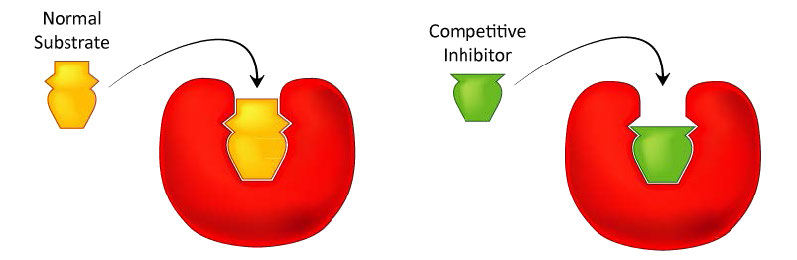
Figura 4.33 - Los inhibidores competitivos se asemejan al sustrato normal y compiten por la unión en el sitio activo. Imagen de Aleia Kim
Inhibidor de unión
Cuando el medicamento metotrexato está presente, parte de la enzima DHFR se une a él, en lugar de al folato, y durante el tiempo que el metotrexato se une, la enzima está inactiva e incapaz de unirse al folato. Así, se inhibe la enzima. Notablemente, el sitio de unión en DHFR para metotrexato es el sitio activo, el mismo lugar al que normalmente se uniría el folato. Como resultado, el metotrexato 'compite' con el folato para unirse a la enzima. Cuanto más metotrexato haya, más efectivamente compite con el folato por el sitio activo de la enzima. Por el contrario, cuanto más folato hay, menor efecto tiene el metotrexato sobre la enzima porque el folato lo supera.
Sin efecto sobre Vmax
¿Cómo estudiamos la inhibición competitiva? Por lo general, se realiza de la siguiente manera. Primero, se realiza un conjunto de reacciones V0 vs. [S] sin inhibidor (aproximadamente 20 tubos, con tampón y cantidades constantes de enzima, cantidades variables de sustrato, tiempos de reacción iguales). Se grafica V0 vs. [S] (Figura 4.35 línea roja), así como 1/V0 vs. 1/ [S] (Figura 4.36 línea verde). A continuación, se realiza un segundo conjunto de reacciones de la misma manera que antes, excepto que se agrega una cantidad fija del inhibidor de metotrexato a cada tubo. A bajas concentraciones de sustrato, el metotrexato compite por la enzima de manera efectiva, pero a altas concentraciones de sustrato, el inhibidor tendrá un efecto muy reducido, ya que el sustrato lo supera, debido a su mayor concentración (recuerde que el inhibidor está a concentración fija).
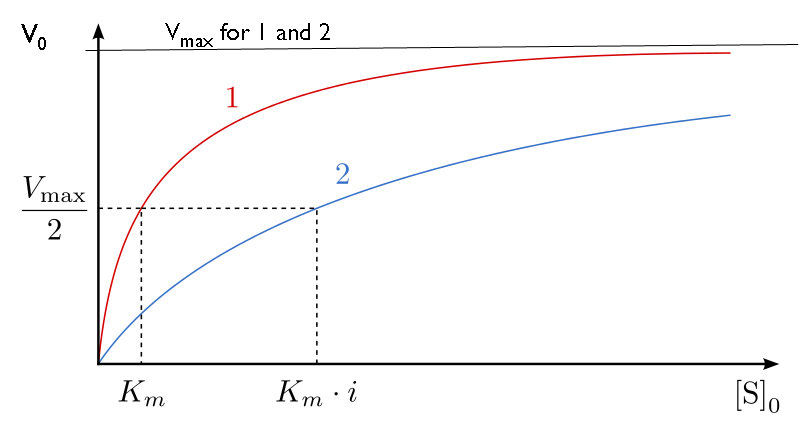
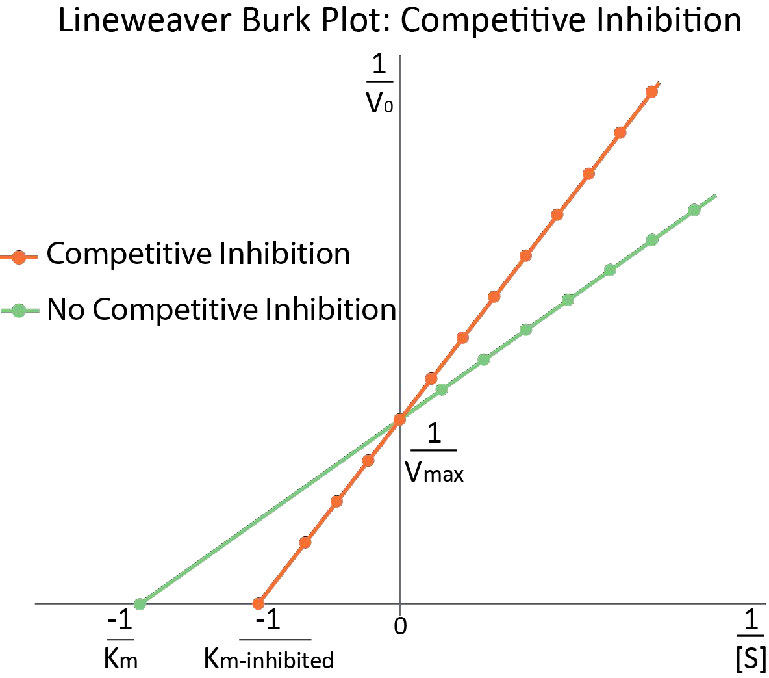
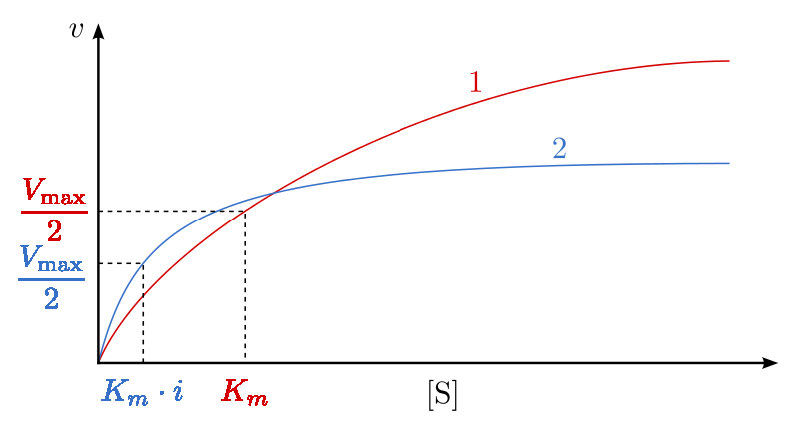
Gráficamente, los resultados de estos experimentos de inhibidores se muestran en la Figura 4.35 (línea azul) y la Figura 4.36 (línea naranja). Observe que a altas concentraciones de sustrato, el inhibidor competitivo esencialmente no tiene ningún efecto, provocando que el\(V_{max}\) para la enzima permanezca sin cambios. Para reiterar, esto se debe a que a altas concentraciones de sustrato, el inhibidor no compite bien. Sin embargo, a concentraciones de sustrato más bajas, lo hace.
Incrementado \(K_m\)
En reacciones inhibidas competitivamente, la km aparente de la enzima para el sustrato aumenta (\(-1/K_m\)se acerca a la línea cero - roja en la Figura 4.36) cuando el inhibidor está presente en comparación con cuando el inhibidor está ausente, ilustrando así la mejor competencia del inhibidor en sustrato inferior concentraciones. Puede que no sea obvio por qué llamamos al Km cambiado el Km aparente de la enzima. La razón es que el inhibidor en realidad no cambia la afinidad de la enzima por el sustrato de folato. Sólo parece hacerlo. Esto se debe a la forma en que funciona la inhibición competitiva. Cuando el inhibidor competitivo se une a la enzima, efectivamente se 'saca de acción'. Las enzimas inactivas no tienen afinidad por el sustrato y tampoco tienen actividad. No podemos medir Km para una enzima inactiva.
Las moléculas enzimáticas que no están unidas por metotrexato pueden, de hecho, unirse al folato y son activas. El metotrexato no tiene ningún efecto sobre ellos y sus valores de Km se mantienen inalterados. ¿Por qué entonces, el Km aparece más alto en presencia de un inhibidor competitivo? La razón es que el inhibidor competitivo está teniendo un mayor efecto de reducir la cantidad de enzima activa a concentraciones menores de sustrato que a concentraciones más altas de sustrato. Cuando se reduce la cantidad de enzima, se debe tener más sustrato para suministrar la cantidad reducida de enzima suficientemente para llegar a Vmax/2.
Cabe señalar que en la inhibición competitiva, el porcentaje de enzimas inactivas cambia drásticamente en el rango de valores [S] utilizados. Para comenzar, a valores bajos de [S], se inhibe el mayor porcentaje de la enzima. A alta [S], no se inhibe ningún porcentaje significativo de enzima. Esto no siempre es así, como veremos en la inhibición no competitiva.
Inhibición no competitiva
Un segundo tipo de inhibición emplea inhibidores que no se asemejan al sustrato y no se unen al sitio activo, sino a un sitio separado en la enzima (Figura 4.37). El efecto de unirse a un inhibidor no competitivo es significativamente diferente de unirse a un inhibidor competitivo porque no hay competencia. En el caso de la inhibición competitiva, el efecto del inhibidor podría ser reducido y eventualmente abrumado con cantidades crecientes de sustrato. Esto se debió a que aumentar el sustrato hizo que los porcentajes crecientes de la enzima fueran activos. Con inhibición no competitiva, aumentar la cantidad de sustrato no tiene efecto sobre el porcentaje de enzima que está activa. De hecho, en la inhibición no competitiva, el porcentaje de enzima inhibida sigue siendo el mismo en todos los rangos de [S].
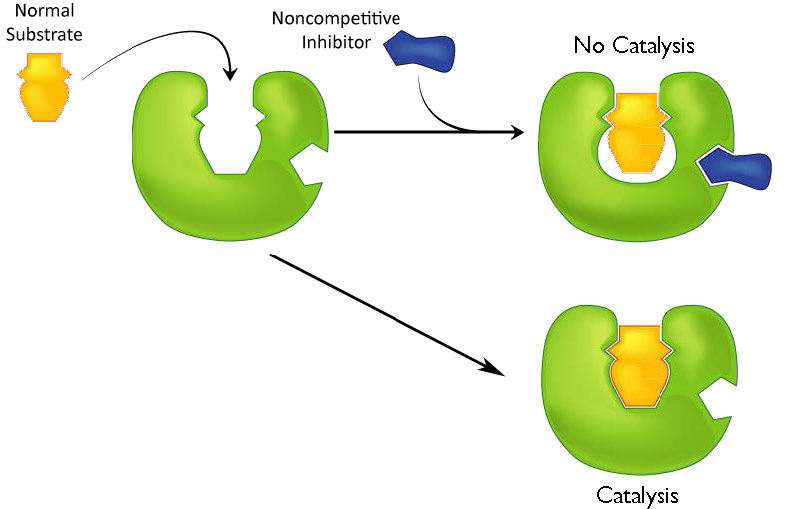
Esto significa, entonces, que la inhibición no competitiva reduce efectivamente la cantidad de enzima en la misma cantidad fija en un experimento típico a cada concentración de sustrato utilizada El efecto de esta inhibición se muestra en la Figura 4.38 y 4.39. Como puede ver,\(V_{max}\) se reduce en la inhibición no competitiva en comparación con las reacciones desinhibidas.
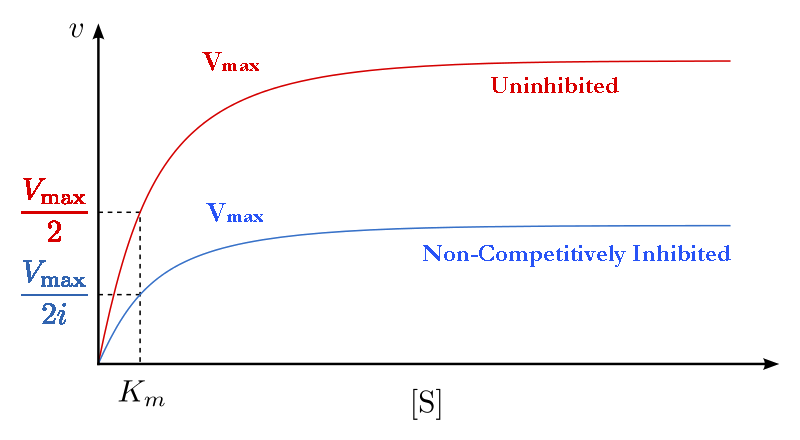
Esto tiene sentido si recordamos que Vmax depende de la cantidad de enzima presente. Reducir la cantidad de enzima presente reduce\(V_{max}\). En la inhibición competitiva, esto no ocurre de manera detectable, ya que a altas concentraciones de sustrato, existe esencialmente el 100% de la enzima activa y la\(V_{max}\) parece no cambiar. Adicionalmente, Km para reacciones inhibidas no competitivamente no cambia de las reacciones desinhibidas. Esto se debe a que, como se señaló anteriormente, solo se puede medir la\(K_m\) de enzimas activas y\(K_m\) es una constante para una enzima dada.
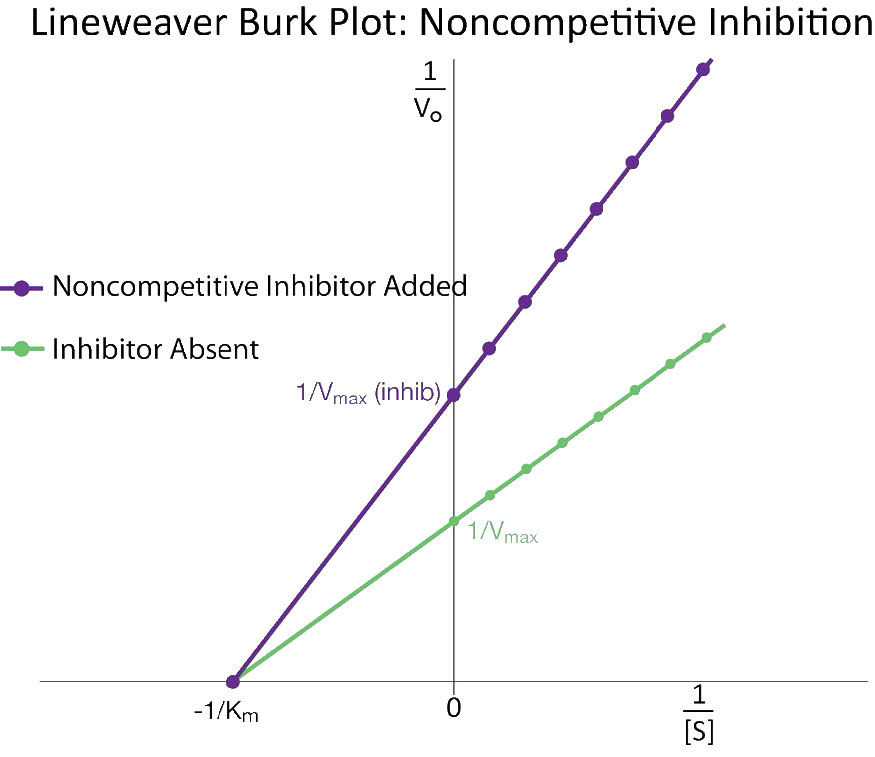
Inhibición no competitiva
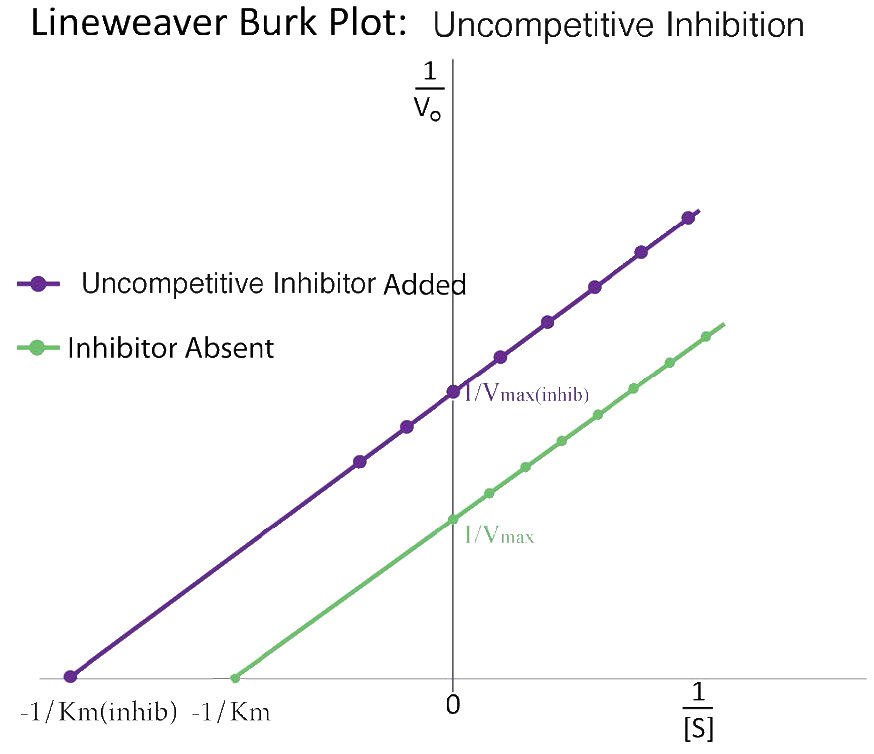
Un tercer tipo de inhibición enzimática es la inhibición no competitiva, que tiene la propiedad impar de un Vmax reducido así como de un Km reducido. La explicación de estos resultados aparentemente impares se basa en el hecho de que el inhibidor no competitivo se une solo al complejo enzima-sustrato (ES) (Figura 4.40). El complejo unido al inhibidor se forma principalmente bajo concentraciones de sustrato alto y el complejo ES-I no puede liberar producto mientras el inhibidor está unido, explicando así la reducción\(V_{max}\).
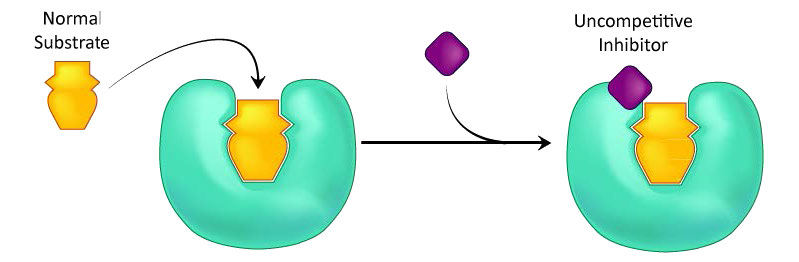
El Km reducido es un poco más difícil de conceptualizar. La razón es que el complejo unido a inhibidor reduce efectivamente la concentración del complejo ES. Por el Principio de Le Chatelier, se produce un cambio para formar complejo ES adicional, resultando en menos enzima libre y más enzima en las formas ES y ESI (ES con inhibidor). Las disminuciones en la enzima libre corresponden a una enzima con mayor afinidad por su sustrato. Así, paradójicamente, la inhibición no competitiva disminuye\(V_{max}\) e incrementa la afinidad de una enzima por su sustrato (\(K_m\)- Figuras 4.41 y 4.42).
Inhibición del suicidio
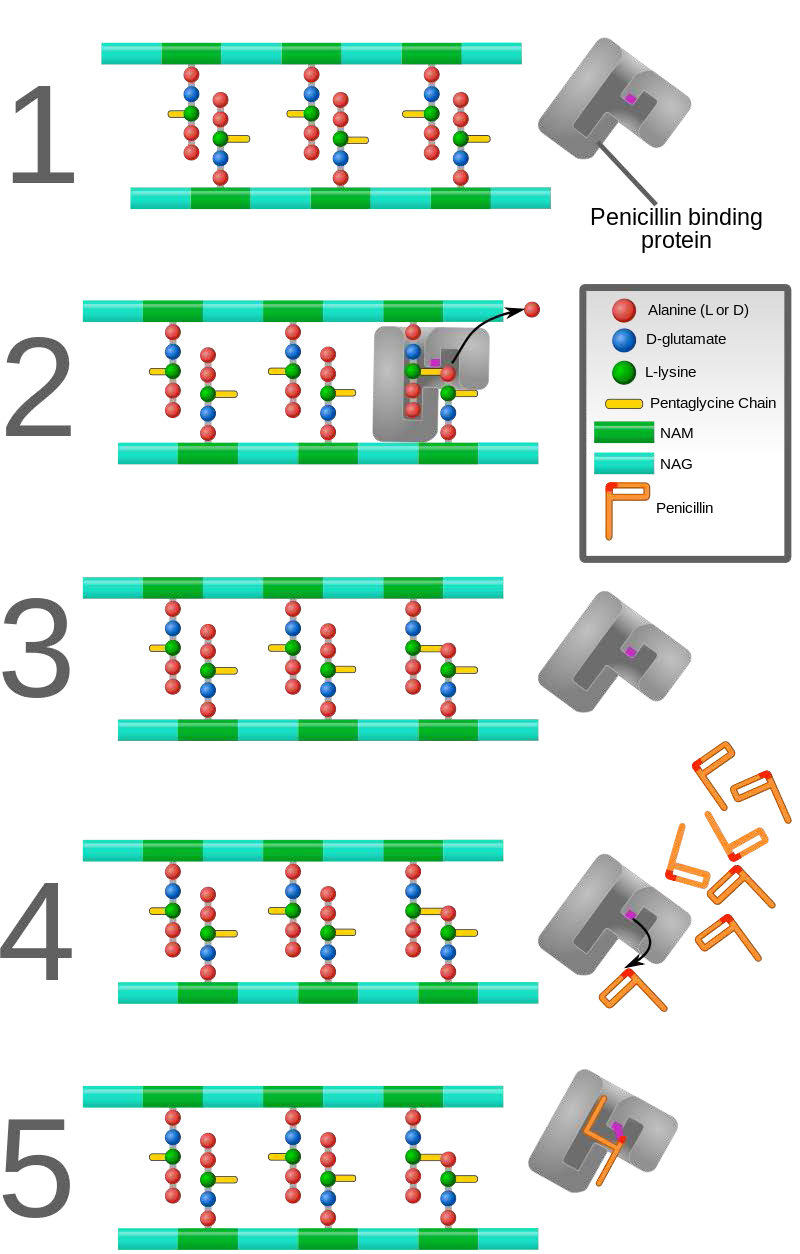
En contraste con los tres primeros tipos de inhibición, que implican la unión reversible del inhibidor a la enzima, la inhibición suicida es irreversible, debido a que el inhibidor se une covalentemente a la enzima durante la inhibición. La inhibición suicida se asemeja bastante a la inhibición competitiva porque el inhibidor generalmente se parece al sustrato y se une al sitio activo de la enzima. La principal diferencia es que el inhibidor suicida es químicamente reactivo en el sitio activo y hace un enlace con él que impide su eliminación. Tal mecanismo es el empleado por la penicilina (Figura 4.43), que se une covalentemente a la enzima bacteriana, transpeptidasa DD e impide que funcione. Dado que la función normal de la enzima es hacer un enlace necesario para el complejo peptidoglicano de la pared celular bacteriana, la pared celular no puede formarse adecuadamente y las bacterias no pueden reproducirse.
Control de enzimas
Es apropiado platicar en este punto sobre los mecanismos que las células utilizan para controlar las enzimas. Hay cuatro métodos generales que se emplean:
- alosterismo,
- modificación covalente,
- acceso al sustrato, y
- control de la síntesis/descomposición de enzimas.
Algunas enzimas están controladas por más de uno de estos métodos.
Alosterismo
El término alosterismo se refiere al hecho de que la actividad de ciertas enzimas puede verse afectada por la unión de moléculas pequeñas. Las moléculas que causan efectos alostéricos vienen en dos clasificaciones. Los que son sustratos para las enzimas que afectan se denominan efectores homotrópicos y los que no son sustratos se denominan efectores heterotrópicos.
Los efectores homotrópicos suelen ser activadores de las enzimas a las que se unen y los resultados de su acción pueden verse en la conversión de la curva hiperbólica típica de una gráfica V0 vs. [S] para una enzima (Figura 4.18), convirtiéndose en una gráfica sigmoidal (Figura 4.44). Esto se debe a la conversión de la enzima del estado T al estado R al unirse al sustrato/efector homotrópico.
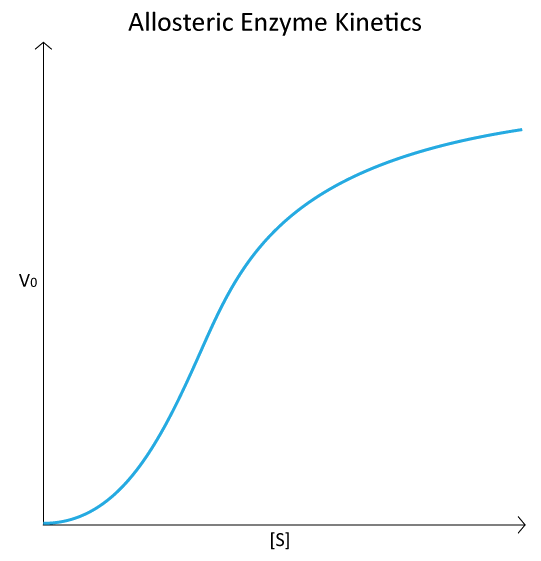
La gráfica V0 vs. [S] de las reacciones enzimáticas alostéricas se asemeja a la curva de unión al oxígeno de la hemoglobina (ver Figura 2.83). A pesar de que la hemoglobina no es una enzima y, por lo tanto, no está catalizando una reacción, la similitud de las parcelas no es casual. En ambos casos, se está midiendo la unión de una molécula externa —directamente, en la gráfica de hemoglobina, e indirectamente por la gráfica V0 vs. [S], ya que la unión al sustrato es un factor en la velocidad de reacción enzimática.
Inhibición alostérica
Alostéricamente, la regulación de estas enzimas funciona induciendo diferentes estados físicos (formas, por así decirlo) que afectan su capacidad de unirse al sustrato. Cuando una enzima se inhibe al unirse a un efector, se convierte al estado T (T=apretado), tiene una afinidad reducida por el sustrato y es a través de este medio que la reacción se ralentiza.
Activación alostérica
Por otro lado, cuando una enzima se activa por unión efectora, se convierte al estado R (R=relajado) y se une al sustrato mucho más fácilmente. Cuando no hay ningún efector presente, la enzima puede estar en una mezcla de estados T y R.
Inhibición de retroalimentación
Un interesante tipo de control alostérico es exhibido por la HMG-CoA reductasa, que cataliza una reacción importante en la vía que conduce a la síntesis de colesterol. La unión del colesterol a la enzima reduce significativamente la actividad de la enzima. El colesterol no es un sustrato para la enzima, por lo que es por lo tanto un efector heterotrópico.
Notablemente, sin embargo, el colesterol es el producto final de la ruta en la que la HMG-CoA reductasa cataliza una reacción. Cuando las enzimas son inhibidas por un producto final de la vía en la que participan, se dice que exhiben inhibición por retroalimentación.
La inhibición de retroalimentación siempre opera por alosterismo y además, proporciona un control importante y eficiente de una vía completa. Al inhibir una enzima temprana en una vía, se detiene o reduce el flujo de materiales (y la hidrólisis de ATP requerida para su procesamiento) para toda la ruta, asumiendo que no hay métodos de suministro alternativos.
Control de vías
En la vía de biosíntesis del colesterol, detener esta enzima tiene el efecto de cerrar (o al menos ralentizar) toda la vía. Esto es significativo porque después de la catálisis por HMG-CoA reductasa, hay más de 20 reacciones adicionales necesarias para producir colesterol, muchas de ellas requiriendo energía ATP. El cierre de una reacción detiene a todas ellas. Otro excelente ejemplo de control alostérico e inhibición por retroalimentación es la enzima ATCasa, que se analiza a continuación.
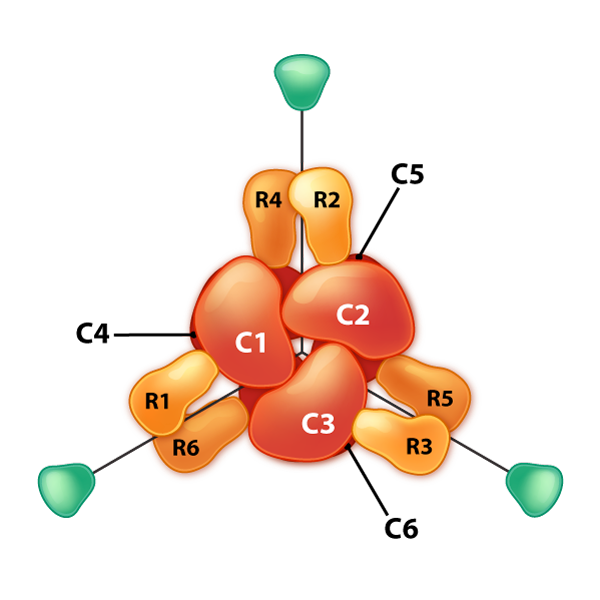
ATCase
Otro ejemplo interesante de control alostérico e inhibición de retroalimentación se asocia con la enzima Aspartato Transcarbamilasa (ATCasa). Esta enzima, que cataliza una etapa en la síntesis de nucleótidos de pirimidina, tiene 12 subunidades. Estas incluyen seis subunidades catalíticas idénticas y seis subunidades reguladoras idénticas. Las subunidades catalíticas se unen al sustrato y catalizan una reacción. Las subunidades reguladoras se unen a ATP o CTP. Si se unen a ATP, las subunidades enzimáticas se disponen en el estado R.
R-estado
El estado R de la ATCasa permite que el sustrato tenga un acceso más fácil a los seis sitios activos y la reacción ocurre más rápidamente. Para la misma cantidad de sustrato, una enzima en el estado R tendrá una velocidad mayor que la misma enzima que no está en el estado R. Por el contrario, si la enzima se une a CTP en una de sus subunidades reguladoras, las subunidades se dispondrán en el estado T y en esta forma, el sustrato no tendrá fácil acceso a los sitios activos, resultando en una velocidad más lenta para la misma concentración de sustrato en comparación con el estado R. ATCasa es interesante porque también puede voltear al estado R cuando uno de los sustratos (aspartato) se une a un sitio activo dentro de una de las subunidades catalíticas.
El aspartato tiene el efecto de activar la acción catalítica de la enzima favoreciendo el estado R. Así, el aspartato, que es un sustrato de la enzima es un efector homotrópico y ATP y CTP, que no son sustratos de la enzima son efectores heterotrópicos de ATCasa.
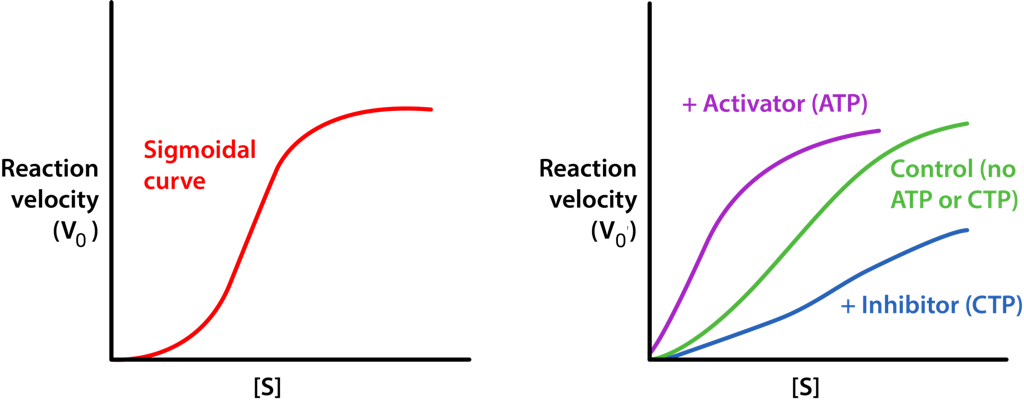
Modelos alostéricos
Hay tres modelos comúnmente utilizados para explicar cómo el alosterismo regula la actividad enzimática de múltiples subunidades. Se les conoce como
- el modelo Monod-Wyman-Changeux (MWC) (también conocido como el modelo concertado),
- el modelo secuencial (también conocido como KNF),
- y el modelo morpheein.
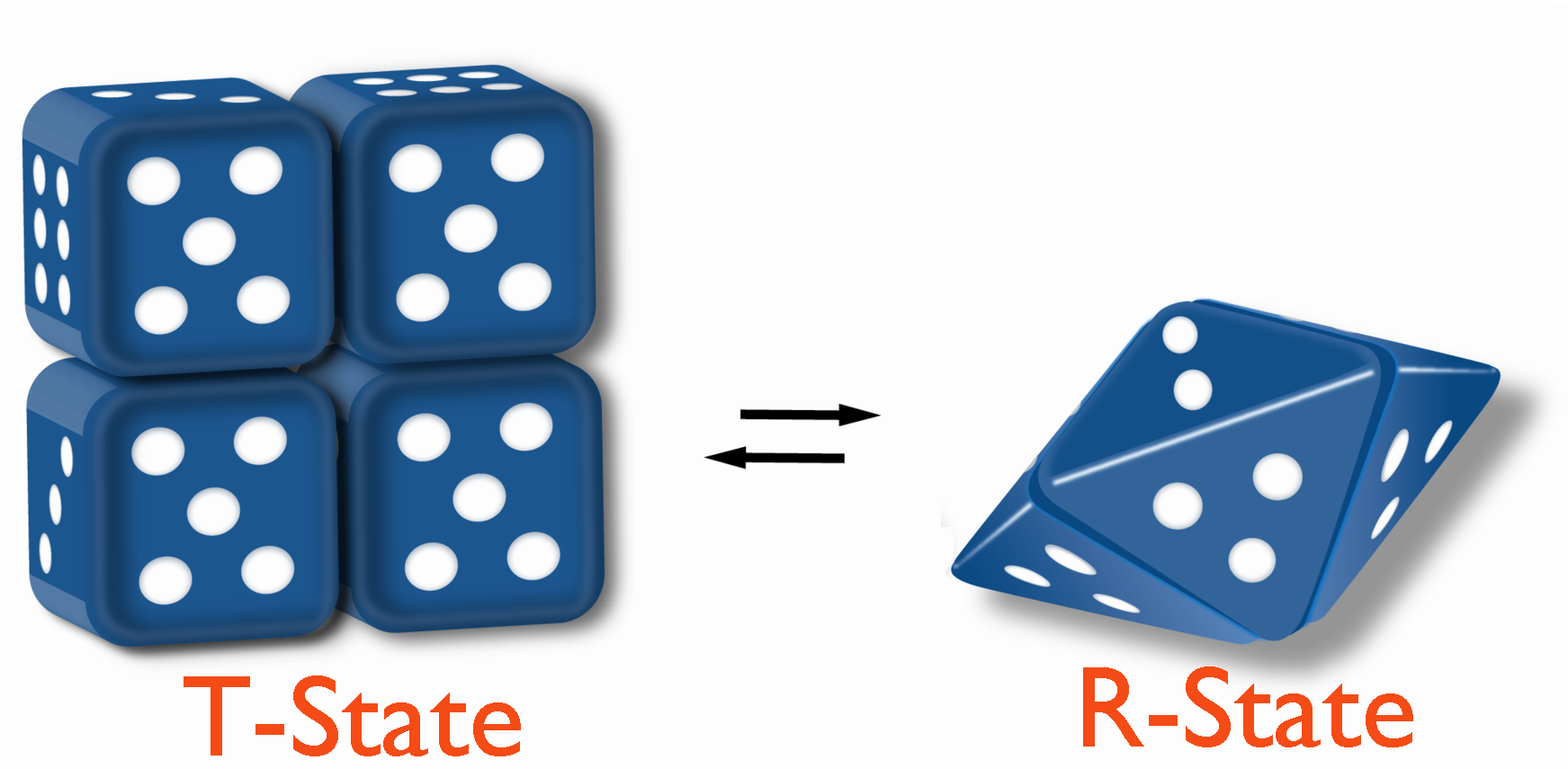
Todos los modelos describen un estado Tenso (T) que es menos activo catalíticamente y un estado Relajado (R) que es más activo catalíticamente. Los modelos difieren en cómo cambian los estados.
Modelo secuencial
En el modelo secuencial, la unión de un efector alostérico por una subunidad hace que cambie del estado T al R (o viceversa) y ese cambio facilita que las subunidades adyacentes cambien de manera similar el estado. Con este modelo, existe una relación causa/efecto entre la unión de un efector por una subunidad y el cambio de estado por una subunidad adyacente.
En la hemoglobina, por ejemplo, la unión de un oxígeno por una unidad del complejo puede inducir a esa unidad a voltear al estado R y, a través de interacciones con otras subunidades, hacer que favorezcan la adopción de la configuración R antes de que se unan al oxígeno. De esta manera, la unión de una subunidad favorece la unión de otras y la cooperatividad puede explicarse por el cambio en la afinidad de unión a medida que cambia la concentración de oxígeno.
Modelo MWC
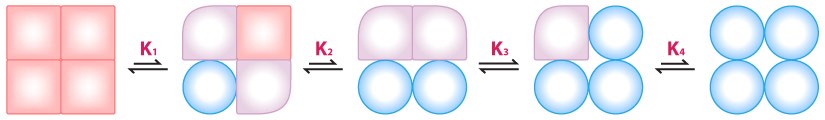
El modelo MWC es menos intuitivo. En él, todo el complejo cambia de estado de T a R (o viceversa) independientemente de la unión de efectores. Se postula que el volteo entre los estados T y los estados R está en equilibrio de estados en ausencia de efector (por ejemplo, una relación 50 a 1 de T/R, a esta relación se le conoce como L, entonces L = T/R). La unión del efector por el complejo enzimático tiene la tendencia de “bloquear” el complejo en un estado. La unión de inhibidores aumentará la relación de T/R mientras que la unión de activadores incrementará R y así disminuirá la ración de T/R.
Modelo Morpheein
El modelo de morfeeína es similar al modelo MWC, pero con un paso agregado de disociación de las subunidades. El modelo MWC propone que el cambio entre los estados R y T ocurre por el complejo como un todo y ocurre en todas las unidades simultáneamente. En cambio, el modelo de morfeeína propone que la enzima multisubunidad se descompone en unidades individuales que luego pueden voltear en estructura y volver a formar el complejo. En el modelo morpheein, solo las unidades de forma idéntica (todas las R, por ejemplo) pueden unirse en el complejo, explicando así el estado “todo-R-” o “todo-T-” que se encuentra en el modelo MWC.
Un gran número de enzimas, incluyendo prominentes como citrato sintasa, acetil-CoA carboxilasa, glutamato deshidrogenasa, ribonucleótido reductasa y lactato deshidrogenasa tienen un comportamiento consistente con el modelo de morfeeína.

Control covalente de enzimas
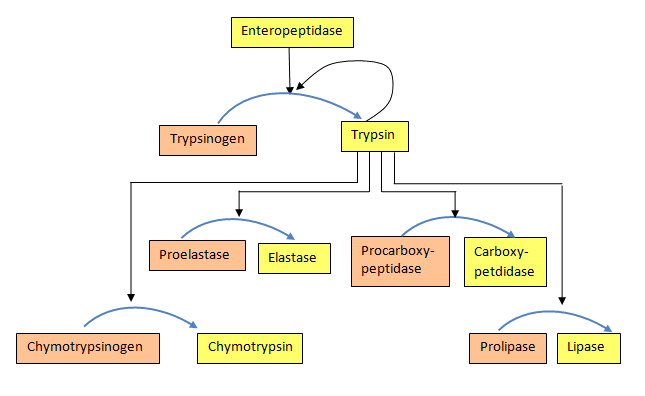
Algunas enzimas se sintetizan en una forma completamente inactiva y su activación requiere de enlaces covalentes en ellas para ser escindidos. Tales formas inactivas de enzimas se llaman zimógenos. Los ejemplos incluyen las proteínas involucradas en la coagulación de la sangre y las enzimas proteolíticas del sistema digestivo, como la tripsina, la quimotripsina, la pepsina y otras.
Sintetizar algunas enzimas en una forma inactiva tiene muy buen sentido cuando la actividad de una enzima puede ser dañina para el tejido donde se está fabricando. Por ejemplo, el padecimiento doloroso conocido como pancreatitis surge cuando las enzimas digestivas elaboradas en el páncreas se activan demasiado pronto y terminan atacando el páncreas.
Cascadas
Tanto para las enzimas coagulantes de la sangre como para las enzimas digestivas, los zimógenos se activan en una cascada de proteasas. Esto ocurre cuando la activación de una enzima activa a otras en una especie de reacción en cadena. En tal esquema la primera enzima activada escinde proteolíticamente el segundo zimógeno, provocando que se active, lo que a su vez activa un tercero y esto puede proceder a través de varios niveles de acción enzimática (Figura 4.50).
La ventaja de las cascadas es que permiten que una gran cantidad de zimógenos se active con bastante rapidez, ya que existe una amplificación de la señal en cada nivel de catálisis.
Los zimógenos también abundan en la sangre. La coagulación de la sangre implica la polimerización de una proteína conocida como fibrina. Dado que la formación aleatoria de fibrina es extremadamente peligrosa porque puede bloquear el flujo de sangre, provocando potencialmente un ataque cardíaco/accidente cerebrovascular, el cuerpo sintetiza la fibrina como zimógeno (fibrinógeno) y su activación resulta de una “cascada” de activaciones de proteasas que surgen cuando se recibe una señal de una herida. De igual manera, la enzima que cataliza la eliminación de coágulos de fibrina (plasmina) también se sintetiza como un zimógeno (plasminógeno), ya que la eliminación aleatoria de coágulos también sería peligrosa (ver más adelante también).
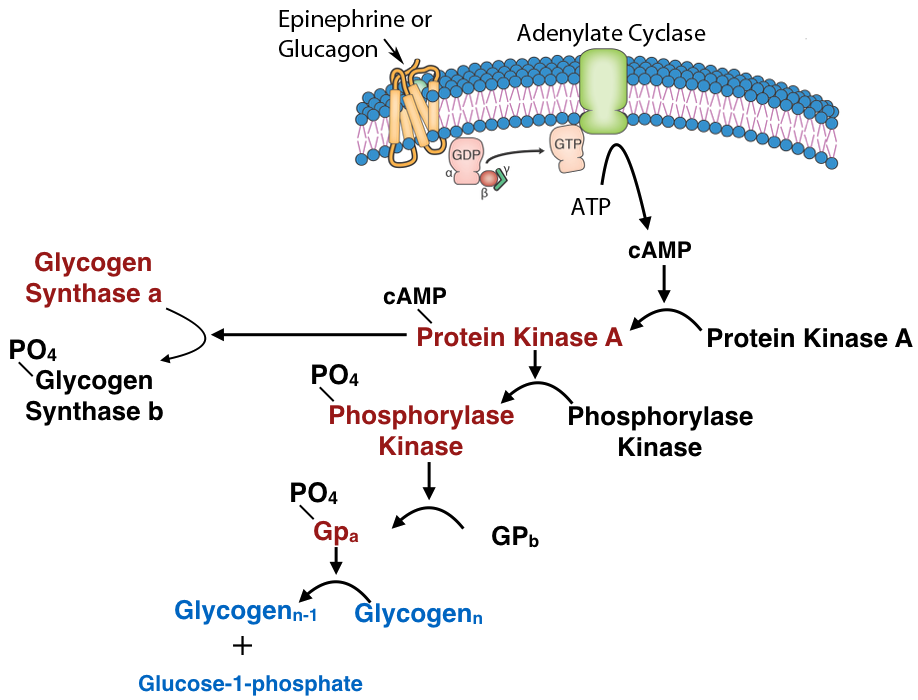
Fosforilación/desfosforilación
Otro mecanismo común para el control de la actividad enzimática por modificación covalente es la fosforilación. La fosforilación de enzimas (en las cadenas laterales de residuos de serina, treonina o tirosina) se lleva a cabo por las proteínas quinasas. Las enzimas activadas por fosforilación pueden regularse mediante la adición de grupos fosfato por quinasas o su eliminación por fosfatasas. Así, este tipo de modificación covalente es fácilmente reversible, en contraste con la escisión proteolítica.
Reducción/oxidación
Un interesante control covalente de enzimas mediante reducción/oxidación se exhibe en plantas fotosintéticas. En la fase de luz de la fotosíntesis, los electrones son excitados por la luz y fluyen a través de portadores hacia NADP+, formando NADPH. Así, a la luz, la concentración de NADPH es alta. Cuando la concentración de NADPH es alta, la concentración de ferredoxina reducida (una molécula que dona electrones a NADP+) también es alta.
La ferredoxina reducida puede transferir electrones a tiorredoxina, reduciéndola. La tiorredoxina reducida puede, a su vez, transferir electrones a proteínas para reducir sus enlaces disulfuro. Cuatro enzimas relacionadas con el ciclo Calvino pueden recibir electrones de la tiorredoxina y activarse, como resultado.
Estos incluyen sedoheptulosa 1,7-bisfosfatasa, ribulosa-5-fosfato quinasa, fructosa 1,6-bisfosfatasa y gliceraldehído 3-fosfato deshidrogenasa. Así, a la luz, los electrones fluyen, haciendo que el NADPH se acumule y la ferredoxina empuje electrones en la dirección de estas enzimas arriba, activándolos y favoreciendo el ciclo Calvino. En la oscuridad, la concentración de NADPH reducido, ferredoxina reducida y disminución de la caída de tiorredoxina, resultando en la pérdida de electrones por las enzimas del ciclo Calvino (oxidaciones que reforman enlaces disulfuro) y el ciclo Calvino se inactiva.
Otros mecanismos de control enzimático
Otros medios de control de enzimas se relacionan con el acceso al sustrato (control a nivel de sustrato) y el control de la síntesis enzimática. La hexoquinasa es una enzima que está ampliamente regulada por la disponibilidad de su sustrato, la glucosa. Cuando la concentración de glucosa es baja, el producto de la catálisis de la enzima, glucosa-6-fosfato, inhibe la función de la enzima.
La regulación de las enzimas mediante el control de su síntesis se aborda más adelante en el libro en la discusión relativa al control de la expresión génica.


