8.6: Aislamiento de genes
- Page ID
- 53252
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Anteriormente en este capítulo, discutimos métodos como la cromatografía en columna que se utilizan para purificar proteínas de interés. Mediante combinaciones de estos métodos, es posible aislar una proteína con un alto grado de pureza, lo que nos permite estudiar la actividad y propiedades de la proteína. Este problema es más difícil de resolver para los ácidos nucleicos. El ADN genómico se puede obtener fácilmente de las células, pero es demasiado complejo para analizarlo en su conjunto. Los genes individuales son las unidades de ADN que corresponden a las proteínas, y así, tiene más sentido aislar genes específicos para su estudio. Los métodos para aislar genes no estuvieron disponibles hasta la década de 1970, cuando el descubrimiento de enzimas de restricción y la invención de la clonación molecular proporcionaron, por primera vez, formas de obtener grandes cantidades de fragmentos específicos de ADN, para su estudio. Si bien, con el propósito de obtener grandes cantidades de un fragmento de ADN específico, la clonación molecular ha sido reemplazada en gran medida por amplificación directa utilizando la reacción en cadena de la polimerasa descrita más adelante, los ADN clonados siguen siendo muy útiles por diversas razones. El desarrollo de la clonación molecular dependió del descubrimiento de endonucleasas de restricción, descritas a continuación.
Enzimas de restricción
Las enzimas de restricción, o endonucleasas de restricción, son enzimas elaboradas por bacterias. Estas enzimas protegen a las bacterias degradando moléculas de ADN foráneas que son transportadas a sus células por, por ejemplo, un bacteriófago invasor. Cada enzima de restricción reconoce una secuencia específica, generalmente de cuatro o seis nucleótidos en el ADN. Estas secuencias, cuando se presentan en el propio ADN de la bacteria, se modifican químicamente por metilación, de manera que no son reconocidas y degradadas. Donde estas secuencias ocurren en ADN extraño, son cortadas por la enzima de restricción.
La utilidad e importancia de las enzimas de restricción radica en su capacidad para reconocer secuencias específicas en el ADN y cortar cerca o (generalmente) en el sitio que reconocen. Se conocen más de 3000 enzimas de este tipo. Las secuencias reconocidas por estas enzimas suelen tener una longitud de 4-8 pares de bases y las enzimas más utilizadas reconocen secuencias descritas como palindrómicas.
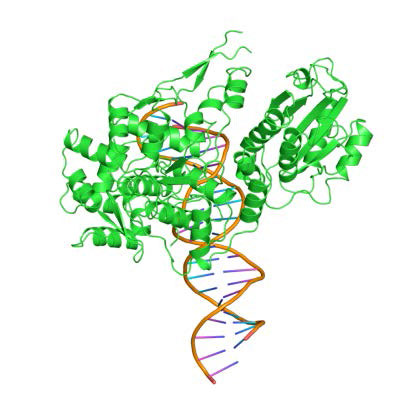
Figura\(\PageIndex{1}\): -Una enzima de restricción unida a su secuencia de reconocimiento en el ADN. Wikipedia
Palíndromo
En biología molecular, el término palíndromo significa que la secuencia del sitio de reconocimiento cuando se lee en la dirección 5' a 3' para la cadena superior es exactamente la misma que la de la cadena inferior. Considere la secuencia reconocida por la enzima de restricción conocida como Hind III (pronunciada hin-dee-three). Es
5' -A-A-G-C-T-3'
3' -T-T-C-G-A-A-5'
En la cadena superior, la secuencia de reconocimiento es
5' AAGCTT 3'
que es lo mismo que el hilo inferior (leído en la misma dirección 5' a 3').
Si bien todas las enzimas de restricción deben reconocer y unirse a secuencias de ADN particulares, la mancha exacta en la que cortan el ADN varía. Algunas enzimas dejan una secuencia escalonada después del corte que tiene un saliente en el extremo 5' de una hebra del dúplex; algunas dejan una secuencia escalonada después del corte que tiene un saliente en el extremo 3'; y algunas cortan ambas hebras en el mismo lugar, sin dejar ninguna secuencia que sobresalga, llamadas cortadoras de extremo romo.
Considere cortar una secuencia de ADN que contenga el sitio de reconocimiento Hind III, que es
5' -A-A-G-C-T-3'
3' -T-T-C-G-A-A-5'
Incrustada dentro de una secuencia de ADN, la secuencia de Hind III se vería así (las Ns corresponden a cualquier base y representan todo el ADN alrededor del sitio de reconocimiento).
5'
-N-N-N-A-A-G-C-T-T-N-N-3' -N-N-N-T-T-C-G-A-A-N-N-N-5'
Después de cortar con Hind III, se vería de la siguiente manera:
5' -N-N-N-A 3' 5'A-G-C-T-T-N-N-N-3'
-N-N-3' -N-N-T-T-C-G-A-5' 3' A-N-N-N-5' 3' A-N-N-N-5'
donde se han insertado huecos para ilustrar dónde se ha producido el corte. Hind III corta entre los dos nucleótidos que contienen 'A' cerca del extremo 5' de la secuencia de reconocimiento y así deja salientes 5' (Figura\(\PageIndex{2}\)).
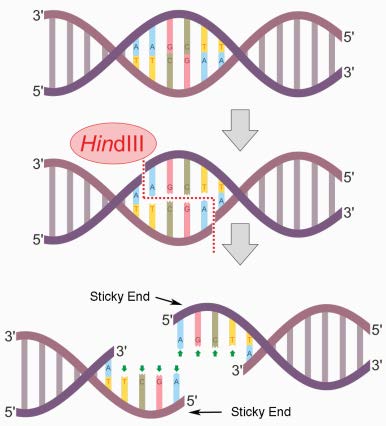
Figura\(\PageIndex{2}\): Resultado del corte de ADN con Hind III. Wikipedia
La enzima de restricción Pst I, por otro lado, reconoce la siguiente secuencia
5' -N-N-N-C-T-G-C-A-G-N-N-N-3'
3' -N-N-N-G-A-C-G-T-C-N-N-N-N-5'
y cortes entre la A y la G cerca del extremo 3' de la secuencia de reconocimiento.
5' -N-N-N-C-T-G-C-A 3' 5'G-N-N-N-N 3'
3' -N-N-N-G 5' 3' A-C-G-T-C-N-N-N-N 5'
Como puede ver, cortar un ADN con Pst I deja salientes 3' de la secuencia de reconocimiento. “Los extremos dejados después del corte por una enzima de restricción que sobresalen en el extremo 5' o en el extremo 3' se denominan “" pegajosos "” porque pueden formar pares de bases apropiados y unirse más fácilmente a un “" extremo pegajoso "” similar.” Esto significa que puedes tomar dos piezas de ADN no relacionadas, cortarlas con la misma enzima de restricción para que tengan extremos pegajosos compatibles, y luego “pegarlas” juntas usando ADN ligasa para formar una nueva molécula híbrida, o recombinante.
Elaboración de ADN recombinantes
La unión de fragmentos de ADN de diferentes fuentes crea ADN recombinante. La capacidad de cortar y pegar ADN puede parecer puramente una hazaña técnica, pero una aplicación clave que surgió de esto es la clonación molecular. En la clonación molecular se puede insertar un gen de interés en un vector, generalmente un plásmido, cortando tanto el vector como el gen (llamado inserto) con la misma enzima para generar extremos pegajosos y uniendo las dos piezas entre sí para generar un recombinante (Figura\(\PageIndex{3}\)). Un plásmido es un tipo de ADN extracromosómico de replicación autónoma. Es bastante sencillo extraer plásmidos de las células, diseñarlos para que contengan el gen de interés y reintroducir el plásmido recombinante en la bacteria. La idea era que cuando se replicara el ADN plasmídico, también se copiara el gen extra insertado. Así, al crecer gran parte de las bacterias portadoras del plásmido, se podrían obtener muchas copias del gen de interés, para proporcionar cantidades suficientes del gen para usar en experimentos. Si bien ahora tenemos métodos más fáciles para lograr este objetivo, los ADN clonados siguen siendo muy útiles. Por ejemplo, es posible clonar un gen que codifica una proteína de interés para que pueda expresarse a altos niveles en las células en las que se introduce el plásmido recombinante.
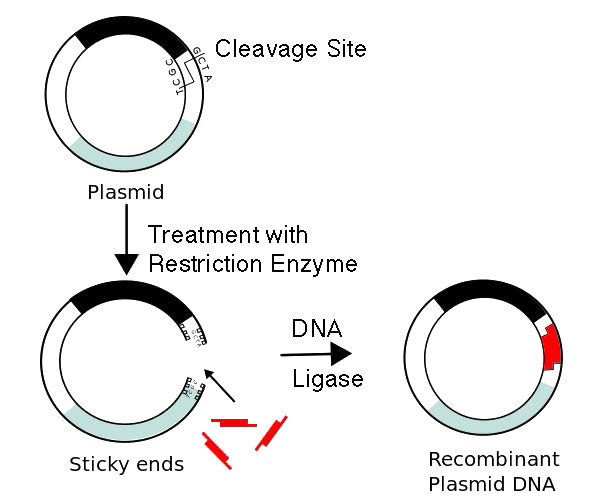
Figura\(\PageIndex{3}\): Construcción de ADN recombinante. Wikipedia
Cualquiera que sea el propósito para el que se elabora el plásmido recombinante, normalmente lleva un gen (o genes) de resistencia a antibióticos, llamado marcador seleccionable. Las células que absorben el plásmido podrán crecer en presencia del antibiótico. Si las células bacterianas a las que se ha agregado el plásmido se colocan en placas sobre agar que contiene el antibiótico, las células que tomaron el plásmido podrán crecer, mientras que las demás no.
Clonación de expresión
Como se mencionó anteriormente, un gen de interés puede insertarse en un vector y el plásmido recombinante colocarse en una célula donde se pueda expresar el gen. Por ejemplo, uno podría desear clonar el gen que codifica para la hormona del crecimiento humano o insulina u otras proteínas médicamente importantes y tener una bacteria o levadura que haga grandes cantidades de ella de manera muy barata. Recuerde que estas son proteínas humanas, y por lo tanto no es factible extraer las proteínas en ninguna cantidad de sujetos humanos.
Para clonar un gen para que pueda expresarse, es necesario establecer las condiciones adecuadas para que la proteína humana se haga en las células bacterianas. Esto implica típicamente el uso de plásmidos especialmente diseñados. Estos plásmidos han sido diseñados para 1) replicarse en números altos; 2) portar marcadores que permiten a los investigadores identificar células que los portan (resistencia a antibióticos, por ejemplo) y 3) contienen secuencias (como un promotor y una secuencia Shine Dalgarno) necesarias para la expresión de la proteína deseada, con sitios convenientes para la inserción del gen de interés en el lugar apropiado en relación con las secuencias de control. Un plásmido que tiene todas estas características se denomina vector de expresión. Además de los plásmidos que pueden usarse para la expresión en células bacterianas, también están disponibles vectores de expresión que permiten la expresión de proteínas en una variedad de células eucariotas.
Se han creado muchas variaciones sofisticadas en tales vectores que han facilitado la producción y purificación de grandes cantidades de cualquier proteína de interés para la que se haya clonado el gen. Una característica útil en algunos vectores de expresión es una secuencia que codifica una etiqueta de afinidad hacia arriba o aguas abajo del gen que se expresa. Esta secuencia permite que una etiqueta de afinidad corta (tal como una serie de residuos de histidina) se fusione a la proteína codificada. La etiqueta se puede utilizar para purificar fácilmente la proteína, como se describe en la sección sobre cromatografía de afinidad.


