8.8: Transcripción inversa
- Page ID
- 53288
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)En el dogma central, el ADN codifica ARNm, que codifica para proteína. Una excepción conocida al dogma central es exhibida por los retrovirus. Estos virus codificados por ARN tienen una fase en su ciclo de vida en la que su ARN genómico es convertido de nuevo en ADN por una enzima codificada viralmente conocida como transcriptasa inversa. La capacidad de convertir ARN en ADN es un método que es deseable en el laboratorio por numerosas razones. Por ejemplo, la conversión de ARN de interés en ADNc se usa en RT-PCR así como en otras aplicaciones como el análisis de micromatrices.
Proceso
Primero, se crea un oligonucleótido de ADN que sirve como cebador para que la transcriptasa inversa se use en un ARN diana. El cebador debe, por supuesto, ser complementario a un segmento (cerca del extremo 3') del ARN a amplificar. Se mezclan el ARN, la transcriptasa inversa, el cebador y cuatro dNTPs. Con una ronda de replicación, el ARN se convierte en una sola cadena de ADN. La desnaturalización libera el ADNc monocatenario, que puede usarse tal cual, o convertirse en ADNc bicatenario, dependiendo de la aplicación.
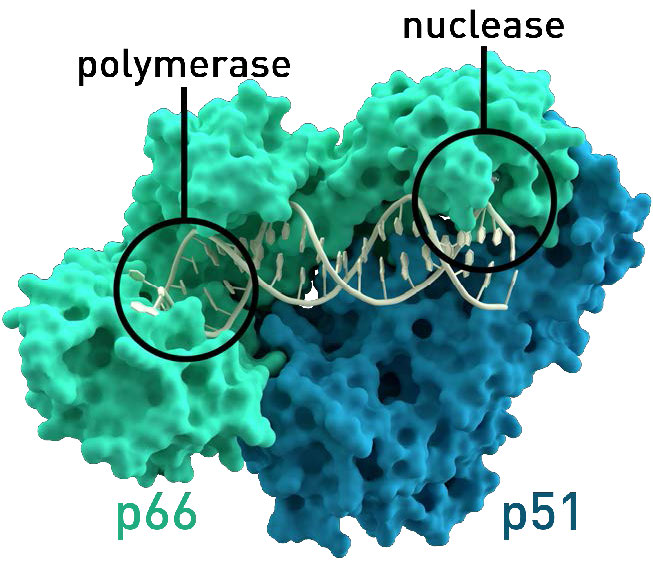
Figura 8.37 - Transcriptasa inversa del VIH. La función nucleasa es necesaria para el ciclo de vida viral, pero no para uso en laboratorio. Wikipedia
Detección de interacciones moleculares
El estudio de la bioquímica es básicamente el estudio de las interacciones de las moléculas celulares. Los métodos para detectar interacciones entre biomoléculas son, por esta razón, muy útiles para los bioquímicos. Ahora discutiremos un par de métodos muy diferentes para detectar estas interacciones intermoleculares.
Sistema de dos híbridos de levadura (Y2H)

El cribado de dos híbridos de levadura es una técnica sofisticada para identificar qué proteína (s), de una colección de todas las proteínas de una célula, interactúa con una proteína específica de interés. El método se basa en la interacción entre dos proteínas para reconstituir un activador transcripcional funcional dentro de las células de levadura. Quizás recuerdes que muchos activadores transcripcionales son proteínas modulares que tienen un dominio que se une al ADN y otro dominio que activa la transcripción (Figura 8.38).
Si el factor de transcripción se divide, de manera que el dominio de unión se une a una proteína, y el dominio de activación a otra proteína, un activador transcripcional funcional solo puede recrearse si las dos proteínas “portadoras” entran en estrecha proximidad, es decir, interactúan. La presencia de este activador funcional puede detectarse mediante la expresión de un gen informador.
Una manera sencilla de entender esta idea es pensando en un activador transcripcional como un dispositivo, como una linterna, que tiene dos partes, la batería y la lámpara, que deben estar juntas para funcionar. Ni una persona que solo tiene una batería ni una que solo tiene la lámpara podrá ver en un cuarto oscuro. Pero si los dos interactúan acercando lo suficiente como para insertar la batería en la linterna, su interacción puede ser detectada por el hecho de que la linterna ahora será funcional como lo demuestra la luz producida.
Se necesitan dos para bailar tango
La Figura 8.39 (A) muestra el activador transcripcional normal de levadura, GAL4, con los dominios de unión al ADN (DBD) y Activación (AD). Es capaz de estimular la transcripción del gen indicador aguas abajo, lac z. Los paneles B y C muestran constructos que producen el DBD GAL4 y AD, respectivamente, fusionados con otras proteínas, una de las cuales se denomina “cebo” y la otra como “presa”. Ninguna de estas proteínas de fusión puede estimular la transcripción del gen lac z. Cuando los constructos que codifican tanto el cebo como la presa están en la misma célula de levadura, si la proteína cebo interactúa con la presa, el DBD y AD del GAL4 se unirán para reconstituir un GAL4 funcional. La presencia de GAL4 funcional es fácilmente detectable porque estimulará la expresión del gen indicador lac z. Si las proteínas de cebo y presa no interactúan, entonces no habrá expresión de lac z. Cuando se detecta interacción a través de la expresión del gen informador, se puede identificar la proteína presa específica.
El sistema de dos híbridos de levadura permite el cribado simultáneo de muchas proteínas presas, mediante la construcción de grandes colecciones de construcciones de fusión, con cada pareja proteica potencial de la proteína cebo fusionada al dominio de activación de GAL4.
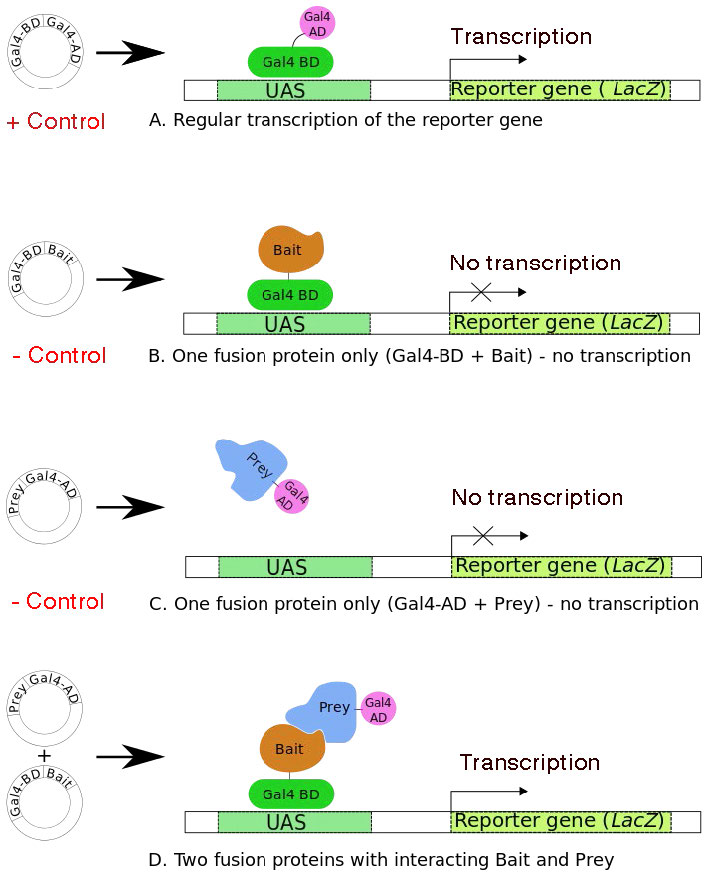
Figura 8.39 - Cuatro escenarios para el sistema de dos híbridos de levadura. UAS = Secuencias Activadoras Aguas arriba - actúa como un promotor. El escenario A muestra que los dos factores de transcripción parten como una sola proteína. Wikipedia


