8.11: Escisión de Proteína
- Page ID
- 53236
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Debido a su gran tamaño, las proteínas intactas pueden ser difíciles de estudiar utilizando técnicas analíticas, como la espectrometría de masas. En consecuencia, a menudo es deseable descomponer un polipéptido grande en trozos más pequeños. Las proteasas son enzimas que típicamente rompen los enlaces peptídicos al unirse a secuencias de aminoácidos específicas en una proteína y catalizar su hidrólisis.
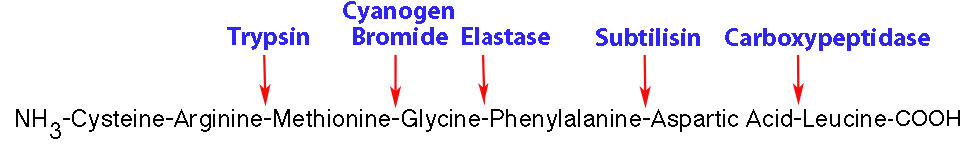
Los reactivos químicos, como el bromuro de cianógeno, que escinde enlaces peptídicos en el lado C-terminal de un residuo de metionina también se pueden usar para cortar proteínas más grandes en péptidos más pequeños. Las proteínas comunes que realizan esta actividad se encuentran en el sistema digestivo y se muestran a continuación.
- Subtilisina - lado C-terminal de grandes cadenas laterales sin carga
- Quimotripsina - lado C terminal de aromáticos (Phe, Tyr, Trp)
- Tripsina - lado C-terminal de lisina y argininas (no junto a la prolina)
- Carboxipeptidasa - lado N-terminal del aminoácido C-terminal
- Elastasa - Hidroliza el lado C de AA pequeños (Gly, Ala)
- Bromuro de cianógeno (químico) - Hidroliza el lado C de Met
Determinar la masa y la secuencia proteica
La espectrometría de masas, como su nombre indica, es un método que puede utilizarse para determinar las masas de moléculas. Una vez limitado al análisis de moléculas pequeñas, desde entonces se ha adaptado y mejorado para permitir el análisis de moléculas biológicamente importantes como proteínas y ácidos nucleicos. Los espectrómetros de masas utilizan un campo eléctrico para acelerar una molécula ionizada hacia un detector. El tiempo que tarda una molécula ionizada en pasar de su punto de ionización al detector dependerá tanto de su masa como de su carga y se denomina su tiempo de vuelo (TOF).
MALDI-TOF

MALDI-TOF (Matrix-Assisted Laser Desorption Ionization - Time of Flight) es una técnica analítica que permite determinar las masas moleculares de moléculas biológicamente relevantes con gran precisión. Se usa comúnmente en proteómica y determinación de masas de biomoléculas grandes, incluyendo ácidos nucleicos. El desarrollo de MALDI, que permite la producción de formas iónicas de moléculas relativamente grandes, fue crucial para el uso exitoso de la espectrometría de masas de biomoléculas. La Figura 8.46 muestra un sistema compacto MALDI-TOF.
El proceso MALDI-TOF implica tres etapas básicas. Primero, el material a analizar se incrusta en material de soporte sólido (matriz) que puede volatilizarse en una cámara de vacío mediante un rayo láser. En la segunda parte del proceso, un láser enfocado en la matriz volatiliza la muestra, provocando que las moléculas dentro de ella se vaporicen y, en el proceso, formen iones ya sea ganando o perdiendo protones. Tercero, los iones así creados en la muestra son acelerados por un campo eléctrico hacia un detector. Su velocidad de movimiento hacia el detector es una función de la relación de su masa a carga (m/z). Un ion con una masa de 100 y una carga de +1 se moverá dos veces más rápido que un ion con una masa de 200 y una carga de +1 y a la misma velocidad que un ion con una masa de 200 y una carga de +2. Por lo tanto, al determinar con precisión el tiempo que tarda un ion en pasar de la ionización (tiempo cero del tratamiento con láser) a ser detectado, se puede determinar fácilmente la relación masa/carga para todas las moléculas en una muestra.
La ionización puede resultar en la desestabilización de moléculas más grandes, que se fragmentan en otras más pequeñas en la cámara de detección MALDI-TOF. El tamaño de cada uno de los subfragmentos de una molécula más grande permite determinar su identidad si ésta no se conoce previamente. Esta fragmentación se puede mejorar intencionalmente haciendo que los iones acelerados colisionen con un gas inerte, como el argón.
La fragmentación de una molécula también se puede llevar a cabo antes del análisis, como por ejemplo, escindiendo una proteína en péptidos más pequeños mediante el uso de enzimas o agentes químicos. La secuencia de aminoácidos de una proteína se puede determinar usando MALDI-TOF analizando las masas moleculares precisas de los muchos fragmentos peptídicos cortos obtenidos de una proteína. Cuando un aminoácido, por ejemplo, fragmentos de un péptido más grande, esto puede detectarse como la diferencia de masa entre el fragmento con y sin el aminoácido, ya que cada aminoácido tendrá una masa molecular característica. Mediante la toma de huellas de masa peptídica y el análisis de fragmentos más pequeños de péptidos individuales, se puede determinar la secuencia completa de un polipéptido.


