1.5: Replicación de ADN - Introducción a la replicación procariota
- Page ID
- 57192
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
1953
- El modelo de ADN de Watson y Crick sugirió cómo se podría replicar la información genética: cualquiera de las cadenas del dúplex puede usarse como molde para replicar la información de la secuencia.
- Pero, ¿la replicación era conservadora (es decir, las cadenas parentales originales permanecen juntas después de la replicación) o semiconservadora (una hebra parental se empareja con una hebra recién sintetizada)?
La respuesta para los organismos procariotas (es decir, carecen de un núcleo unido a la membrana verdadera y orgánulos celulares; por ejemplo, bacterias) provino del experimento de 1958 de Meselson y Stahl.
1958
El nitrógeno en las sales de amonio en el caldo de cultivo se incorpora a las bases de ADN. El isótopo más común de nitrógeno es 14 N. Sin embargo, también se pueden obtener sales de amonio 15 N (un isótopo más pesado).
- El ADN de células de E. coli cultivadas con sales de amonio 15 N tendrá una densidad mayor que el ADN cultivado en sales de amonio “normales” (14 N).
- Dicho ADN migrará de manera diferente en la centrifugación en gradiente de densidad de equilibrio de cloruro de cesio (CsCl 2).
- El ADN más denso migrará como una banda inferior (en este tipo de centrifugación la posición de migración característica es una función de la densidad, y es independiente de la longitud del ADN).
.png)
Figura 1.5.1: Densidad de ADN de 15 N y 14 N
Meselson y Stahl razonaron que si crecían E. coli en sales 15 N luego cambiaban los medios a 14 N para rondas adicionales de replicación, el modo de replicación podría deducirse de la densidad del ADN.
.png)
Figura 1.5.2: Experimento de Meselson y Stahl
- Después de cambiar a los medios 14 N y permitir que las células pasaran por una ronda de replicación, se observó una sola banda de densidad intermedia (es decir, entre 14 N y 15 N muestras de ADN control).
- Después de una segunda ronda de replicación en medios 14 N, dos bandas estuvieron presentes en cantidades aproximadamente equimolares; una fue intermedia en densidad y la otra migró como ADN marcado puramente con 14 N.
Los resultados fueron consistentes con un modo semi-conservador de replicación para el ADN. Desde entonces se ha obtenido evidencia de replicación semi-conservadora de ADN tanto con ADN vegetal como animal.
Replicación de ADN en E. coli
Características de la ADN polimerasa de E. coli:
- La polimerasa solo alargará un polinucleótido existente. No puede iniciar la formación de polinucleótidos:
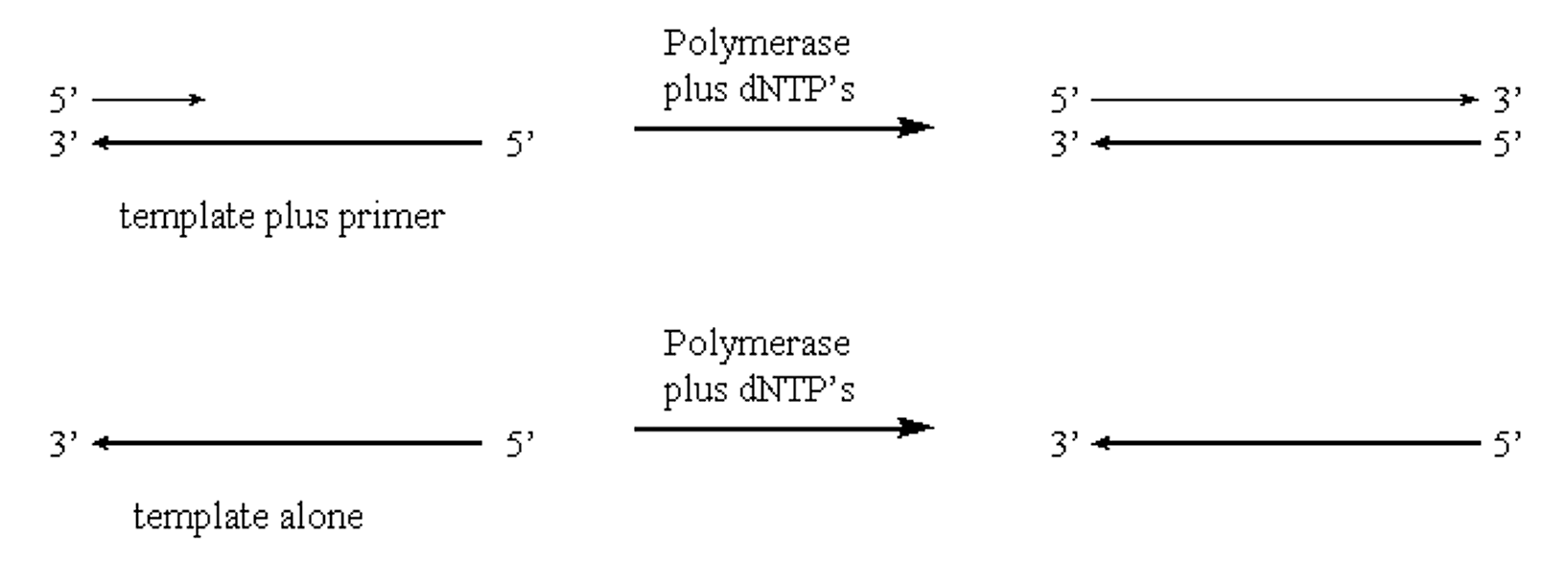.png)
Figura 1.5.3: Actividad de ADN Polimerasa
- La polimerasa catalizará la polimerización de nucleótidos solo en una dirección (5'>3') a través de un enlace fosfodiéster entre un grupo hidroxilo 3' y 5' fosfato.
- La ADN polimerasa es incapaz de desenrollar el ADN dúplex para separar las dos cadenas que deben copiarse
El genoma de E. coli es ADN dúplex circular de aproximadamente 4 x 10 6 pares de bases (es decir, 4 Mb)
- El genoma tiene un único origen de replicación.
- La duplicación de ADN en E. coli comienza en un sitio específico en el ADN llamado “ORic”.
ORiC es una región de ADN de aproximadamente 240 nucleótidos de longitud.
- Contiene secuencias repetitivas de 9 pares de bases y 13 pares de bases (conocidas como las regiones '9-mer' y '13-mer').
- Estas secuencias son regiones ricas en AT, que se funden a temperaturas más bajas que el ADN que contiene pares GC.
- Estas regiones se postulan para ayudar a fundir el dúplex de ADN en la región ORic para el inicio de la replicación del ADN.
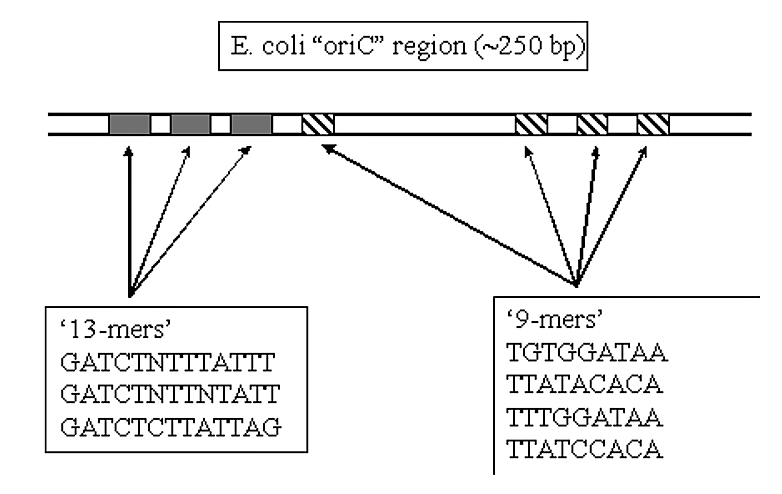.png)
Figura 1.5.4: Región ORi
El producto del gen DNAA: (proteína ADNA)
- Las cepas de E. coli con mutaciones en el gen DNAA pudieron crecer a 30 °C, pero no a 39-42 °C.
- Sin embargo, si la síntesis de ADN se inició a 30 °C, y luego la temperatura se desplazó a 42 °C, la síntesis de ADN continuó hasta que se replicó el genoma (y la célula se dividió), pero no fue posible una nueva iniciación de la síntesis de ADN.
Conclusión: De alguna manera, el producto del gen DNAA (es decir, la proteína DNAA) es necesario para iniciar la síntesis de ADN.
Estudios de proteína DNAA purificada:
- La proteína DNAA se une a la región '9-mer' en ORic y forma un complejo multimérico con 10-20 subunidades de proteína (es decir, en una sola región ORic habrá 10-20 moléculas de proteína de ADN unidas).
- La encuadernación requiere ATP.
- Se observó una adición adicional de ATP para dar como resultado una fusión y apertura del dúplex de ADN en la región ORic. Esto se determinó mediante la adición de nucleasa S1 (como frijol mungo, pero también cortará ADN en el sitio de una mella interna), lo que resultó en la escisión del ADN en el sitio de ORic.
El producto del gen DnaB: (proteína DnaB)
La proteína codificada por el gen DnaB parece ser esencial para la replicación del ADN. La proteína DnaB ha sido identificada como una helicasa. Una helicasa se mueve a lo largo de una cadena de ADN abriendo el dúplex para fundir y separar las cadenas de ADN.
- La proteína DnaB se une al ADN monocatenario en la región general del segmento de ADN ORic.
- La unión requiere ATP así como el producto génico de DnaC (la proteína DnaC).
- Después de que la helicasa/DNAc se une al ADN, se libera la proteína DNAc.
- Dos helicasas se unen en la región ORi, una helicasa en cada hebra del ADN.
Esta etapa representa el complejo de precebado:
.png)
Figura 1.5.5: Complejo de precebado
Se evita que las cadenas separadas en la región ORic vuelvan a aparearse mediante la unión de la proteína de unión monocatenaria (proteína ssb).
La proteína del gen DnAG:
La proteína del gen DnaG se llama primasa.
- La primasa cataliza la síntesis de moléculas cortas de ARN que funcionan como cebadores para la síntesis de ADN por la ADN polimerasa III de E. coli (pol III).
- La primasa se une a la proteína DnaB en orIC y forma un primosoma.
- La primasa dentro del complejo primosoma proporciona cebadores de ARN para la síntesis de ambas cadenas de ADN dúplex.
- La primasa establece huellas de PPPac (N) 7-10 (ARN).
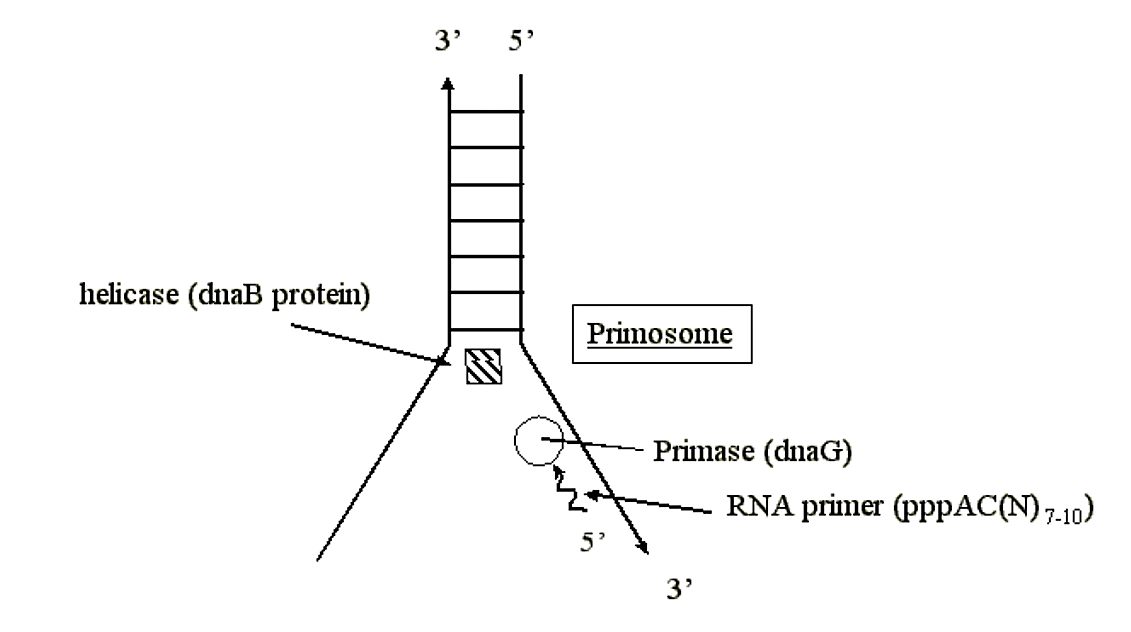.png)
Figura 1.5.6: Actividad de primasa
- Después de la síntesis del cebador de ARN 9-12 mer, la holoenzima DNA Pol III ingresa a la horquilla de replicación y es capaz de utilizar el ARN como cebador para la síntesis de ADN.
- A medida que se abre la bifurcación de replicación, la síntesis de la cadena principal puede continuar, pero se desarrolla una brecha en la hebra rezagada:
.png)
Figura 1.5.7: Filamento rezagado
DNA Pol III es una gran enzima multicompleja (holoenzima) que es algo dimérica por naturaleza (hay dos sitios activos de polimerasa). Los dos sitios de polimerasa activos en Pol III podrían funcionar para sintetizar ambas cadenas nacentes en la horquilla. Sin embargo, la síntesis de la hebra plantilla rezagada estaría en la dirección opuesta al movimiento del complejo Pol III:
.png)
Figura 1.5.8: Movimiento de Pol III
La primasa puede unirse al complejo Pol III, pero la disposición de la cadena de ADN a medida que pasa a través del complejo Pol III/primasa es bastante única. Forma una estructura de bucle tal que la primasa y el sitio activo de Pol III pueden lograr la síntesis discontinua de la cadena molde rezagada aunque la dirección general del complejo Pol III sea opuesta a la dirección requerida de la síntesis de ADN:
.png)
Figura 1.5.9: Bucle para la actividad de Pol III
Después de que la primasa haga otro cebador en el molde rezagado, el sitio activo Pol III adyacente puede extender el cebador (incorporando dNTP) utilizando la misma estructura de bucle y alimentando el molde en la dirección mostrada.
.png)
Figura 1.5.10: Síntesis de la hebra rezagada
El bucle de cadena retrasada no puede alimentarse a través del complejo Pol III para siempre, y después de sintetizar una cadena de ADN naciente, se libera el bucle y se forma uno nuevo usando el ADN molde abierto más arriba en la horquilla:
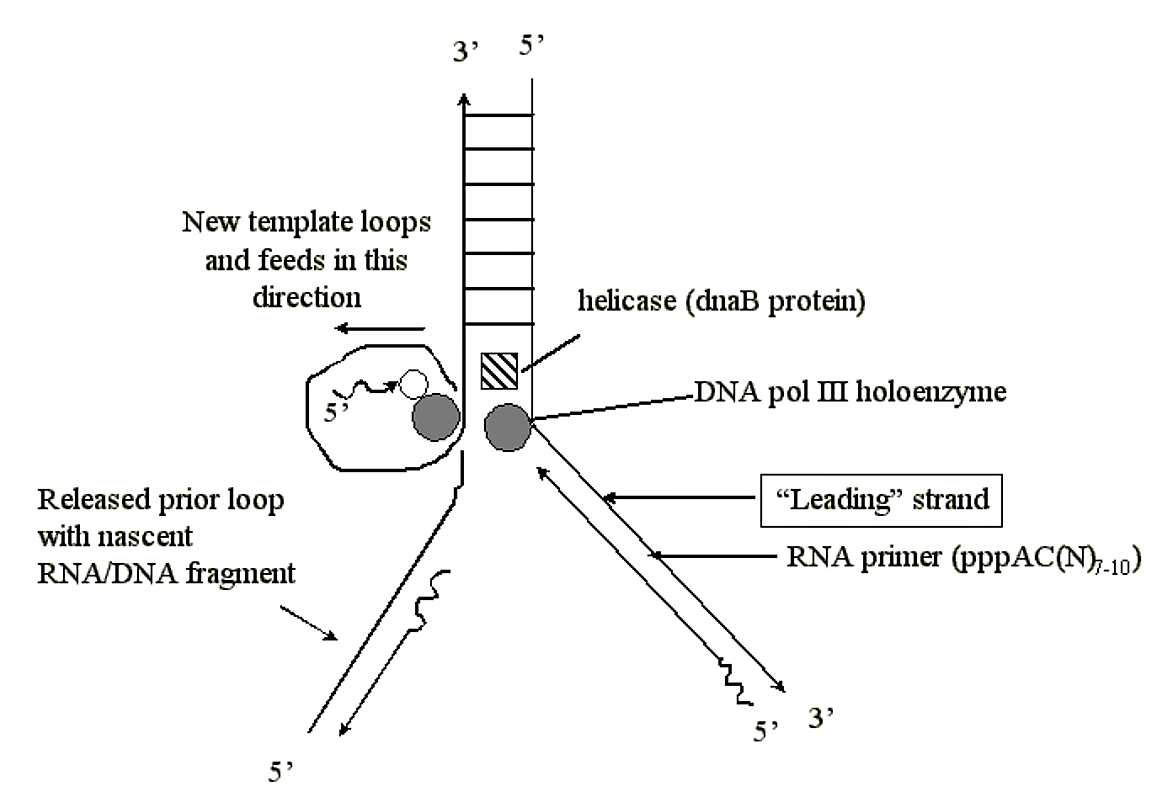.png)
Figura 1.5.11: Continuación de la síntesis de hebras rezagadas
A medida que continúa la síntesis:
- habrá una sola cadena continua de ADN en la cadena principal
- habrá una serie de fragmentos cortos en la hebra rezagada, que contienen tanto ARN como ADN, llamados fragmentos de Okazaki:
.png)
Figura 1.5.12: Fragmentos de Okazaki
¿Cómo se convierten estos fragmentos de ARN/ADN en una larga cadena continua de ADN? El ARN podría ser eliminado por una polimerasa que tiene actividad 5'->3' exonucleasa, sin embargo, Pol III carece de esta actividad.
- El ADN Pol I tiene actividad exonucleasa 5'->3'
- puede extender la síntesis de ADN a través de la traducción de mellas.
- La actividad de traducción de mellas se regenera en la degradación de los cebadores de ARN.
- El resultado final es una serie de “mellas” en la hebra rezagada, ahora 100% ADN:
.png)
Figura 1.5.13: Mallas en hebra rezagada
- Las hojas de ADN Pol I y la ADN ligasa luego se unen a estos fragmentos de ADN discontinuos para formar un dúplex de ADN continuo en la hebra rezagada.
Resumen de pasos en la síntesis de ADN de E. coli
- La proteína DNAA se funde dúplex en la región ORi.
- DnaB (helicasa), junto con DnaC y ATP se une a la horquilla de replicación (salidas de proteína DnaC). 1 (Complejo de precebado)
- La proteína de unión monocatenaria (proteína ssb) se une a cadenas separadas de ADN y evita la reasociación.
- Los complejos de primasa con helicasa, crean cebadores de ARN (PPPac (N) 7-10) en las cadenas del dúplex abierto 2 (Primasa+helicasa constituyen el Primosoma).
- Después de hacer los cebadores de ARN, entra la holoenzima ADN pol III y extiende el cebador de ARN (que establece dNTP) en la cadena principal.
- A medida que la bifurcación de replicación se abre (a través de la acción helicasa + ATP), la síntesis de la cadena principal es un proceso ininterrumpido, la cadena rezagada experimenta una brecha.
- La región gap de la cadena rezagada puede enrollarse alrededor de una unidad de sitio activo del complejo Pol III, y la Primase unida inicia un cebador de ARN en la región de brecha 3.
- En la cadena retrasada, Pol III extiende el cebador de ARN con dNTP a medida que la cadena molde rezagada se enrolla a través del complejo Pol III
- Después de la síntesis de un fragmento naciente, se libera el bucle de cadena retrasada y la región monocatenaria más arriba cerca de la horquilla de replicación se enlaza posteriormente a través del complejo Pol III.
- Se repiten los pasos 7-9.
- Mientras tanto, Pol I elimina las regiones del cebador de ARN de los fragmentos de Okazaki a través de la actividad exonucleasa 5' a 3' (traducción de mella
- Pol I sale y la ligasa une los fragmentos de ADN (en la hebra rezagada).
Notas desde arriba
- Las polimerasas son incapaces de abrir el ADN dúplex, por lo que el requisito de helicasa
- Las polimerasas no pueden replicar un molde de ADN en ausencia de un cebador (ya sea ADN o ARN).
- Las polimerasas extienden un polinucleótido solo en la dirección 5' a 3'. Los huecos en el extremo 5' deben ser llenados por síntesis discontinua “aguas arriba”.
|
|
ADN Pol I |
ADN Pol II |
ADN Pol III |
|---|---|---|---|
|
5'->3' Actividad de la polimerasa |
* |
* |
* |
|
3'->5' Actividad exonucleasa (lectura de prueba) |
* |
* |
* |
|
5'->3' Actividad exonucleasa (traducción de mella) |
* |
||
|
Síntesis a partir de: |
|||
|
ADN dúplex |
|||
|
Una sola hebra imprimada |
* |
||
|
Proteína monocatenaria cebada más ssb |
* |
* |
|
|
Velocidad de elongación de cadena (in vitro) bp/min |
600 |
? |
30,000 |
|
Moléculas/célula |
400 |
? |
10-20 |
|
¿Mutación letal? |
* |
* |
Funciones de Pol:
- Pol I: llenado de huecos durante la síntesis y reparación de ADN, eliminación de cebadores de ARN
- Pol II: involucrado en la síntesis de ADN de plantillas dañadas
- Pol III: polimerasa funcional en la horquilla de replicación
Pol III Estructura y función
- Un complejo “holoenzimático” de 10 polipéptidos diferentes
- el peso molecular resultante es mayor de 600 kDa (es decir, es un complejo grande).
- Es estructuralmente un dímero asimétrico - contiene dos copias de la mayoría de los polipéptidos que lo comprenden, incluyendo dos sitios catalíticos para la adición de nucleótidos (es decir, polimerización).
Las diversas subunidades proteicas tienen una variedad de funciones:
- Subunidades para actividad polimerasa: a, e, subunidades
- Subunidades para dimerizar la polimerasa central (t)
- Subunidades para aumentar la procesividad (es decir, para aumentar la capacidad de sintetizar tramos largos sin liberación del molde de ADN): subunidades b
- Subunidades para unir b al sustrato de cebador de ADN: (g, d, d ', c,)
Terminación de la replicación del ADN
- Los sitios específicos de terminación de la replicación del ADN existen en E. coli.
- La terminación implica la unión del producto del gen tus (proteína tus).
- Esta proteína puede actuar para evitar que la helicasa desenrolle el ADN (por lo tanto, detendrá la acción de pol III y pol I).
- La replicación del ADN produce dos anillos entrelazados que deben ser separados.
- Esto se logra a través de la enzima topoisomerasa.
ColE1 Plasmídico
E. coli puede contener un pequeño elemento extracromosómico llamado plásmido ColE1. Este plásmido tiene las siguientes características generales:
- ADN dúplex circular de 6.4 Kb
- Replicación autónoma
- 10-15 copias por célula (es decir, por cromosoma de E. coli)
Aunque se está replicando de manera autónoma, no contiene un tipo de secuencia ORic para el inicio de la replicación, y no experimenta los mismos pasos en la replicación.
- El plásmido produce (entre otras cosas) dos oligonucleótidos de ARN (ARN I y ARN II)
- El ARN II tiene complementariedad con el origen de replicación ColE1, que contiene una secuencia rica en AT
- La molécula de ARN II unida puede servir como cebador para poliII
- Dado que el ARN II se une solo en una cadena, la replicación del plásmido ColE1 es unidireccional
- El ARN I tiene complementariedad con RNAII, y dicho ARN dúplex no puede servir como cebador de replicación, por lo que el control del número de copias plasmídicas se logra mediante la interacción entre ARN I y ARN II.


