6.3: Unión a ligando
- Page ID
- 57194
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
El reconocimiento molecular es un aspecto clave de prácticamente toda la bioquímica, e implica:
- Replicación de ADN
- Regulación génica
- Transcripción de ADN en ARNm
- Traducción de ARNm en proteínas
- Función enzimática
- Ensamblaje molecular
- Transducción de señales
- Comunicación célula-célula
- Inmunología
Las interacciones intermoleculares, en el ejemplo más simple, implican que dos moléculas se reconozcan y se unan entre sí:
- El ligando
- El receptor
Estos son términos amplios, pero las propiedades generales del receptor y ligando son las siguientes:
- El ligando es típicamente, una molécula pequeña, y se difunde por todo el ambiente hasta que se une a un receptor específico
- El receptor es típicamente una molécula grande, relativamente estacionaria, que contiene un sitio de unión específico para el ligando. Al unirse al ligando, las propiedades estructurales y/o químicas del receptor se alteran típicamente.
Las interacciones entre el receptor y el ligando son típicamente no covalentes
- Existe una complementariedad molecular general entre el receptor y el ligando
- A menudo, pero no siempre, existe un costo entrópico asociado con la unión del ligando al receptor, pero esto está más que compensado por una entalpía favorable (es decir, la complementariedad molecular)
- La energía de unión se utiliza típicamente para causar un cambio estructural asociado con un evento de comunicación molecular
Existen dos métodos generales para estudiar las interacciones receptor/ligando:
-
- Termodinámica de equilibrio, y
- Cinética de asociación y disociación
Análisis de equilibrio ligando/receptor
Los dos estados posibles de una interacción ligando/receptor, y las constantes de velocidad asociadas con su formación, se dan como:
La constante de asociación (formación) de equilibrio, Kf, se da como:
- [RL] es la concentración del complejo, [R] es la concentración de R libre en equilibrio y [L] es la concentración de L libre en equilibrio
- K f tiene unidades de M -1
- Un gran valor para Kf significa que el equilibrio favorece el complejo - hay una alta afinidad del receptor por el ligando
- Un pequeño valor para Kf significa que el equilibrio favorece la separación del receptor y el ligando, y el receptor tiene una baja afinidad por el ligando
Solo para dificultar la vida, el marco de referencia típico al describir la afinidad del receptor por el ligando es hacer referencia a la constante de disociación, Kd:
- K d tiene unidades de M
- Un pequeño valor para Kd significa que el equilibrio favorece el complejo - hay una alta afinidad del receptor por el ligando
- Un gran valor para Kd significa que el equilibrio favorece la separación del receptor y el ligando, y el receptor tiene una baja afinidad por el ligando
En equilibrio, la tasa de formación del complejo RL es igual a la velocidad de disociación:
La expresión de la izquierda es la definición de la constante de disociación, K d, por lo tanto:
La expresión de energía libre y K f y K d
De la expresión original de la formación compleja:
El cambio de energía libre de estado estándar, Δ G 0, para el proceso (es decir, comenzando con 1M todo) se daría como:
Dado que K d es la inversa de K f:
En este caso, se producirá un valor negativo para DG 0 (indicando espontaneidad) cuando K d < 1.0. Esto sucederá cuando haya alta afinidad y el equilibrio favorezca la formación compleja.
Relaciones prácticas entre [R], [L], K d y [RL]
Para la misma concentración de ligando y receptor añadidos, se producirá una formación más compleja con un valor menor de K d
Ejemplo 6.3.1
Se agregan receptor y ligando a una concentración total de 1.0 x 10 -4 M. La K d para el complejo RL es 1.0 x 10 -4 M. ¿Cuál es la concentración de [R], [L] y [RL] en equilibrio?
.png)
El ligando añadido, L, se repartirá entre formas libres y unidas:
L total = L + RL = 1.0 x 10 -4 M
L = 1.0 x 10 -4 M - RL
De manera similar, el receptor agregado, R, se repartirá entre formas libres y unidas:
R total = R + RL = 1.0 x 10 -4 M
R = 1.0 x 10 -4 M - RL
Sustituyendo estos valores de L y R en la expresión por K d rinde:
.png)
Se trata de una cuadrática con valores a = 1, b = -3.0 x 10 -4 y c = 1.0 x 10 -8. Esto produce dos valores posibles para RL:
RL = 2.62 X 10 -4 M o
RL = 3.82 X 10 -5
Dado que el valor máximo posible para [RL] es 1.0 x 10 -4 (es decir, dadas las concentraciones iniciales de R y L, esta es la mayor cantidad de RL que se puede formar), el primer resultado no es posible y [RL] = 3.82 X 10 -5 M
Así, 38.2% del receptor agregado se encuentra en la formación del complejo (es decir, 3.82 X 10 -5 M/1.0 x 10 -4) x 100%
Ejemplo 6.3.2
Misma cantidad de R y L añadidos, pero esta vez el K d para el complejo RL es 1.0 x 10 -6 M. ¿Cuál es la concentración de [R], [L] y [RL] en equilibrio?
.png)
his es un cuadrático con valores a = 1, b = -2.01 x 10 -4 y c = 1.0 x 10 -8. Esto produce dos valores posibles para RL:
RL = 1.11 X 10 -4 M o
RL = 9.05 X 10 -5
Dado que el valor máximo posible para [RL] es 1.0 x 10 -4 (es decir, dadas las concentraciones iniciales de R y L, esta es la mayor cantidad de RL que se puede formar), el primer resultado no es posible y [RL] = 9.05 X 10 -5 M
Así, el 90.5% del receptor agregado se encuentra en la formación del complejo (es decir, 9.05 X 10 -5 M/1.0 x 10 -4) x 100%
Para la misma concentración de ligando y receptor añadidos, se producirá una formación más compleja con un valor menor de K d
Determinación de constantes de disociación (K d) o unión (K f)
La relación entre K d, [R], [L] y [RL] viene dada por:
- Esta es una ecuación con cuatro variables, si conoces tres de ellas, puedes resolver para la cuarta
- En principio, puedes resolver para K d si conoces la concentración de receptor libre [R], ligando libre [L] y complejo receptor/ligando [RL] en equilibrio
Para poder resolver K d, se necesita un ensayo que proporcione las concentraciones molares para cada componente. Sin embargo, esto suele ser difícil de lograr. Es mucho más común tener un ensayo que te proporcione información respecto a la fracción de receptor agregado (o ligando) que está presente en la forma libre o compleja.
- Si [R t] es la concentración total de receptor en la muestra, sabemos que se dividirá en dos poblaciones - unidas y libres:
- La fracción de sitios receptores unidos viene dada por la relación:
- La fracción de receptores unidos es conocida por la variable a, y a tiene un valor entre 0 y 1
- Sustituyendo el valor por [R t] en términos de [R] y [RL] dado anteriormente produce:
- La relación entre [RL] y K d viene dada por la expresión para Kd:
- Esta expresión para [RL] se puede sustituir en la expresión para a para producir:
- Simplifique dividiendo primero por [R]:
- Luego multiplica por K d:
Esta ecuación tiene tres variables:
- La concentración de ligando libre en equilibrio [L]
- La constante de disociación, K d, para el complejo RL
- La fracción de receptor que se encuentra en la forma unida en equilibrio, a
Si conoces dos de estas variables, puedes resolver para la tercera (es decir, si el ensayo experimental cuantifica [L] y a, puedes resolver para K d)
La ecuación anterior se puede manipular adicionalmente sustituyendo el valor de a en términos de [RL] y [R t]:
Esta ecuación tiene cuatro variables:
- La concentración de ligando libre en equilibrio [L]
- La constante de disociación, K d, para el complejo RL
- La concentración de receptor que está en la forma unida en el eqilibrium [RL]
- La concentración total de receptor en la muestra
Por lo general, el experimento de unión al ligando se establece de manera que [Rt] se mantenga constante y [RL] se controle como una función de [L]
Esta ecuación se parece mucho a la ecuación de Michaelis-Menten:
Trazar V versus [S] en la ecuación M-M produce una hipérbola rectangular. Así, trazando [RL] versus [L] producirá igualmente una hipérbola rectangular
- La asíntota de la curva dará el valor de [R t]
- La concentración de ligando libre [L] que da como resultado la mitad del valor máximo de [RL] es igual al valor de K d
Otra modificación es darse cuenta de que si a es igual a la fracción de receptor unido en un complejo (con un valor 0 ≤ α ≤ 1.0), entonces (1 - α) es la fracción de receptor libre (es decir, fracción de receptor no unido)
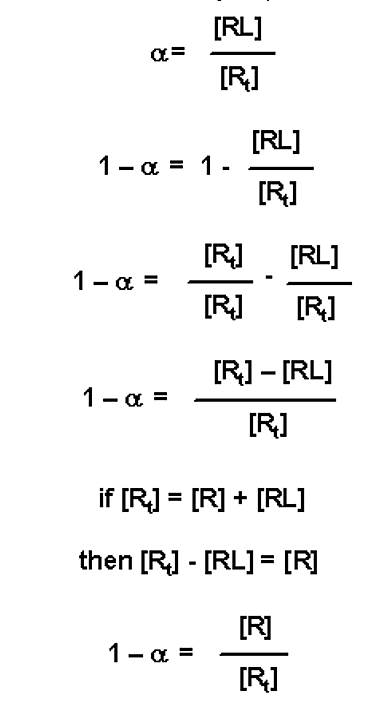.png)
Este valor se puede sustituir en las tres expresiones variables anteriores:
.png)
Esta ecuación tiene cuatro variables:
- La concentración de ligando libre en equilibrio [L]
- La constante de disociación, K d, para el complejo RL
- La concentración de receptor que se encuentra en forma libre en el eqilibrio [R]
- La concentración total de receptor en la muestra [R t]
Por lo general, el experimento de unión al ligando se establece de manera que [Rt] se mantenga constante y [R] se controle como una función de [L]
(Nota:Aunque esto parece una ecuación M-M, K d es una constante.)
Las derivaciones anteriores se denominan "isotermas de unión" en referencia a su capacidad para resolver Kd en condiciones de equilibrio (y la ecuación termodinámica de equilibrio original utilizó temperatura constante)
.png)
Trazar datos para varios valores de [L], [R t], y ya sea [RL] o [R], y ajustar a la función apropiada anterior, permite una determinación más precisa de K d (aunque un solo experimento proporcionará una respuesta).
Debido a la similitud de la gráfica [RL] versus [L] con la gráfica M-M, a menudo es la configuración experimental más deseable. En particular, se puede usar una gráfica de doble reciprocidad (al igual que la gráfica M-M de doble reciprocidad) para determinar con mayor precisión K d
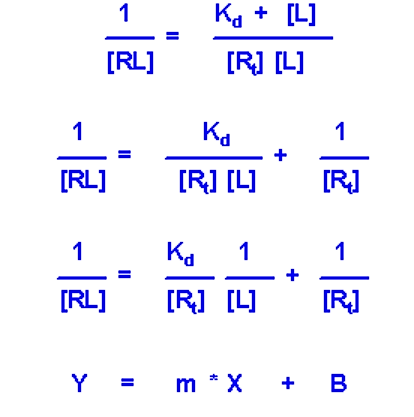.png)
- Si trazas 1/ [RL] versus 1/ [L] obtendrás una línea recta cuya pendiente es igual a K d/[R t]
- Y intercepción será igual a 1/ [R t]. Dado que este valor es fijo y se conoce, el valor de la intercepción y puede servir como una comprobación para asegurarse de que el experimento se ajusta al modelo
Un experimento de diálisis en equilibrio
Se puede usar una membrana semipermeable en un experimento de equilibrio basado en dialización para determinar la Kd para un par ligando-receptor.
- La membrana debe ser libremente permeable en lo que respecta al ligando (generalmente es una molécula pequeña), pero presentar una barrera al receptor
- El experimento se establece con una concentración conocida de receptor en un lado y una concentración conocida de ligando en el otro
- Se permite que el sistema llegue al equilibrio
- El experimento requiere un ensayo para el ligando
.png)
Figura 6.3.1: Barrera semipermeable
Después de alcanzar el equilibrio, se ensaya la concentración de ligando en ambos lados de la membrana semipermeable:
.png)
Figura 6.3.2: Concentración en equilibrio
- Dado que el ligando no se ve afectado por la membrana, la concentración de ligando en el lado sin receptor es igual a la concentración de ligando libre [L] en todas partes de la muestra (es decir, en ambos lados de la membrana)
- La concentración de ligando será igual a la concentración de ligando libre [L] más el ligando presente en el complejo receptor [RL] (en otras palabras, debería haber una mayor concentración de ligando en el lado con el receptor si el receptor tiene afinidad por el ligando)
- La concentración del complejo receptor/ligando [RL] se puede determinar a partir de la diferencia entre estos dos valores
- La concentración total del receptor es un valor conocido (experimentador determinado al inicio del experimento, y no cambia)
- Cualquiera de las isotermas de unión se puede usar para resolver K d (ya que también se puede determinar [R])


