6.4: Mapeo de restricción
- Page ID
- 57193
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
El sistema de restricción/modificación en bacterias es un sistema inmune a pequeña escala para la protección contra la infección por ADN extraño.
A finales de la década de 1960 se descubrió que E. coli contiene enzimas que metilarán bases nucleotídicas específicas en el ADN
· Diferentes cepas de E. coli contenían diferentes tipos de estas metilasas
- Los sitios típicos de metilación incluyen la posición N 6 de la adenina, la posición N 4 de la citosina o la posición C 5 de la citosina.
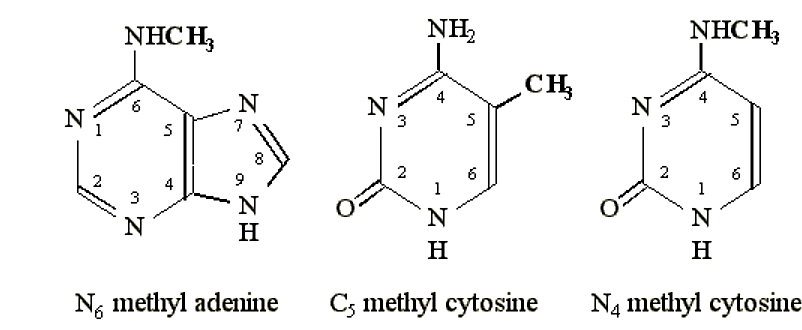.png)
Figura 6.4.1: Sitios de metilación
- Además, solo un porcentaje fraccional de bases estaban metiladas (es decir, no todas las adeninas estaban metiladas, por ejemplo) y estas ocurrieron en sitios muy específicos en el ADN.
- Un rasgo característico de los sitios de metilación, fue que involucraron secuencias de ADN palindrómico.
- Aquí hay un ejemplo de una cepa R1 particular de E. coli:
.png)
Figura 6.4.2: ADN palindrómico
(Especificidad de EcoR1 metilasa. Rubin y Modrich, 1977)
- Además de poseer una metilasa particular, las cepas bacterianas individuales también contenían actividades específicas de endonucleasa acompañantes.
- Las endonucleasas se escindieron en o cerca del sitio de reconocimiento de metilación.
Figura 6.4.3: Escisión cerca del sitio de metilación
- Estas nucleasas específicas, sin embargo, no se escindirían en estas secuencias palindrómicas específicas si el ADN estuviera metilado.
Así, esta combinación de una metilasa específica y una endonucleasa asociada funcionó como un tipo de sistema inmune para cepas bacterianas individuales, protegiéndolas de la infección por ADN extraño (por ejemplo, virus).
- En la cepa bacteriana EcoR1, la secuencia GAATTC será metilada en la base adenina interna (por la metilasa EcoR1).
- La endonucleasa EcoR1 dentro de la misma bacteria no escindirá el ADN metilado.
- El ADN viral foráneo, que no está metilado en la secuencia “GAATTC”, será reconocido como ADN “extraño” y será escindido por la endonucleasa EcoR1.
- La escisión del ADN viral lo hace no funcional.
“Dichas endonucleasas se denominan “" endonucleasas de restricción "” porque restringen el ADN dentro de la célula a ser “" auto "”.”
La combinación de endonucleasa de restricción y metilasa se denomina sistema de “restricción-modificación”.
Dado que diferentes cepas y especies bacterianas tienen potencialmente diferentes sistemas R/M, su caracterización ha hecho disponibles cientos de endonucleasas con diferentes sitios de escisión específicos de secuencia.
- Son una de las principales herramientas de la biología molecular moderna para la manipulación e identificación de secuencias de ADN.
- Las endonucleasas de restricción suelen denominarse así por la bacteria de la que se aisló.
|
Nombre |
Fuente |
Secuencia de reconocimiento |
Comentarios |
|
Alu I |
Artrobacter luteus |
| 5'… A G C T … 3' 3'… T C G A … 5' | |
“Cuatro cortador”. Deja extremos romos al ADN. |
|
Bfa I |
Bacteroides fragilis |
| 5'… C T A G … 3' 3'… G A T C … 5' | |
“Cuatro cortador”. Hojas 5' voladizo. |
|
Nci I |
Neisseria cinerea |
| C 5'… C C G G G … 3' 3'… G G C C C … 5' G | |
“Cinco cortador”. La base media puede ser citosina o guanina. Hojas 5' voladizo. Diferentes sitios de reconocimiento pueden tener secuencias no complementarias. |
|
Eco R1 |
Escherichia coli |
| 5'… G A A T T C … 3' 3'… C T T A A G … 5' | |
“Seis cortador”. Hojas 5' voladizo. Se comporta como un “cuatro cortador” (actividad 'estrella') en búfer alto en sal. $44 por 10,000 unidades. |
|
Hae II |
Haemophilus aegyptius |
| 5'… Pu G C G C Py … 3' 3'… Py C G C G Pu … 5' | |
“Seis cortador”. Pu es cualquier purina, Py es cualquier pirimidina. Voladizo de las hojas 3'. |
|
ECOO109i |
Escherichia coli |
| 5'… Pu G G N C C Py … 3' 3'… Py C C N G G Pu … 5' | |
“Siete cortador”. Pu es cualquier purina, Py es cualquier pirimidina, N es cualquier base. Hojas 5' voladizo. Diferentes sitios de reconocimiento pueden tener secuencias no complementarias. |
|
Bgl I |
Bacillus globigii |
| 5'… GCCN NNNNGGC … 3' 3'… CGGNNNN NCCG … 5' | |
“Seis cortador con palíndromo interrumpido”. Hojas 5' voladizo. Diferentes sitios de reconocimiento pueden tener secuencias no complementarias. |
|
Bsa HI |
Bacillusstearothermophilus |
| 5'… G Pu C G Py C … 3' 3'… C Py G C Pu G … 5' | |
“Seis cortador”. Diferentes sitios de reconocimiento serán complementarios. |
|
Aat II |
Acetobacter aceti |
| 5'… G A C G T C … 3' 3'… C T G C A G … 5' | |
“Seis cortadoras” con voladizo de 3'. Misma secuencia de reconocimiento que Bsa HI, pero diferente posición de escisión. |
|
Bpm I |
Bacillus pumilus |
| 5'… C T G G A G N16 … 3' 3'… G A C C T C N14 … 5' | |
No palíndromo, escisión distal. Hojas 3' voladizo. $50 por 50 unidades. |
|
No yo |
Nocardia otitidiscaviarum |
| 5'… G C G G C C G C … 3' 3'… C G C C G G C G … 5' | |
“Ocho cortador”. Hojas 5' voladizo. |
|
BSM I |
Bacillusstearothermophilus |
| 5'… G A A T G C N … 3' 3'… C T T A C G N … 5' | |
“Rara”. Voladizo de las hojas 3'. |
- La utilidad de las endonucleasas de restricción radica en su especificidad y la frecuencia con la que sus sitios de reconocimiento ocurren dentro de cualquier muestra de ADN dada.
- Si hay una probabilidad del 25% para una base específica en cualquier sitio dado, entonces la frecuencia con la que se producirán diferentes sitios de endonucleasas de restricción se puede calcular fácilmente (0.25 n):
|
Especificidad |
Ejemplo |
Frecuencia de ocurrencia |
|
Secuencia de cuatro bases |
Alu I |
1 sitio Alu por cada 256 bases (0.25 Kb) |
|
Secuencia de cinco bases |
Nci I |
1 sitio Nci I en cada 1024 bases (1.0 Kb) |
|
Secuencia de seis bases |
EcoR I |
1 sitio EcoR1 por cada 4.096 bases (4.1 Kb) |
|
Secuencia de siete bases |
ECOO109i |
1 sitio EcoO109i por cada 16.384 bases (16.4 Kb) |
|
Secuencia de ocho bases |
No yo |
1 No sitio en cada 65,536 bases (65.5 Kb) |
Así, en promedio, cualquier ADN dado contendrá un sitio Alu I cada 0.25 kilobases, mientras que un sitio Not I ocurre una vez aproximadamente cada 65.5 kilobases.
- Not I es, por lo tanto, una enzima muy útil para aislar grandes regiones de ADN, típicamente en investigaciones que involucran manipulaciones de ADN genómico.
- Se esperaría que Alu I digiriera una muestra de ADN en muchos trocitos pequeños.
El surtido de fragmentos de ADN representaría una "huella" específica del ADN particular que se está digiriendo. Diferentes ADN no producirían la misma colección de tamaños de fragmentos. Así, el ADN de diferentes fuentes puede emparejarse o distinguirse basándose en el ensamblaje de fragmentos después del tratamiento con endonucleasas de restricción. Estos se llaman “polimorfismos de longitud de fragmentos de restricción”, o RFLP. Este simple análisis se utiliza en diversos aspectos de la biología molecular, así como una aplicación de la ley y genealogía. Por ejemplo, las variaciones genéticas que distinguen a los individuos también pueden dar como resultado menos sitios de reconocimiento de endonucleasas de restricción o adicionales.
Electroforesis en gel de ADN
La matriz de soporte sólido de electroforesis en gel más común para moléculas de ADN es
- agarosa y
- acrilamida.
Geles de agarosa de ADN
La tasa de migración electroforética del ADN a través de geles de agarosa depende de cuatro parámetros principales:
1. El tamaño molecular del ADN. Las moléculas de ADN dúplex lineal viajan a través de geles de agarosa a una velocidad que es inversamente proporcional al log de su peso molecular.
M r α 1/log (Mw)
Ejemplo: Comparar la masa molecular frente a la tasa de migración esperada:
|
Masa molecular (Da) |
tronco (Molec. Masa) |
1/tronco (Molec. Masa) |
|
100,000 |
5.0 |
0.20 |
|
50,000 |
4.7 |
0.21 |
|
10,000 |
4.0 |
0.25 |
|
5,000 |
3.7 |
0.27 |
|
1,000 |
3.0 |
0.33 |
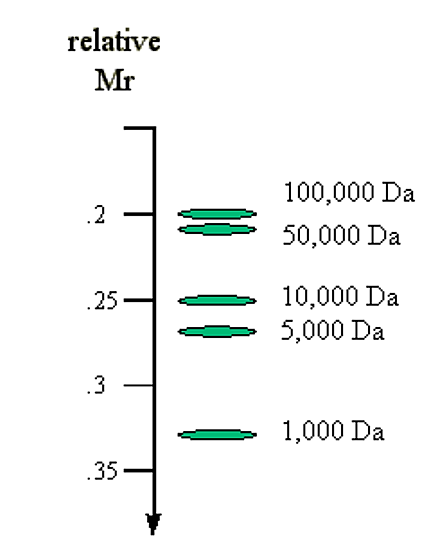.png)
Figura 6.4.4: Masa molecular y tasa de migración
2. La concentración de agarosa. Existe una relación lineal inversa entre el logaritmo de la movilidad electroforética y la concentración de gel.
|
Agarosa (%) |
Rango de separación de ADN lineal (en kilobases) |
|
0.3 |
60 - 5 |
|
0.6 |
20 - 1 |
|
0.7 |
10 - 0.8 |
|
0.9 |
7 - 0.5 |
|
1.2 |
6 - 0.4 |
|
1.5 |
4 - 0.2 |
|
2.0 |
3 - 0.1 |
3. La conformación del ADN.
- ADN circular cerrado (Forma-I) - típicamente superenrollado (compacto)
- circular mellado (Forma-II) - nick relaja cualquier superenrollamiento
- ADN lineal (Forma-III)
Estas diferentes formas del mismo ADN migran a diferentes velocidades a través de un gel de agarosa.
- Casi siempre la forma lineal (forma III) migra a la velocidad más lenta de las tres formas
- El ADN superenrollado (forma I) generalmente migra el más rápido
.png)
Figura 6.4.5: Formas de ADN
4. El voltaje aplicado.
Otros detalles:
- El valor típico para hacer funcionar un gel de agarosa es de 5 voltios por cm (longitud del gel).
- Los geles de agarosa generalmente se vierten y se ejecutan horizontalmente
- Finalmente, siendo el ADN una molécula ácida, migra hacia el electrodo cargado positivamente (cátodo). El ADN naturalmente tiene una relación carga a masa constante, por lo que no es necesario agregar detergentes (como ocurre con las proteínas)
.png)
Figura 6.4.6: Electroforesis en gel
Geles de acrilamida de ADN
- Los geles de acrilamida son útiles para la separación de pequeños fragmentos de ADN
- típicamente oligonucleótidos <100 pares de bases.
- Estos geles suelen ser de baja concentración de acrilamida (<= 6%) y contienen el agente desnaturalizante no iónico Urea (6M).
- El agente desnaturalizante evita la formación de estructuras secundarias en oligonucleótidos y permite una determinación relativamente precisa de la masa molecular.
Tinción de ADN
- El método más conveniente para visualizar el ADN en electroforesis en gel es la tinción con el colorante fluorescente bromuro de etidio.
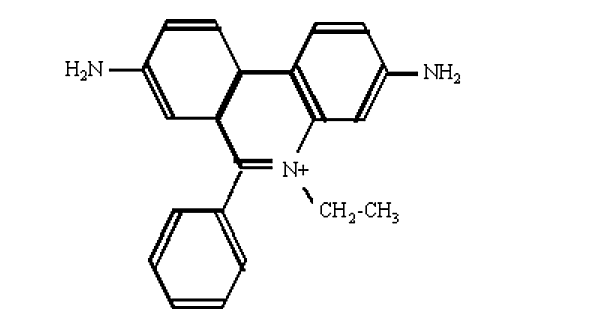.png)
Figura 6.4.7: Bromuro de etidio
- Este compuesto contiene un grupo plano que se intercala entre las bases apiladas del ADN.
- La orientación y proximidad del etidio con las bases apiladas hace que el tinte muestre una mayor luminiscencia en comparación con el tinte libre (en solución).
- La radiación U.V. a 254 nm es absorbida por el ADN y transmitida al colorante unido.
- La energía se reemite a 590 nm en la región rojo-naranja del espectro.
- El bromuro de etidio generalmente se prepara como una solución madre de 10 mg/ml en agua, se almacena a temperatura ambiente y se protege de la luz.
- El colorante generalmente se incorpora al gel y al tampón de funcionamiento, o a la inversa, el gel se tiñe después de correr remojándolo en una solución de bromuro de etidio (0.5 p/ ml durante 30 min).
- La mancha se visualiza irradiando con una fuente de luz UV (es decir, usando un transiluminador) y fotgrafiando con película polaroide.
- La sensibilidad habitual de detección es mejor que 0.1 ug de ADN.
Debido a que el etidio es un agente intercalante de ADN, es un mutágeno potente. La incorporación del etidio en el ADN de los organismos vivos (es decir, usted y yo) puede causar mutaciones (no deseadas).
Combinación de digestión con endonucleasas de restricción con electroforesis en gel de ADN: Mapeo de restricción
Una secuencia dada de ADN (por ejemplo, un gen) tendrá una secuencia específica y, por lo tanto, sitios de endonucleasa de restricción específicos.
- El número y la ubicación de dichos sitios es una propiedad única y predecible para una molécula de ADN dada
- El patrón de fragmentación (es decir, número y tamaño de fragmentos después de la digestión con endonucleasas de restricción) se puede caracterizar por electroforesis en gel como un tipo de “huella digital de ADN”
- Cualquier cambio en la secuencia de ADN de un gen puede resultar en la eliminación de sitios de restricción particulares, y a la inversa, crear otros nuevos:
.png)
Figura 6.4.8: Cambio en los sitios de restricción
- El patrón de fragmentos de electroforesis en gel para el gen de albúmina humana y de perro será característicamente diferente entre sí
- Una muestra de sangre puede identificarse potencialmente como humana o como perro observando el polimorfismo de longitud del fragmento de restricción
- Las diferencias genéticas entre individuos también se pueden identificar mediante análisis de polimorfismo de longitud de fragmentos de restricción
.png)
Figura 6.4.9: Uso de RFLP para ver diferencias genéticas


