8.9: Miniprep de ADN por Lisis Alcalina (Actividad)
- Page ID
- 56247
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Lisis Alcalina
Una vez que el ADN es introducido y transportado en bacterias, nos gustaría aislar el ADN nuevamente para su posterior manipulación. Para ello, las bacterias que contienen el plásmido de interés se cultivan en un cultivo líquido de caldo rico en nutrientes hecho de extracto de levadura llamado Caldo Luria-Bertani (LB). Estas bacterias cultivadas se cultivan hasta que tienen una alta concentración durante la noche. Se cosechan por centrifugación y se retira el caldo. El sedimento resultante de bacterias se resuspende en un tampón fisiológico que contiene el quelante EDTA. Un quelante es un químico que elimina cationes divalentes como Ca 2+ o Mg 2+ de la solución. Esto es significativo porque los cationes divalentes son necesarios para que las enzimas que digieren el ADN sean activas. Al quelar los iones, el ADN que finalmente deseamos purificar estará a salvo de la degradación.
Después de la resuspensión de la bacteria, se mezcla una solución alcalina de NaOH 0.1N en la mezcla bacteriana. Esta solución también contiene un detergente iónico llamado dodecilsulfato de sodio (SDS) que ayuda a desnaturalizar las proteínas e interrumpir sus interacciones con el ADN. La mezcla se vuelve viscosa a medida que las bacterias se abren y su contenido se escapa a la solución. Esta solución básica se neutraliza luego con un tampón de acetato de potasio a pH 5.5. A medida que las soluciones se mezclan, el pH se acerca a 7 y el potasio interactúa con el SDS para provocar la precipitación del ADN cromosómico genómico y las proteínas. Para separar el precipitado de la solución, la mezcla se centrifuga a alta velocidad para sedimentar el ADN genómico y la proteína. El sobrenadante, o solución, se transfiere a una columna que contiene una membrana de sílice. En condiciones de alto contenido de sal, el ADN se adhiere al vidrio o sílice. Al pasar la solución a través de esta columna, el ADN plasmídico en el sobrenadante se atrapa sobre la membrana de sílice y se retira de la solución. Se utilizan lavados adicionales para eliminar los contaminantes dispersos y eliminar el exceso de sal. El ADN plasmídico se elimina finalmente de la columna a través de elución mediante un tampón bajo en sal. Este tampón bajo en sal es Tris pH 8 con EDTA (TE). El ADN plasmídico se puede almacenar de forma estable en el tampón TE en el congelador durante periodos prolongados.
Ejercicio 1: Mini-preparación de ADN plasmídico por lisis alcalina
- Inocular 2 ml de medio rico (LB, YT o Caldo Terrific) que contenga el antibiótico apropiado con una sola colonia de bacterias transformadas. Incubar el cultivo durante la noche a 37°C con agitación vigorosa. (Esto es lo que te proporcionaron)
- Cada grupo debe tomar 2 cultivos
- Centrifugar los tubos de cultivo directamente al máximo durante 5 minutos.
- Si es incapaz de hilar en estos tubos, transfiera 1.5 ml del cultivo a un tubo de microcentrífuga (Eppendorf Tube).
- Centrifugar a velocidad máxima por 30 seg.
- Cuando se complete la centrifugación, vierta la solución de caldo en un recipiente con lejía.
- Resuspender el sedimento bacteriano en 250 μl de solución P1 helada mediante agitación vigorosa y transfiéralo de nuevo a un tubo de microcentrífuga.
- P1 es una solución fisiológica de Tris 50 mM a pH 8.
- P1 contiene un quelante llamado EDTA.
- Los quelantes unen el exceso de cationes divalentes que se requieren para la actividad de la DNasa.
- Lisar: Añadir 250μl de solución P2 a cada suspensión bacteriana. Cierre bien el tubo y mezcle el contenido invirtiendo el tubo suavemente cinco veces. ¡No vórtices! Almacene el tubo en hielo.
- Este es el tampón de lisis que contiene el detergente Dodecil Sulfato de Sodio y NaOH.
- Neutralizar : Añadir 350 μl de solución P3 enfriada con hielo. Cierre el tubo y disperse la solución de lisis invirtiendo el tubo varias veces. Almacene el tubo en hielo durante 3-5 minutos.
- Este es el tampón de neutralización que contiene acetato de potasio.
- La neutralización restaura el pH a cerca de 7 y también causa la precipitación de ADN genómico y proteínas en un desorden gloopy (tipo mocosa).
- Centrifugar el lisado bacteriano a velocidad máxima durante 5 minutos en una microcentrífuga.
- Después de este paso, las sustancias similares a las manchas deben empaquetarse firmemente en un pellet en la parte inferior del tubo.
- La solución o sobrenadante contiene el ADN plasmídico.
- Purificación en columna de ADN: Transferir el sobrenadante a un tubo fresco con una columna de membrana de sílice
- Al ADN le gusta unirse al vidrio en condiciones de alto contenido de sal.
- La membrana blanca está hecha de fibra de vidrio.

- Centrifugar el sobrenadante a través de la columna durante 1 minuto a velocidad máxima en una microcentrífuga.
- El ADN se unirá a la membrana de la columna (sílice)
- Lavado: Deseche el flujo continuo y vuelva a colocar la columna en el tubo de desechos. Lavar la columna con 500 μl de PE. Centrifugar el sobrenadante a través de la columna durante 1 minuto como máximo en una microcentrífuga.
- El PE es una solución que ayuda a eliminar las sustancias no específicamente unidas
- Deseche el flujo pasante y vuelva a colocar la columna en el tubo de desechos. Lavar la Columna con 700 μl PE. Centrifugar el sobrenadante a través de la columna durante 1 minuto como máximo en una microcentrífuga. Deseche el flujo continuo y repita el centrifugado para secar la columna.
- Colocar la columna en un tubo de centrífuga fresco y Eluir los ácidos nucleicos en 50 μl de TE (pH 8.0) uniéndose por 1 minuto y centrifugando a velocidad máxima durante 1 minuto.
Identificación de ADN plasmídico
Una vez aislados los plásmidos, requieren identificación. Los vectores plasmídicos tienen secuencias conocidas y se mapean de sus características principales. Conocer la secuencia de estas piezas de ADN significa conocer las ubicaciones de los sitios de digestión de RE. Mediante el uso de Res, la digestión de plásmidos en tamaños conocidos ayuda en la verificación de la identidad del plásmido sin la necesidad de tener el plásmido entero re-secuenciado. Un plásmido común se llama pUC18 o pUC19. La “p” significa plásmido, la “UC” significa Universidad de California (donde se diseñó) y 18 o 19 se refieren a la diferencia en el MCS. Este plásmido tiene 2.686 pares de bases o ~2.7kb (kilobase) de largo con un único sitio EcoRI en el MCS. Otro plásmido de interés en el aprendizaje de Biología Molecular se llama pGlo. Este plásmido tiene un gen de medusa en el MCS que codifica para una proteína que fluorescerá verde cuando se expresa bajo luz UV. pGlo tiene 5.4kb de largo y contiene un único sitio EcoRI. Una vez digeridos, por una enzima, estos plásmidos pueden identificarse en base a la separación por tamaños en un gel de agarosa. Por lo general, lo mejor es identificar mediante el uso de 2 RE diferentes. La digestión es importante antes de las comparaciones de tamaño ya que el ADN circular migra a través de agarosa de manera diferente al ADN Adicionalmente, el ADN circular a veces puede ser “súper enrollado” y conducir a una migración muy rápida a pesar del tamaño.

Mapa de características de pUC19 incluyendo la ubicación de algunas enzimas de restricción.

Vista de cerca del Sitio de Clonación Múltiple (MCS) pUC19. Estos sitios de restricción aparecen solo una vez a lo largo de toda la secuencia plasmídica.
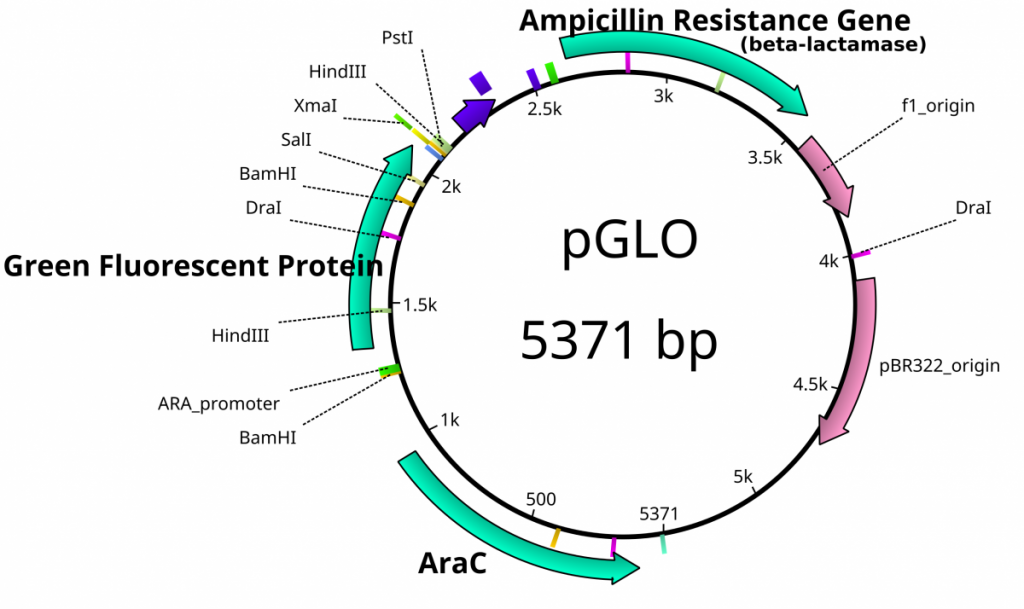
Mapa de características plasmídicas de pGlo. Esto no es un vector de clonación, por lo que no existe un MCS.
Ejercicio 2: Identificación de plásmidos por digestión por restricción
- La clase debe preparar geles de agarosa 2X 0.8% mediante la preparación de agarosa 0.4g en tampón TBE 50ml.
- Fundir solución de agarosa por microondas durante 1 minuto.
- Agregue 5μl de solución Sybr Safe en la solución de gel de 100 ml.
- Vierte esta solución en una bandeja de fundición dentro del refrigerador.
- Inserte un peine.
- A un nuevo tubo agregar 2μl de ADN plasmídico a 8μl de mezcla de digestión rápida Eco RI.
- Buffer de digestión rápida de 1 μl
- Enzima Eco RI de digestión rápida de 1μl
- 6μl H 2 O
- Incubar a 37°C por 10 minutos.
- Agregar 2μl de tampón de carga a la mezcla de digestión.
- En un tubo separado, combine 3μl de ADN plasmídico con 2μl de tampón de carga y 7μl de H 2 O.
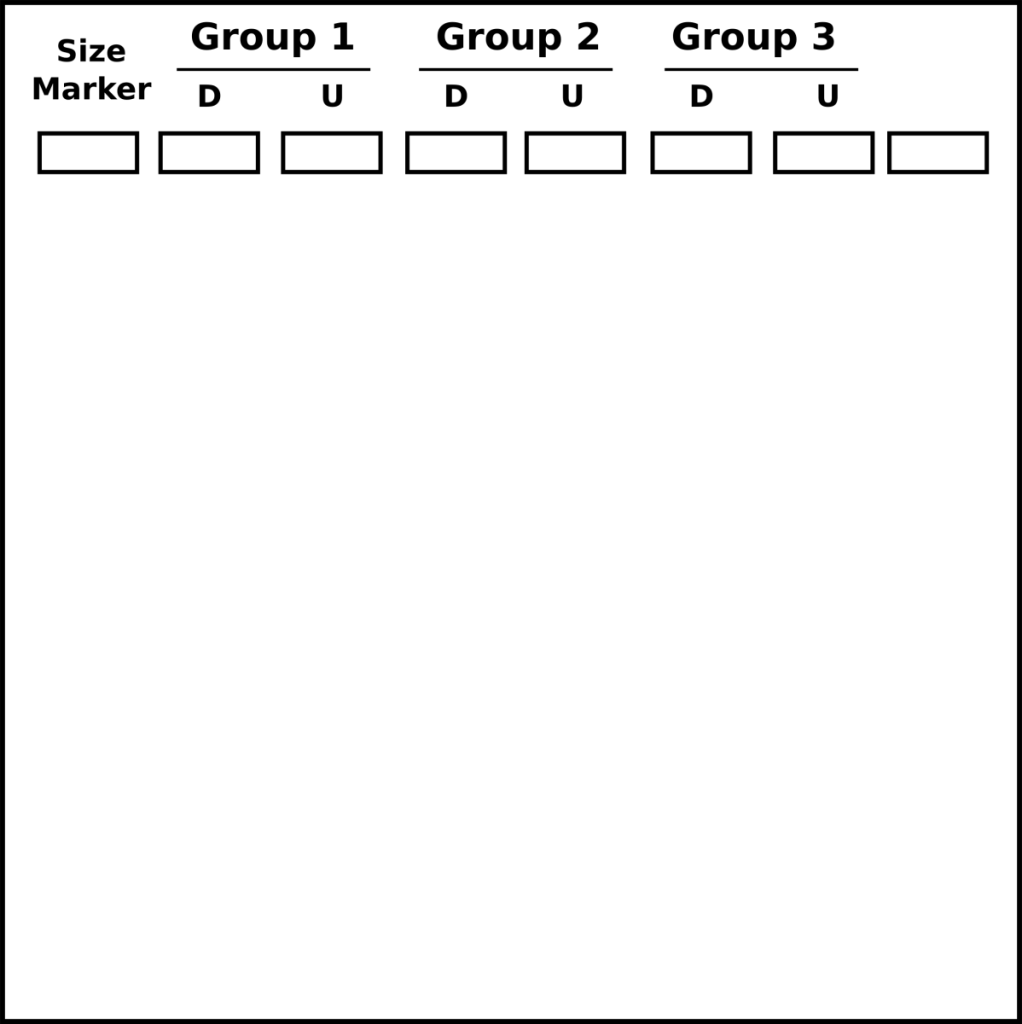
- Cargar gel con una escala de tamaño apropiado en el primer carril, cargar el plásmido digerido en los carriles siguientes, luego el plásmido no digerido.
- 3 grupos pueden cargarse en un gel
- D = plásmido digerido
- U = plásmido no digerido
- Ejecutar el gel a 110V durante 30 minutos y visualizar en un transiluminador UV.
- Documente con su cámara.
Recursos adicionales
- Para obtener ayuda sobre este problema, pruebe la actividad de digestión in silico.


