8.10: Secuenciación de ADN de Sanger
- Page ID
- 56248
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Terminación de cadena radiactiva
La polimerización de ácidos nucleicos ocurre en una dirección 5' → 3'. La posición 5' tiene un grupo fosfato mientras que la posición 3' de la hexosa tiene un grupo hidroxilo. La polimerización depende de estos 2 grupos funcionales para que se produzca una reacción de síntesis de deshidratación y extender la cadena principal de azúcar-fosfato del ácido nucleico. En la década de 1970, el grupo de Fred Sanger descubrió un método fundamentalmente nuevo de 'leer' la secuencia lineal de ADN usando bases especiales llamadas terminadores de cadena o didesoxinnucleótidos. La ausencia de un grupo hidroxilo en la posición 3' bloquea la polimerización, dando como resultado la terminación. Este método todavía está en uso hoy en día y se llama “método de terminación de cadena de didesoxinucleótidos de Sanger”. Este método originalmente utilizó un cebador marcado radiactivamente para iniciar la reacción de secuenciación. Se producen cuatro reacciones donde cada reacción es intencionalmente “envenenada” con un terminador de cadena didesoxilo. Por ejemplo, una reacción tendrá los 4 dNTPs (desoxinucleótidos trifosfatos) con la adición a una pequeña cantidad de ddATP (didesoxiadenosina trifosfato). Esta reacción dará como resultado una serie de terminaciones prematuras de la polimerización específicamente en diferentes lugares donde se incorporaría una Adenina.

dATP es un monómero natural utilizado en la polimerización del ADN. El 3'-OH es el punto de unión del siguiente nucleótido posterior.
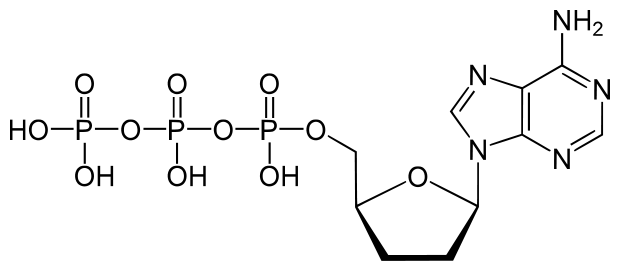
La falta de un 3′-OH en esta molécula de ddATP la convierte en un terminador de cadena que prohibirá la adición de otro nucleótido al polímero de ADN.
El producto de estas 4 reacciones de secuenciación separadas se ejecuta en un gran gel de secuenciación de poliacrilamida. Los fragmentos más pequeños corren a través del gel más rápido y crean un patrón similar a una escalera. Esto se puede visualizar mediante el uso de una película de rayos X que sea sensible a la radiactividad. Cada carril del gel corresponde a una de las cuatro reacciones de terminación de cadena. Las bases se leen secuencialmente de abajo hacia arriba y revelan la secuencia del ADN.
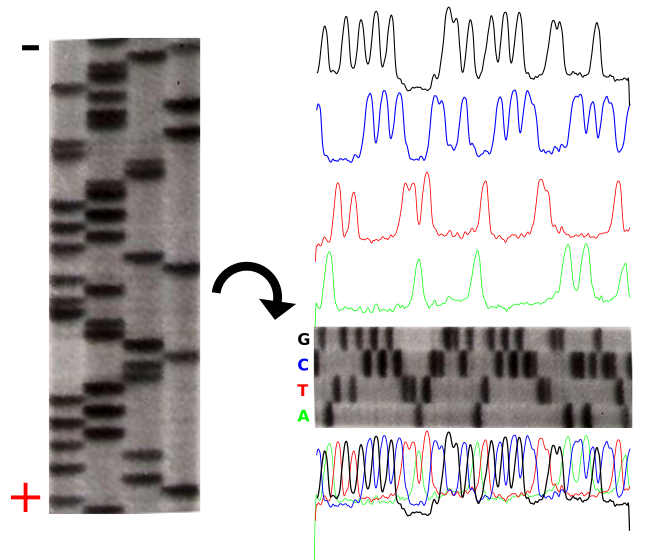
El gel de secuenciación se puede puntuar manualmente. Los perfiles de cada carril se han creado utilizando ImageJ para ilustrar el patrón de bandas y la secuencia posterior.
Crédito: John Schmidt y Jeremy Seto (CC-BY-SA 3.0)
Terminación de cadena fluorescente y electroforesis capilar
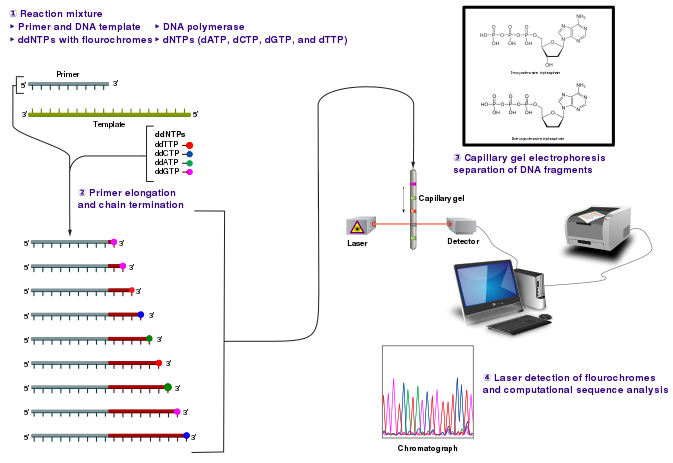
Crédito: Estevezj (CC-BY-SA 3.0)
La radiactividad es peligrosa e indeseable para trabajar con lo que se desarrollaron terminadores de cadena con etiquetas fluorescentes. Este método sintetiza una serie de cadenas de ADN que son específicamente fluorescentes en la terminación que se pasa a través de un sistema de electroforesis capilar. A medida que los fragmentos de ADN pasan por un láser y detector, se identifica la diferente señal fluorescente atribuida a cada ddNTP y genera un cromatograma para representar la secuencia. Los Terminadores de Cadena Fluorescentes ahora se utilizan en reacciones y se ejecutan a través de un pequeño capilar. Los fragmentos más pequeños pasan primero y se detectan para revelar un cromatograma.
Se utilizan cromatogramas fluorescentes para puntuar la terminación de la cadena de nucleótidos. La amplitud de cada pico corresponde a la fuerza o certeza de la llamada nucleotídica. Los archivos de cromatograma generalmente se proporcionan junto con el archivo de secuencia con la extensión*.ab1 mientras que los archivos de secuencia se proporcionan como un archivo de texto en el formato fasta. Puede encontrar más información sobre estos archivos aquí. Los archivos ab1 son extremadamente importantes para analizar cuando hay ambigüedad o errores de secuenciación. Estos archivos ab1 también se pueden utilizar para atribuir una puntuación de calidad en la llamada base.
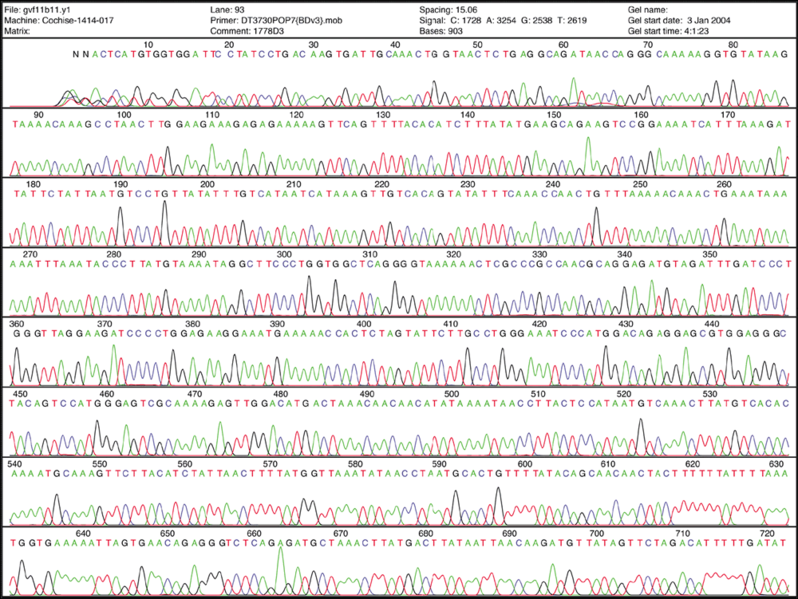
Cuando hay demasiada ambigüedad en la señal debido a múltiples picos, a menudo encontrarás una N en lugar de uno de los 4 nucleótidos (A, T, C y G).
Este video (fuente: www.yourgenome.org CC-BY) ilustra el mecanismo de terminación de cadena fluorescente y electroforesis capilar.
Genomas de Secuenciación
Crédito: Jeremy Seto (CC-BY-NC-SA 3.0)
La secuenciación tradicional de genomas fue un proceso largo y tedioso que clonó fragmentos de ADN genómico en plásmidos para generar una biblioteca de ADN genómico (ADNg). Estos plásmidos se secuenciaron individualmente utilizando la metodología de secuenciación de Sanger y se realizó computacional para identificar piezas superpuestas, como un rompecabezas. Este montaje resultaría en un andamio de tiro.
El siguiente video está tomado de yourgenome.org (CC-BY) e ilustra la secuenciación del genoma humano a través del enfoque de secuenciación de escopeta.


