20: Regulación transcripcional mediante alteraciones de la cromatina
- Page ID
- 58742
{{Template.dropdown {path:” /Genetics/ "}}}
REGULACIÓN POR CAMBIOS EN LA ESTRUCTURA DE LA CROMATINA
Revisión de la estructura del nucleosoma y la cromatina
Composición del nucleosoma
- Los nucleosomas son la subunidad repetitiva de la cromatina.
- Los nucleosomas están compuestos por un núcleo nucleosoma, histona H1 (en eucariotas superiores) y ADN enlazador de longitud variable (0-50 pb).
- El núcleo del nucleosoma contiene un octámero de 2 cada una de las histonas centrales (H2A, H2B, H3 y H4) y 146 pb de ADN envuelto 1.75 vueltas (Figura 4.6.1).
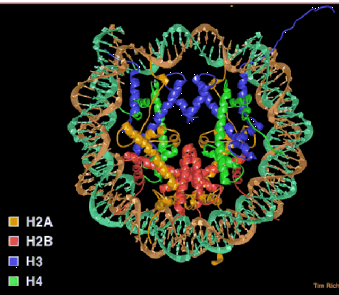
Interacciones de histonas en el nucleosoma
- Las histonas centrales se dimerizan a través de sus motivos de pliegue de histonas generando dímeros H3/H4 y dímeros H2A H2B (Figura 4.6.2.).
- Dos dímeros H3/H4 se asocian para formar un tetrámero, que se une al ADN.
- Dos dímeros H2A/H2B se asocian con el tetrámero para formar el octámero de histona.
- En la sal fisiológica el octámero no es estable a menos que esté unido al ADN y se disocie en el tetrámero H3/H4 y dos dímeros H2A/H2B.
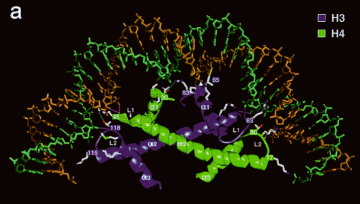
Estructura de orden superior de la cromatina
- Las matrices de nucleosomas se condensan en fibras de cromatina de orden superior (Figura 4.6.3.).
- A pesar de más de 2 décadas de investigación, se desconoce la estructura de la fibra de cromatina “30nm”.
- Esto puede deberse a irregularidad o inestabilidad de la estructura.
- Este nivel de estructura ha sido implicado en los mecanismos de represión de la cromatina, por lo que la falta de información estructural en este nivel es particularmente problemática

Investigación bioquímica de diferentes estados de cromatina y actividad génica en células
Sensibilidad de la cromatina a las nucleasas
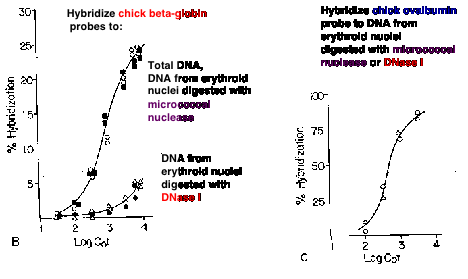
Secuencia propuesta para activación génica
1. Abrir un dominio de cromatina
Reubicar lejos de la heterocromatina pericentromérica
Establecer una configuración de cromatina abierta en todo el locus
Hiperacetilación general de histonas
Sensibilidad a la DNasa I
2. Activar transcripción
Hiperacetilación local de histona H3
Activación del promotor para iniciar y alargar la transcripción
Resumen de elementos cis-reguladores que actúan en la cromatina
Generar una estructura de cromatina abierta y accesible
Puede extenderse sobre cientos de kb
Puede ser específico del tejido
Potenciar la expresión de genes individuales
Puede ser específico del tejido
Puede funcionar en etapas específicas de desarrollo.
Aísle genes de los efectos de posición.
Ensayo de bloqueo del potenciador
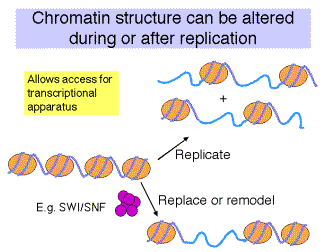
¿Cómo se modifica la estructura de la cromatina en las células para cambiar la actividad transcripcional?
Modelos de competencia vs. reemplazo de cómo los factores de transcripción ocupan sus sitios de unión en una plantilla de cromatina.
Los complejos HAT podrían estar involucrados en otros procesos, o pueden afectarlos indirectamente a través de sus efectos sobre la transcripción. Por ejemplo, un componente del complejo SAGA HAT es Tra1, el homólogo de levadura de una proteína humana involucrada en la transformación celular. Puede ser una diana directa de proteínas activadoras.
Se encuentran múltiples HAT nucleares en levaduras y en otras especies (Cuadro 4.6.2). Todos son grandes con muchas subunidades. En comparación, su sustrato, que es el nucleosoma, es de 0.2 MDa en masa. Tienen diferentes especificidades de sustrato. Algunos acetilados H3 preferencialmente, otros acetilan H4. La razón de la diversidad de HAT es una cuestión de estudio actual.
Cuadro 4.6.2. Los cuatro principales complejos nucleares HAT en levaduras
| Complejo | Masa (MDA=Megadaltons) | |
| SAGA | 1.8 | |
| NuA4 | 1.4 | |
| ADA | 0.8 | |
| NuA3 | 0.5 |
Colaboradores y Atribuciones
- {{template.contribHardison ()}


