7.4: Proteínas
- Page ID
- 54790
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Describir la estructura fundamental de un aminoácido
- Describir las estructuras químicas de las proteínas
- Resumir las características únicas de las proteínas
Al inicio de este capítulo, se describió un famoso experimento en el que los científicos sintetizaban aminoácidos bajo condiciones simulando los presentes en la tierra mucho antes de la evolución de la vida tal como la conocemos. Estos compuestos son capaces de unirse en esencialmente cualquier número, produciendo moléculas de esencialmente cualquier tamaño que poseen una amplia gama de propiedades físicas y químicas y realizan numerosas funciones vitales para todos los organismos. Las moléculas derivadas de aminoácidos pueden funcionar como componentes estructurales de células y entidades subcelulares, como fuentes de nutrientes, como reservorios de almacenamiento de átomos y energía, y como especies funcionales como hormonas, enzimas, receptores y moléculas de transporte.
Aminoácidos y enlaces peptídicos
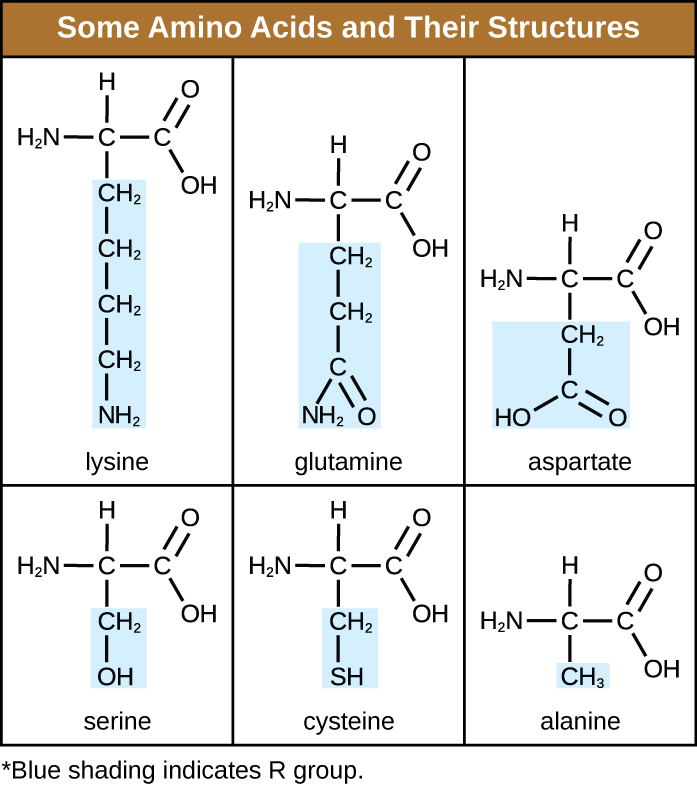
Un aminoácido es una molécula orgánica en la que un átomo de hidrógeno, un grupo carboxilo (—COOH) y un grupo amino (—NH 2) están todos unidos al mismo átomo de carbono, el llamado carbono α. El cuarto grupo unido al carbono α varía entre los diferentes aminoácidos y se denomina residuo o cadena lateral, representado en fórmulas estructurales por la letra R. Un residuo es un monómero que resulta cuando dos o más aminoácidos se combinan y eliminan moléculas de agua. La estructura primaria de una proteína, una cadena peptídica, está compuesta por residuos de aminoácidos. Las características únicas de los grupos funcionales y los grupos R permiten que estos componentes de los aminoácidos formen enlaces hidrógeno, iónicos y disulfuro, junto con interacciones polar/no polares necesarias para formar estructuras proteicas secundarias, terciarias y cuaternarias. Estos grupos están compuestos principalmente por carbono, hidrógeno, oxígeno, nitrógeno y azufre, en forma de hidrocarburos, ácidos, amidas, alcoholes y aminas. Algunos ejemplos que ilustran estas posibilidades se proporcionan en la Figura\(\PageIndex{1}\).
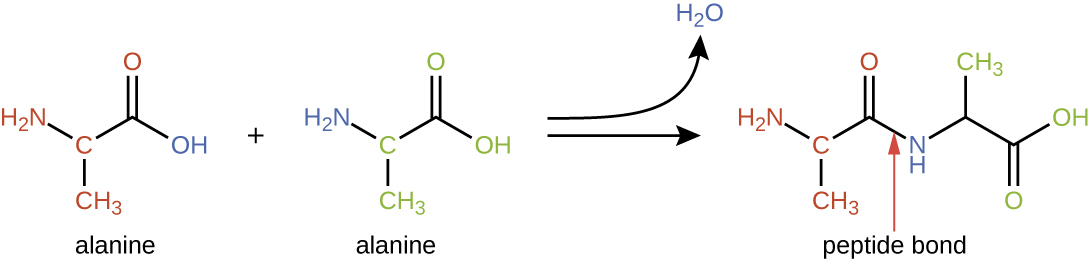
Los aminoácidos pueden unirse químicamente por reacción del grupo ácido carboxílico de una molécula con el grupo amina de otra. Esta reacción forma un enlace peptídico y una molécula de agua y es otro ejemplo de síntesis por deshidratación (Figura\(\PageIndex{2}\)). Las moléculas formadas por la unión química de números relativamente modestos de aminoácidos (aproximadamente 50 o menos) se denominan péptidos, y a menudo se usan prefijos para especificar estos números: dipéptidos (dos aminoácidos), tripéptidos (tres aminoácidos), etc. De manera más general, se designa el número aproximado de aminoácidos: los oligopéptidos se forman uniendo hasta aproximadamente 20 aminoácidos, mientras que los polipéptidos se sintetizan a partir de hasta aproximadamente 50 aminoácidos. Cuando el número de aminoácidos unidos entre sí se vuelve muy grande, o cuando se utilizan múltiples polipéptidos como subunidades constructoras, las macromoléculas que resultan se denominan proteínas. La longitud continuamente variable (el número de monómeros) de estos biopolímeros, junto con la variedad de posibles grupos R en cada aminoácido, permite una diversidad casi ilimitada en los tipos de proteínas que se pueden formar.
Ejercicio\(\PageIndex{1}\)
¿Cuántos aminoácidos hay en los polipéptidos?
Estructura de Proteínas
El tamaño (longitud) y la secuencia específica de aminoácidos de una proteína son determinantes principales de su forma, y la forma de una proteína es crítica para su función. Por ejemplo, en el proceso de fijación biológica de nitrógeno (ver Ciclos Biogeoquímicos), los microorganismos del suelo conocidos colectivamente como rizobios interactúan simbióticamente con raíces de plantas leguminosas como la soja, el maní o el frijol para formar una estructura novedosa llamada nódulo en las raíces de las plantas. La planta produce entonces una proteína portadora llamada leghemoglobina, una proteína que transporta nitrógeno u oxígeno. La leghemoglobina se une con una afinidad muy alta al oxígeno de su sustrato en una región específica de la proteína donde la forma y la secuencia de aminoácidos son apropiadas (el sitio activo). Si la forma o el ambiente químico del sitio activo se altera, incluso ligeramente, el sustrato puede no ser capaz de unirse con tanta fuerza, o puede que no se una en absoluto. Así, para que la proteína esté completamente activa, debe tener la forma adecuada para su función.
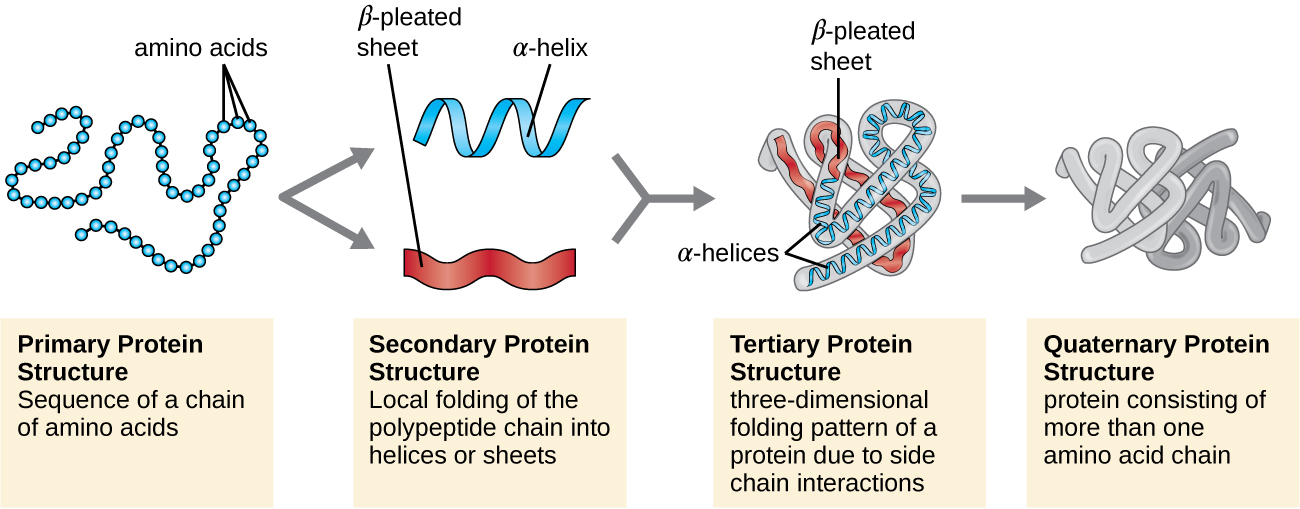
La estructura proteica se clasifica en términos de cuatro niveles: primario, secundario, terciario y cuaternario. La estructura primaria es simplemente la secuencia de aminoácidos que componen la cadena polipeptídica. La Figura\(\PageIndex{3}\) representa la estructura primaria de una proteína.
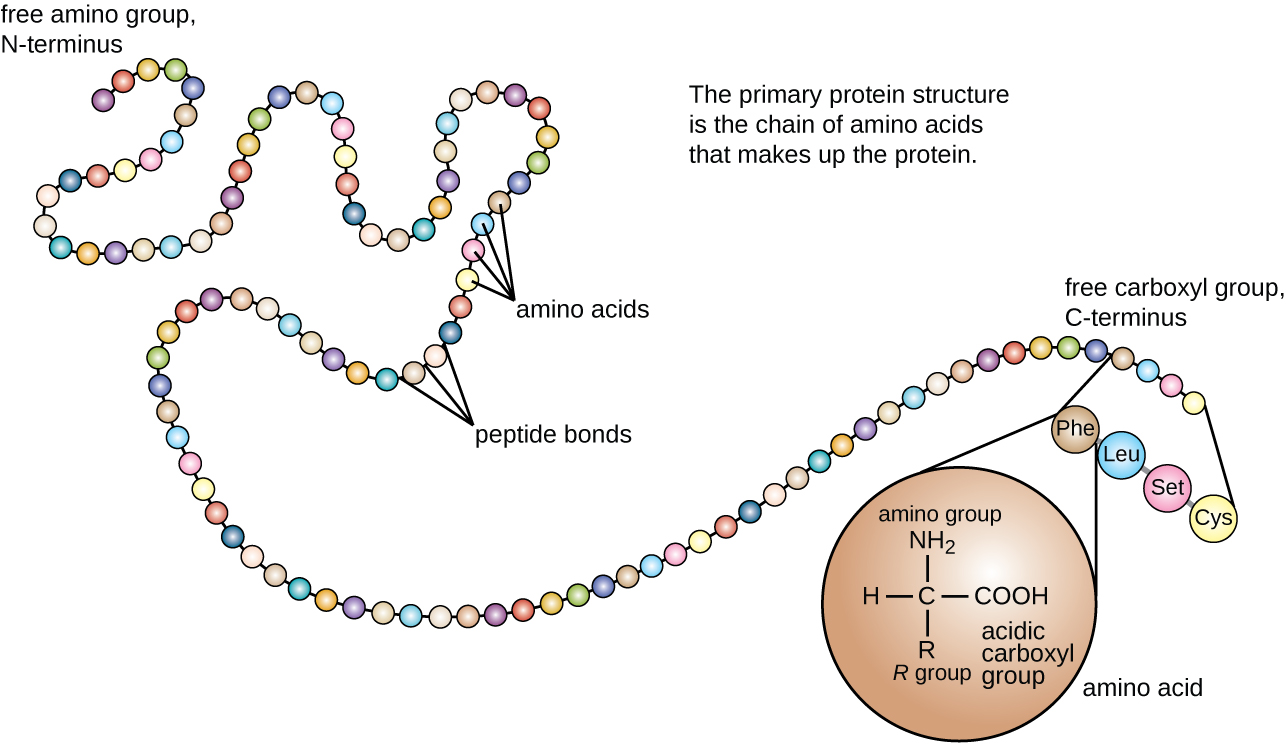
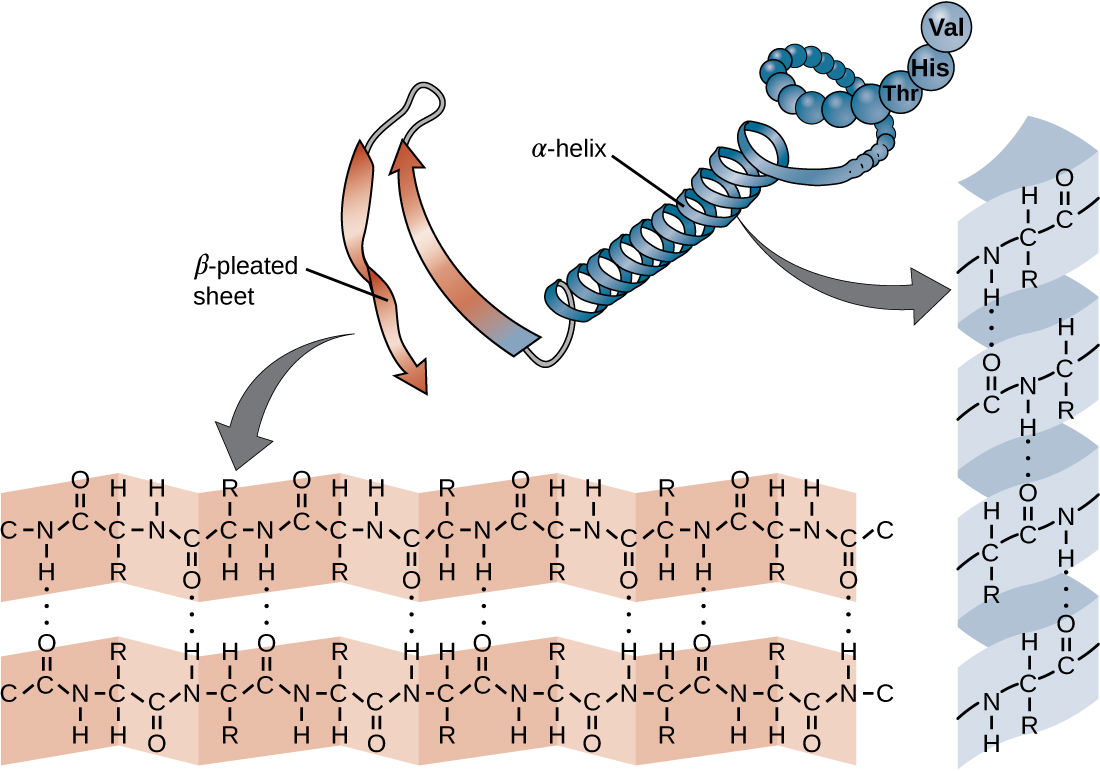
La cadena de aminoácidos que define la estructura primaria de una proteína no es rígida, sino que es flexible debido a la naturaleza de los enlaces que mantienen unidos los aminoácidos. Cuando la cadena es suficientemente larga, pueden producirse enlaces de hidrógeno entre los grupos funcionales amina y carbonilo dentro de la cadena principal peptídica (excluyendo el grupo lateral R), dando como resultado el plegamiento localizado de la cadena polipeptídica en hélices y láminas. Estas formas constituyen la estructura secundaria de una proteína. Las estructuras secundarias más comunes son la hélice α y la lámina plisada β. En la estructura de hélice α, la hélice está sostenida por enlaces de hidrógeno entre el átomo de oxígeno en un grupo carbonilo de un aminoácido y el átomo de hidrógeno del grupo amino que está a solo cuatro unidades de aminoácidos más lejos a lo largo de la cadena. En la lámina plisada β, los pliegues están formados por enlaces de hidrógeno similares entre secuencias continuas de grupos carbonilo y amino que se separan aún más en la cadena principal de la cadena polipeptídica (Figura\(\PageIndex{4}\)).
El siguiente nivel de organización proteica es la estructura terciaria, que es la forma tridimensional a gran escala de una sola cadena polipeptídica. La estructura terciaria está determinada por interacciones entre residuos de aminoácidos que están muy separados en la cadena. Una variedad de interacciones dan lugar a la estructura terciaria de la proteína, como puentes disulfuro, que son enlaces entre los grupos funcionales sulfhidrilo (-SH) en los grupos laterales de aminoácidos; enlaces de hidrógeno; enlaces iónicos; e interacciones hidrófobas entre cadenas laterales no polares. Todas estas interacciones, débiles y fuertes, se combinan para determinar la forma tridimensional final de la proteína y su función (Figura\(\PageIndex{5}\)).
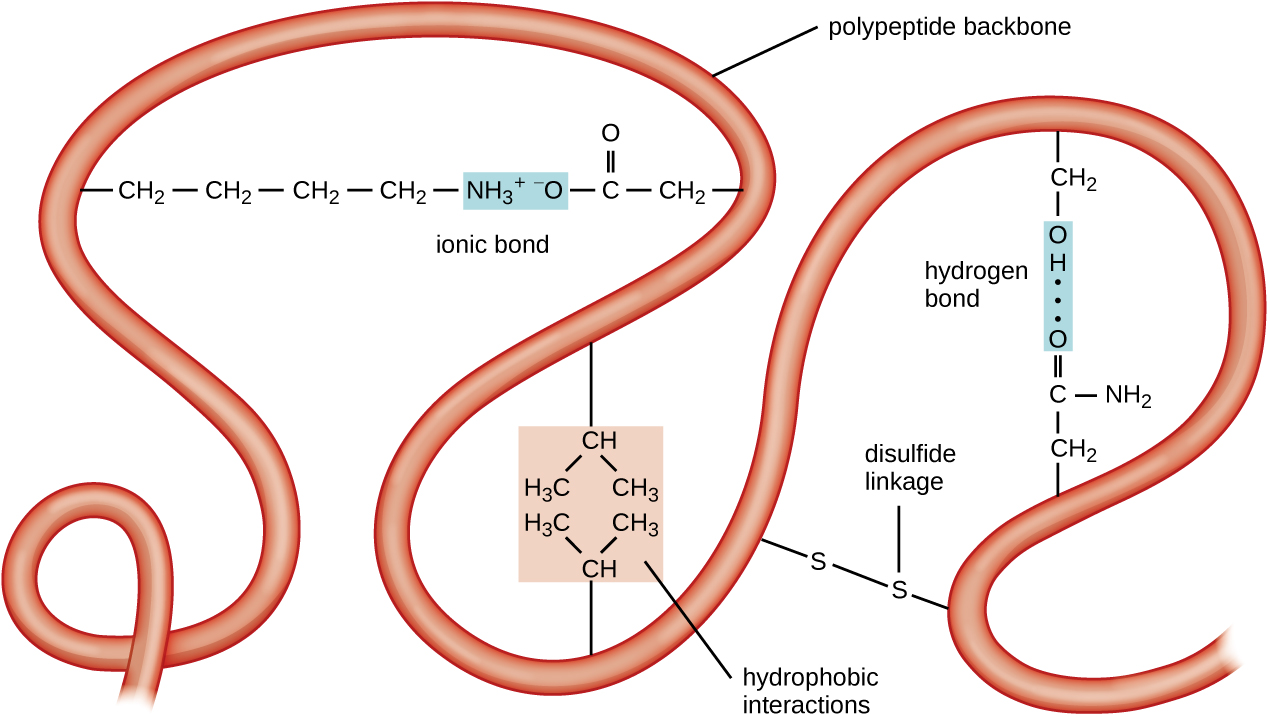
El proceso por el cual una cadena polipeptídica asume una forma tridimensional a gran escala se denomina plegamiento de proteínas. Se dice que las proteínas plegadas que son completamente funcionales en su papel biológico normal poseen una estructura nativa. Cuando una proteína pierde su forma tridimensional, puede que ya no sea funcional. Estas proteínas desplegadas están desnaturalizadas. La desnaturalización implica la pérdida de la estructura secundaria y la estructura terciaria (y, si está presente, la estructura cuaternaria) sin la pérdida de la estructura primaria.
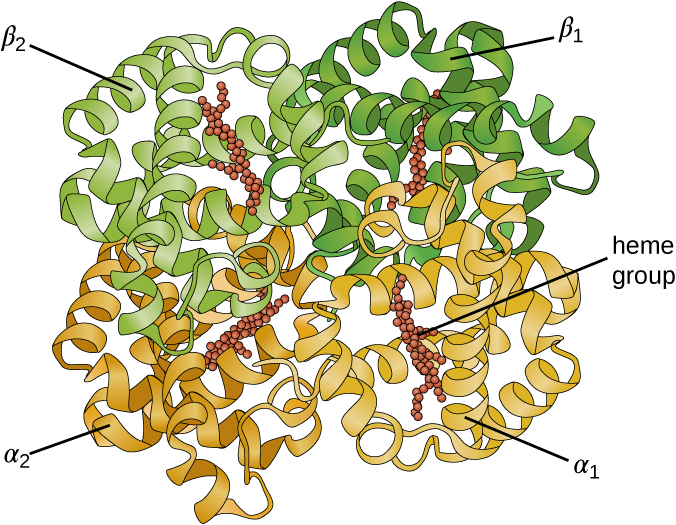
Algunas proteínas son ensamblajes de varios polipéptidos separados, también conocidos como subunidades proteicas. Estas proteínas funcionan adecuadamente solo cuando todas las subunidades están presentes y configuradas adecuadamente. Las interacciones que mantienen juntas estas subunidades constituyen la estructura cuaternaria de la proteína. La estructura cuaternaria general se estabiliza por interacciones relativamente débiles. La hemoglobina, por ejemplo, tiene una estructura cuaternaria de cuatro subunidades proteicas globulares: dos polipéptidos α y dos β, cada uno conteniendo un hemo a base de hierro (Figura\(\PageIndex{6}\)).
Otra clase importante de proteínas son las proteínas conjugadas que tienen una porción no proteica. Si la proteína conjugada tiene un carbohidrato unido, se llama glicoproteína. Si tiene un lípido adherido, se le llama lipoproteína. Estas proteínas son componentes importantes de las membranas. La figura\(\PageIndex{7}\) resume los cuatro niveles de estructura proteica.
Ejercicio\(\PageIndex{2}\)
¿Qué puede pasar si se cambia la estructura primaria, secundaria, terciaria o cuaternaria de una proteína?
Estructura Primaria, Proteínas Disfuncionales y Fibrosis Quística
Las proteínas asociadas a membranas biológicas se clasifican como extrínsecas o intrínsecas. Las proteínas extrínsecas, también llamadas proteínas periféricas, se asocian libremente con un lado de la membrana. Las proteínas intrínsecas, o proteínas integrales, están incrustadas en la membrana y a menudo funcionan como parte de los sistemas de transporte como proteínas transmembrana. La fibrosis quística (FQ) es un trastorno genético humano causado por un cambio en la proteína transmembrana. Afecta principalmente a los pulmones pero también puede afectar el páncreas, el hígado, los riñones y el intestino. La FQ es causada por una pérdida del aminoácido fenilalanina en una proteína transmembrana de fibrosis quística (CFTR). La pérdida de un aminoácido cambia la estructura primaria de una proteína que normalmente ayuda a transportar sal y agua dentro y fuera de las células (Figura\(\PageIndex{8}\)).
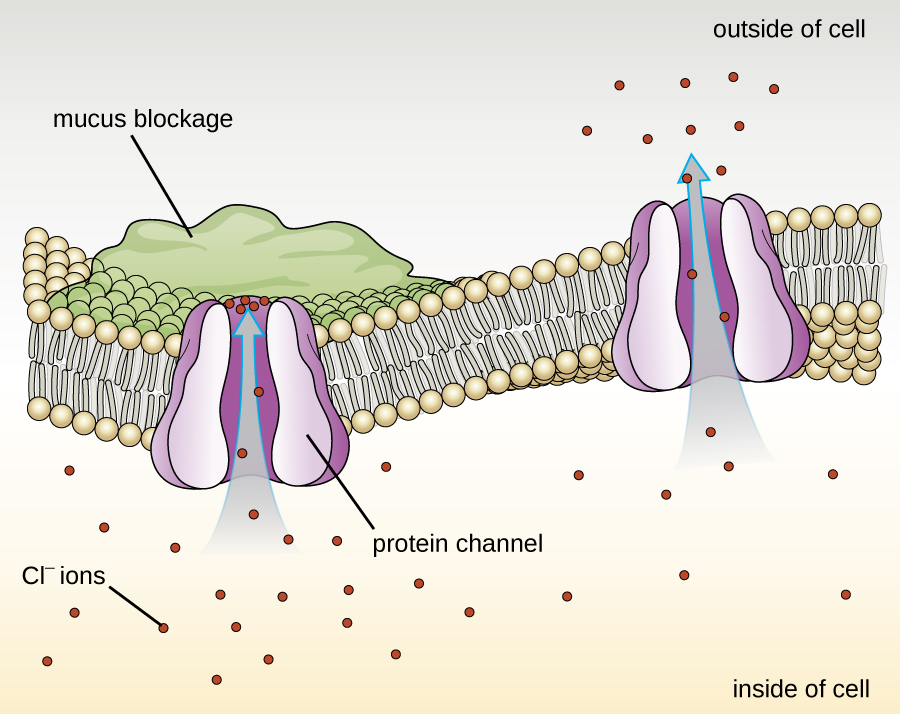
El cambio en la estructura primaria impide que la proteína funcione correctamente, lo que hace que el cuerpo produzca moco inusualmente espeso que obstruye los pulmones y conduce a la acumulación de moco pegajoso. El moco obstruye el páncreas y evita que las enzimas naturales ayuden al cuerpo a descomponer los alimentos y absorber nutrientes vitales.
En los pulmones de individuos con fibrosis quística, el moco alterado proporciona un ambiente donde las bacterias pueden prosperar. Esta colonización conduce a la formación de biopelículas en las pequeñas vías respiratorias de los pulmones. Los patógenos más comunes encontrados en los pulmones de los pacientes con fibrosis quística son Pseudomonas aeruginosa (Figura\(\PageIndex{9}\)) y Burkholderia cepacia. Pseudomonas se diferencia dentro de la biopelícula en el pulmón y forma colonias grandes, llamadas Pseudomonas “mucoides”. Las colonias tienen una pigmentación única que se manifiesta en pruebas de laboratorio (Figura\(\PageIndex{9}\)) y proporciona a los médicos la primera pista de que el paciente tiene FQ (tales colonias son raras en individuos sanos).
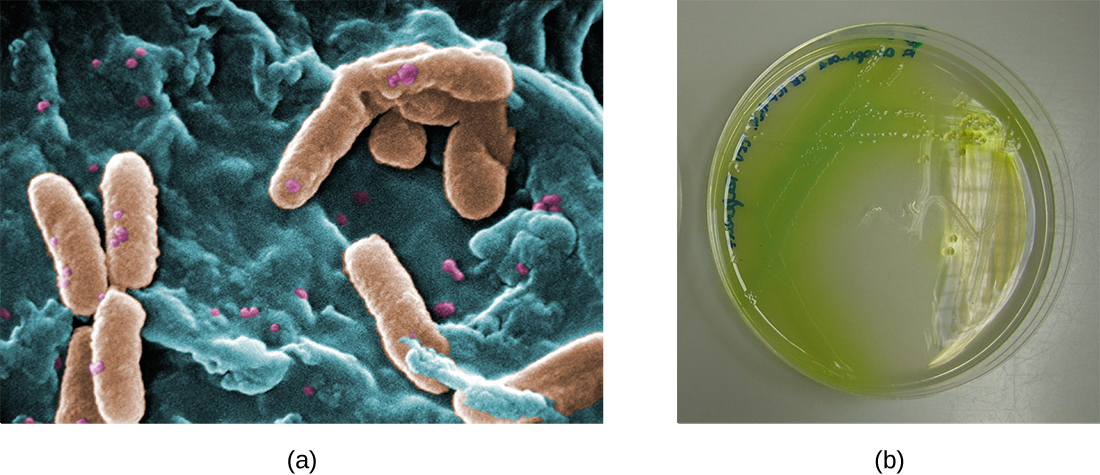
Para más información sobre la fibrosis quística, visite el sitio web de la Fundación de Fibrosis
Conceptos clave y resumen
- Los aminoácidos son pequeñas moléculas esenciales para toda la vida. Cada uno tiene un carbono α al que están unidos un átomo de hidrógeno, un grupo carboxilo y un grupo amina. El cuarto grupo unido, representado por R, varía en composición química, tamaño, polaridad y carga entre diferentes aminoácidos, proporcionando variación en las propiedades.
- Los péptidos son polímeros formados por la unión de aminoácidos a través de la síntesis por deshidratación. Los enlaces entre los aminoácidos enlazados se denominan enlaces peptídicos. El número de aminoácidos unidos entre sí puede variar de unos pocos a muchos.
- Las proteínas son polímeros formados por la unión de un número muy grande de aminoácidos. Realizan muchas funciones importantes en una célula, sirviendo como nutrientes y enzimas; moléculas de almacenamiento de carbono, nitrógeno y energía; y componentes estructurales.
- La estructura de una proteína es un determinante crítico de su función y se describe mediante una clasificación graduada: primaria, secundaria, terciaria y cuaternaria. La estructura nativa de una proteína puede verse alterada por la desnaturalización, resultando en la pérdida de su estructura de orden superior y su función biológica.
- Algunas proteínas están formadas por varias subunidades proteicas separadas, la interacción de estas subunidades componen la estructura cuaternaria del complejo proteico.
- Las proteínas conjugadas tienen una porción no polipeptídica que puede ser un carbohidrato (formando una glicoproteína) o una fracción lipídica (formando una lipoproteína). Estas proteínas son componentes importantes de las membranas.


