3.9: Respuestas a ejercicios
- Page ID
- 150025
Respuestas a la pregunta de coloración de barplot:
Código\(\PageIndex{1}\) (R):
o
Código\(\PageIndex{2}\) (R):
(Por favor, pruebe estos comandos usted mismo. La segunda respuesta es preferible porque funcionará incluso en los casos en que el factor tenga más de dos niveles.)
Respuestas a la pregunta de recuentos de barplot. Para ver frecuencias, de mayor a menor, ejecute:
Código\(\PageIndex{3}\) (R):
o
Código\(\PageIndex{4}\) (R):
Respuesta a la pregunta de las cabezas florecientes. Primero, necesitamos cargar el archivo en R. Con url.show () o simplemente examinando el archivo en la ventana del navegador, revelamos que el archivo tiene múltiples columnas divididas con espacios amplios (probablemente símbolos Tab) y que la primera columna contiene nombres de especies con espacios. Por lo tanto, el encabezado y el separador deben definirse explícitamente:
Código\(\PageIndex{5}\) (R):
El siguiente paso es siempre verificar la estructura del nuevo objeto:
Código\(\PageIndex{6}\) (R):
Dos columnas (incluyendo especies) son factores, otras son numéricas (enteras o no). El objeto resultante es un marco de datos.
Lo siguiente es seleccionar nuestra especie y eliminar los niveles no utilizados:
Código\(\PageIndex{7}\) (R):
Para seleccionar especies de árboles en un comando, utilizamos la expresión lógica hecha con el operador %in% (por favor vea cómo funciona con?” comando %en%”).
La eliminación de niveles redundantes ayudará a usar los nombres de las especies para las parcelas de dispersión:
Código\(\PageIndex{8}\) (R):
Por favor, haz esta trama tú mismo. La clave es usar el factor ESPECIES como número, con el comando.numeric (). La función con () permite ignorar cc$ y por lo tanto guarda la escritura.
Sin embargo, hay un gran problema que al principio no es fácil de reconocer: en muchos lugares, los puntos se superponen entre sí y por lo tanto la cantidad de puntos de datos visibles es mucho menor que en el archivo de datos. Lo que es peor, no podemos decir si la primera y la tercera especie están bien o no bien segregadas porque no vemos cuántos valores de datos se encuentran en la “frontera” entre ellas. Este problema de diagrama de dispersión es bien conocido y hay soluciones alternativas:
Código\(\PageIndex{9}\) (R):
Por favor, ejecute este código usted mismo. La función jitter () agrega ruido aleatorio a las variables y caga puntos permitiendo ver lo que está a continuación. Sin embargo, todavía es difícil entender la cantidad de valores sobretrazados.
También hay:
Código\(\PageIndex{10}\) (R):
(Prueba estas variantes tú mismo. Cuando ejecutes la primera línea de código, verás la parcela de girasol, desarrollada exactamente a tales “casos de sobretrazado”. Refleja cuántos puntos se superponen. Sin embargo, no es fácil hacer que la parcela de girasoles () muestre un overplotting por separado para cada especie. El otro enfoque, SmoothScatter () sufre del mismo problema\(^{[1]}\).)
Para superar esto, desarrollamos la función pPoints () (Figura\(\PageIndex{1}\)):
Código\(\PageIndex{11}\) (R):
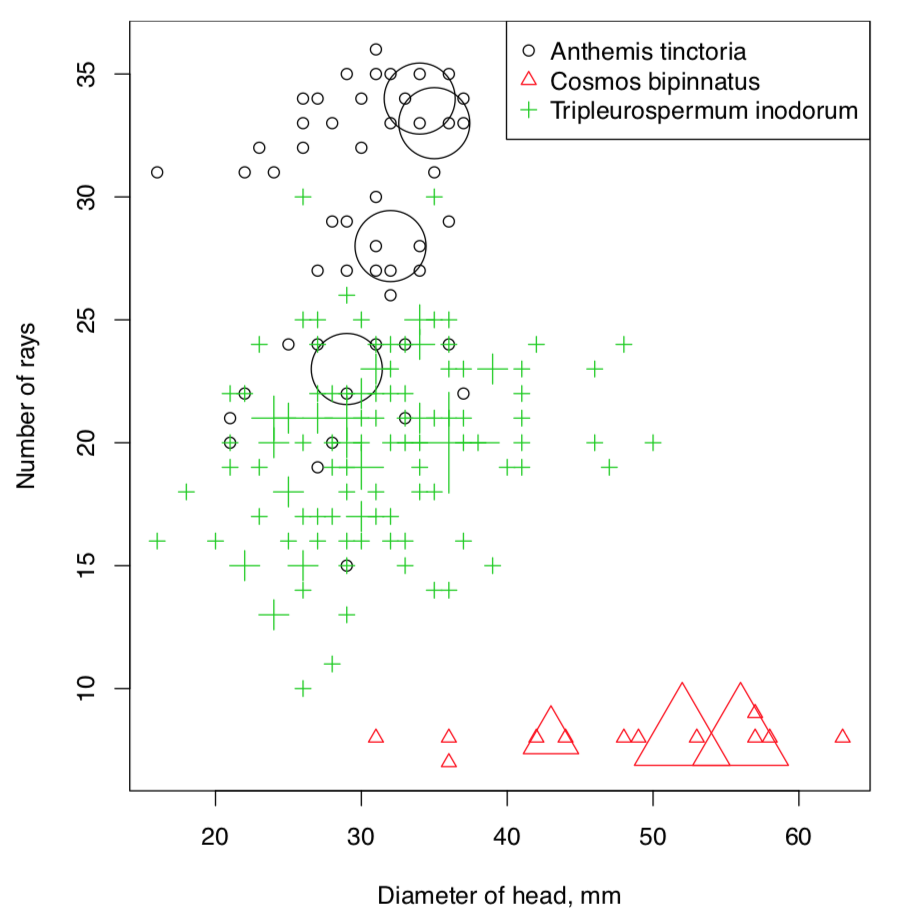
Por último, la respuesta. Como se puede ver, el cosmos del jardín está realmente separado de otras dos especies que a su vez podrían distinguirse con cierta certeza, principalmente porque el número de rayos en la manzanilla amarilla es más de 20. Este enfoque es posible mejorar. El capítulo de “minería de datos” dice cómo hacerlo.
Respuesta a la pregunta matricial. Al crear la matriz ma, definimos byRow=True, es decir, indicamos que los elementos deben unirse en una matriz fila por fila. En caso de byRow=false (default) habríamos obtenido la matriz idéntica a mb:
Código\(\PageIndex{12}\) (R):
Respuesta al ejercicio de clasificación. Para trabajar con columnas, tenemos que usar corchetes con coma y colocar comandos a la derecha:
Código\(\PageIndex{13}\) (R):
Tenga en cuenta que no podemos simplemente escribir order () después de la coma. Este comando devuelve el nuevo orden de columnas, así le dimos nuestros nombres de columna (names () devuelve nombres de columna para un marco de datos dado). Por cierto, sort () también habría funcionado aquí, ya que solo necesitábamos reorganizar un solo vector.
Referencias
1. También hay paquete hexbin que utiliza formas hexagonales y sombreado de color.

