4.7: Respuestas a los ejercicios
- Page ID
- 150115
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Respuesta a la pregunta de shapiro.test () estructura de salida. Primero, tenemos que recordar que casi todo lo que vemos en la consola R, es el resultado de print () 'ing algunas listas. Para extraer el componente de una lista, podemos llamarlo por signo de dólar y nombre, o por corchetes y número (si el componente no se nombra). Comprobemos la estructura con str ():
Código\(\PageIndex{1}\) (R):
Bueno, lo más probable es que el valor p provenga del componente p.value, esto es fácil. Compruébalo:
Código\(\PageIndex{2}\) (R):
Esto es lo que queremos. Ahora podemos insertarlo en el cuerpo de nuestra función.
Respuesta al ejercicio de “normalidad del abedul”. Primero, necesitamos verificar los datos y entender su estructura, por ejemplo con url.show (). Entonces podemos leerlo en R, verificar sus variables y aplicar la función Normality () a todas las columnas apropiadas:
Código\(\PageIndex{3}\) (R):
(Obsérvese cómo solo se seleccionaron columnas no categóricas para la comprobación de normalidad. Usamos Str () porque ayuda a verificar números de variables, y muestra que dos variables, LOBES y WINGS tienen datos faltantes. No hay problema en usar str () en su lugar.)
Solo CATKIN (longitud del catkin hembra) está disponible para los métodos paramétricos aquí. Es un caso frecuente en datos biológicos.
¿Qué pasa con la comprobación gráfica para la normalidad, histograma o gráfica QQ? Sí, debería funcionar pero tenemos que repetirlo 5 veces. Sin embargo, el paquete de celosía permite hacerlo en dos pasos y encajar en una gráfica de enrejado (Figura\(\PageIndex{2}\)):
Código\(\PageIndex{4}\) (R):
(La celosía de biblioteca requiere un formato de datos largo donde todas las columnas se apilan en una sola y los datos se suministran con la columna de identificador, es por eso que utilizamos la función stack () y la interfaz
Hay muchas parcelas de enrejado. Por favor, compruebe usted mismo el histograma de enrejado:
Código\(\PageIndex{5}\) (R):
(También hubo un ejemplo de cómo aplicar el tema en escala de grises a estas parcelas).
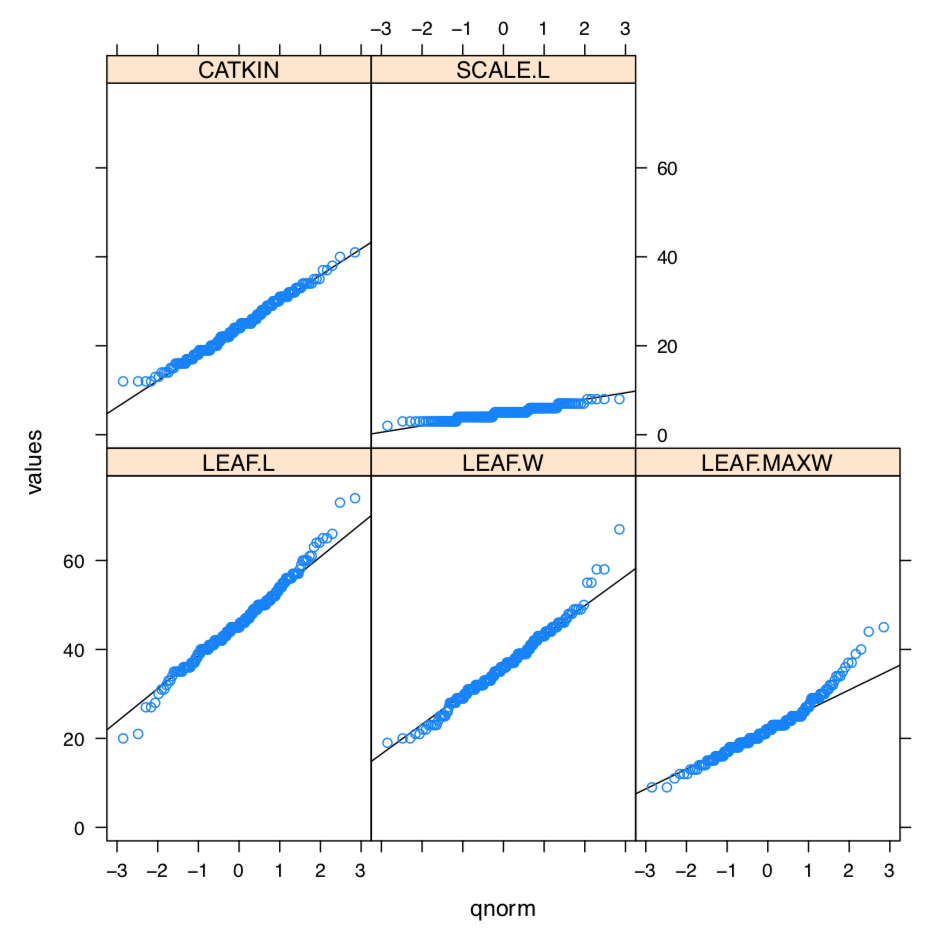
Como se puede ver, SCALE.L también podría aceptarse como “aproximadamente normal”. Entre otros, LEAF.MAXW es “menos normal”.
Respuesta al ejercicio de variabilidad de caracteres de abedul. Para crear una función, es bueno comenzar desde prototipo:
Código\(\PageIndex{6}\) (R):
Este prototipo no hace nada, pero en el siguiente paso puedes mejorarlo, por ejemplo, con el comando fix (CV). Después pruebe CV () con algún argumento sencillo. Si el resultado no es satisfactorio, vuelva a fijar (CV). Al final de este proceso, su función (en realidad, “envuelve” el cálculo de CV explicado anteriormente) podría verse así:
Código\(\PageIndex{7}\) (R):
Entonces sapply () podría usarse para verificar la variabilidad de cada columna de medición:
Código\(\PageIndex{8}\) (R):
Como se puede ver, LEAF.MAXW (ubicación del ancho máximo de hoja) tiene la mayor variabilidad. En el asmisc.r, hay la función CVs () que implementa esta y otras tres mediciones de variación relativa.
Responde a la pregunta sobre los datos dact.txt. El archivo complementario dact_c.txt lo describe como un extracto aleatorio de algunas mediciones de plantas. Desde el primer capítulo, sabemos que es sólo una secuencia de números. En consecuencia, scan () sería mejor que read.table (). Primero, cargue y verifique:
Código\(\PageIndex{9}\) (R):
Ahora, podemos comprobar la normalidad con nuestra nueva función:
Código\(\PageIndex{10}\) (R):
En consecuencia, debemos aplicar para dact solo aquellos análisis y características que sean robustos a la no normalidad:
Código\(\PageIndex{11}\) (R):
Intervalo de confianza para la mediana:
Código\(\PageIndex{12}\) (R):
(Usando la idea de que cada salida de prueba es una lista, extraemos el intervalo de confianza de la salida directamente. Por supuesto, sabíamos de antemano que el nombre de un componente que necesitamos es conf.int; este conocimiento podría obtenerse de la función help (sección “Valor”). El intervalo resultante es amplio).
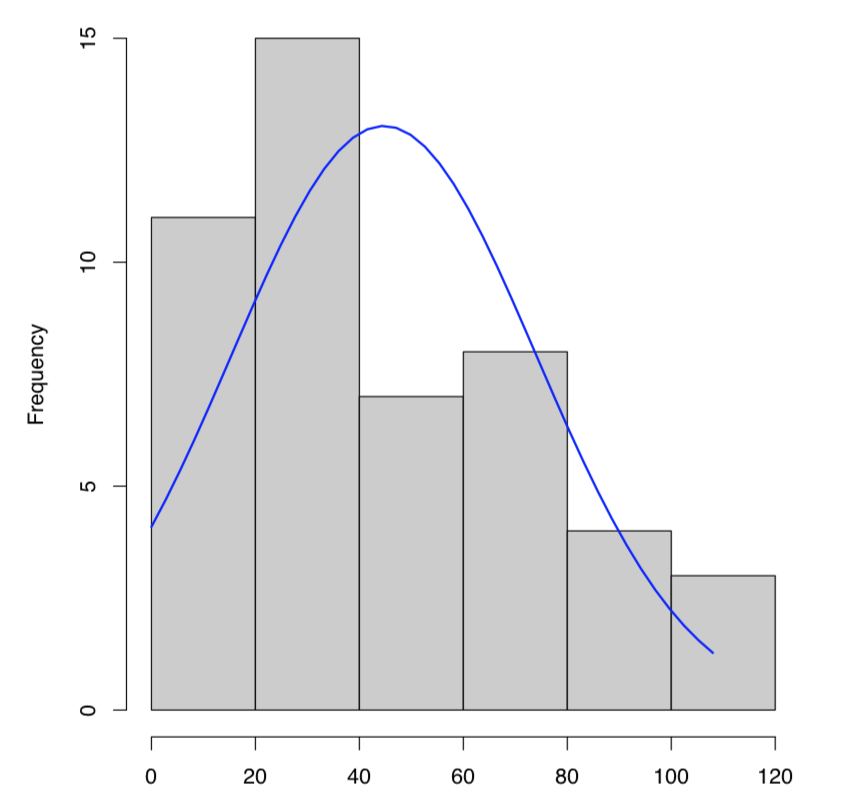
Para trazar datos numéricos simples, es preferible el histograma (Figura\(\PageIndex{3}\)) (las gráficas de caja son mejores para la comparación entre variables):
Código\(\PageIndex{13}\) (R):
Similar al histograma es la gráfica de vapor y hoja:
Código\(\PageIndex{14}\) (R):
Además, aquí calcularemos asimetría y curtosis, tercer y cuarto momentos centrales (Figura\(\PageIndex{4}\)). La asimetría es una medida de cuán asimétrica es la distribución, la curtosis es una medida de lo puntiforme que es. La distribución normal tiene asimetría y curtosis cero mientras que la distribución uniforme “plana” tiene asimetría cero y curtosis aproximadamente\(-1.2\) (compruébalo tú mismo).
¿Qué pasa con los datos dact? A partir del histograma (Figura\(\PageIndex{3}\)) y tallo y hoja podemos predecir asimetría positiva (asimetría de distribución) y curtosis negativa (distribución más plana de lo normal). Para verificar, primero es necesario cargar la biblioteca e1071:
Código\(\PageIndex{15}\) (R):
Respuesta a la pregunta sobre los nenúfares. Primero, necesitamos verificar los datos, cargarlos en R y verificar el objeto resultante:
Código\(\PageIndex{16}\) (R):
(La función Str () muestra los números de columna y la presencia de NA.)
Una de las posibles formas de proceder es examinar las diferencias entre especies por cada carácter, con cuatro parcelas de caja pareadas. Para hacerlos en una fila, vamos a emplear para () ciclo:
Código\(\PageIndex{17}\) (R):
(Aquí no, sino en muchos otros casos, para () en R es mejor reemplazar con comandos de la familia apply (). La función Boxplot acepta argumentos “ordinarios” pero en este caso, la interfaz de fórmula con tilde es mucho más práctica.)
Por favor revise esta parcela usted mismo.
Sin embargo, es aún mejor comparar caracteres escalados en una trama. La primera variante es cargar la biblioteca de celosía y crear una gráfica de enrejado similar a la Figura 7.1.8 o la Figura 7.1.7:
Código\(\PageIndex{18}\) (R):
(Como de costumbre, las parcelas de enrejado “quieren” una interfaz de forma larga y fórmula).
Por favor, revisa esta parcela tú mismo.
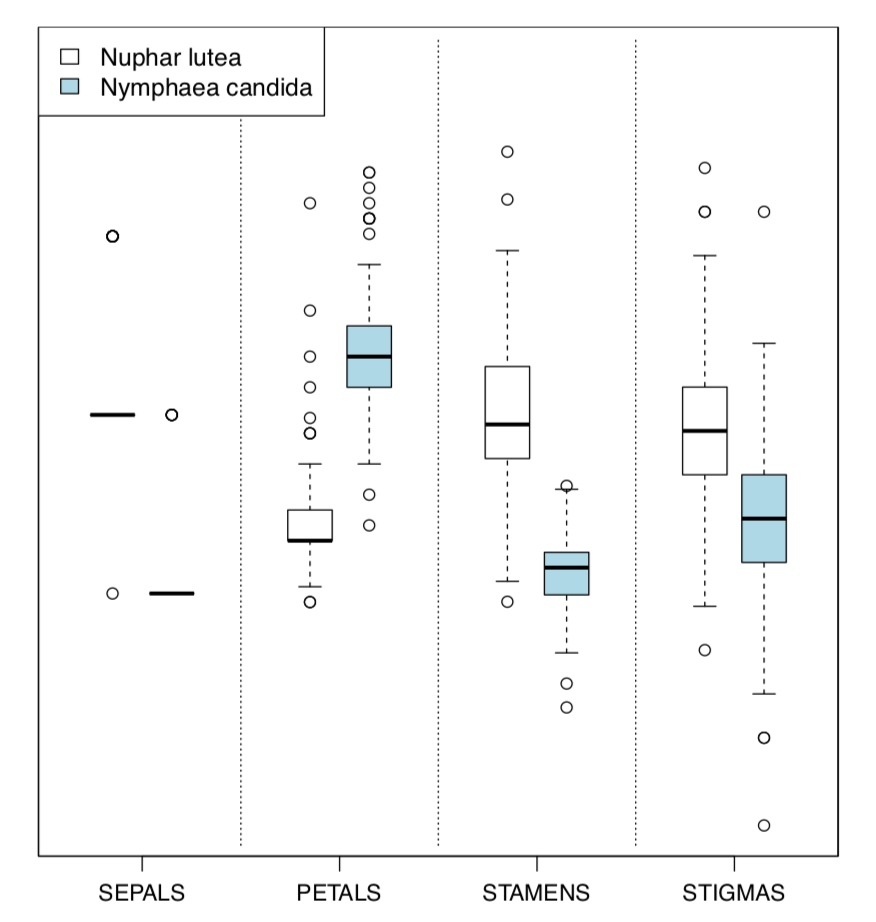
Alternativa es el comando Boxplots () (Figura\(\PageIndex{5}\)). No es una trama enrejada, sino diseñada con un objetivo similar de comparar muchas cosas a la vez:
Código\(\PageIndex{19}\) (R):
(Por defecto, Boxplots () rota las etiquetas de caracteres, pero este comportamiento no es necesario con 4 caracteres. Esta gráfica usa scale () por lo que el eje y, por defecto, no se proporciona.)
O, con un Linechart aún más nítido () (Figura\(\PageIndex{6}\)):
Código\(\PageIndex{20}\) (R):
(A veces, los IQR son mejores para percept si agregas grid () a la trama. Pruébalo tú mismo.)
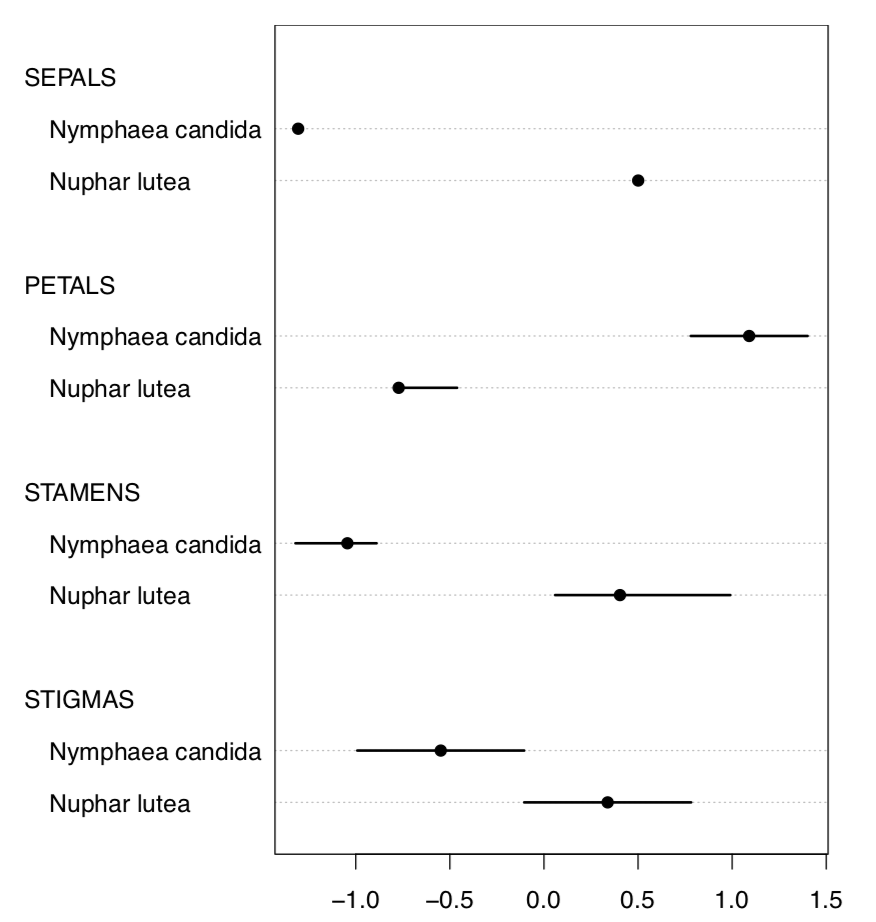
Evidentemente (después de SEPALOS), PÉTALOS y ESTAMENTOS hacen la mejor resolución de especies. Para obtener valores numéricos, es mejor verificar primero la normalidad.
Tenga en cuenta que la identidad de especies es la característica natural e interna de nuestros datos. Por lo tanto, es teóricamente posible que el mismo carácter en una especie exhiba una distribución normal mientras que en otra no. Es por ello que se debe verificar la normalidad por carácter por especie. Esta idea se acerca al concepto de efectos fijos que son tan útiles en los modelos lineales (ver capítulos siguientes). Los efectos fijos se oponen a los efectos aleatorios que no son naturales para los objetos estudiados (por ejemplo, si se muestrea solo una especie de nenúfares en el lago dos veces).
Código\(\PageIndex{21}\) (R):
(Function aggregate () no sólo aplica función anónima a todos los elementos de su argumento, sino que también la divide sobre la marcha con por lista de factor (es). Similar es tapply () pero funciona sólo con un vector. Otra variante es usar split () y luego apply () la función de informes a cada parte por separado.)
Por cierto, el código anterior es bueno para aprender pero en nuestro caso particular, ¡no se requiere verificación de normalidad! Esto se debe a que los números de pétalos y estambres son caracteres discretos y por lo tanto deben tratarse con métodos no paramétricos por definición.
Así, para los intervalos de confianza, debemos proceder con métodos no paramétricos:
Código\(\PageIndex{22}\) (R):
Los intervalos de confianza reflejan la posible ubicación del valor central (aquí la mediana). Pero aún necesitamos reportar nuestros centros y rangos (¡el intervalo de confianza no es un rango!). Podemos usar tanto summary () (pruébalo tú mismo), o algún resultado personalizado que, por ejemplo, puede emplear la desviación absoluta mediana:
Código\(\PageIndex{23}\) (R):
Ahora podemos dar la respuesta como “si hay 12—16 pétalos y 100—120 estambres, esto es probablemente un lirio de agua amarillo, de lo contrario, si hay 23-29 pétalos y 66—88 estambres, esto es probablemente un lirio de agua blanco”.
Respuesta a la pregunta sobre filotaxis. Primero, necesitamos mirar el archivo de datos, ya sea con url.show (), o en la ventana del navegador y determinar su estructura. Hay cuatro columnas separadas por tabuladores con encabezados, y al menos la segunda columna contiene espacios. En consecuencia, necesitamos decirle a read.table () tanto el separador como los encabezados y luego verificar inmediatamente la “anatomía” del nuevo objeto:
Código\(\PageIndex{24}\) (R):
Como ve, tenemos 11 familias y por lo tanto 11 proporciones para crear y analizar:
Código\(\PageIndex{25}\) (R):
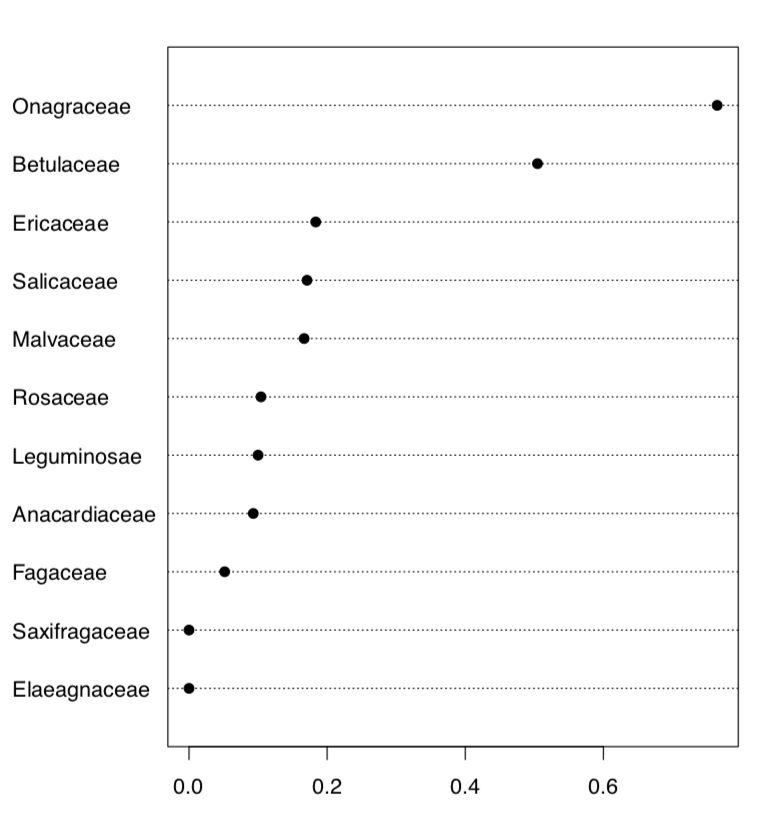
Aquí creamos 10 primeras fórmulas clásicas de filotaxis (diez es suficiente ya que las fórmulas de orden superior son extremadamente raras), luego hicimos estas fórmulas (clásicas y no clásicas) a partir de datos y finalmente hicimos una tabla a partir de la expresión lógica que verifica si las fórmulas del mundo real están presentes en el clásico hecho artificialmente secuencia. Dotchart (Figura\(\PageIndex{7}\)) es probablemente la mejor manera de visualizar esta tabla. Evidentemente, Onagraceae (familia onagra) tiene la mayor proporción de FALSE, ahora necesitamos proporciones reales y finalmente, prueba de proporción:
Código\(\PageIndex{26}\) (R):
Como ves, la proporción de fórmulas no clásicas en Onagraceae (casi 77%) es estadísticamente diferente de la proporción promedio de 27%.
Respuesta a la pregunta de la encuesta de salida del “Prólogo”. Aquí está la manera de calcular cuántas personas podríamos querer pedir para estar seguros de que nuestra muestra 48% y 52% son “reales” (representan la población):
Código\(\PageIndex{27}\) (R):
¡Tenemos que preguntar a casi 5 mil personas!
Para calcular esto, se utilizó una especie de prueba de potencia que se utilizan con frecuencia para planificar experimentos. Hicimos power=0.8 ya que es el valor típico del poder utilizado en las ciencias sociales. El siguiente capítulo da definición de potencia (como término estadístico) y algo más de información sobre la salida de prueba de potencia.

