7.3: Aprendizaje automático
- Page ID
- 150002
Los métodos explicados en esta sección no son solo visualizaciones. Juntos, frecuentemente llamaban “clasificación con aprendizaje”, “clasificación supervisada”, “aprendizaje automático”, o simplemente “clasificación”. Todos ellos se basan en la idea de aprender:
... Se agolpó y se puso de pie.... No veía más que colores, colores que se negaron a formarse en cosas. Además, aún no sabía nada lo suficientemente bien como para verlo: no se pueden ver las cosas hasta que no se sabe más o menos cuáles son\(^{[1]}\). Su primera impresión fue de un mundo brillante y pálido, un mundo de acuarela
de la caja de pintura de un niño; un momento después reconoció el cinturón plano de azul claro como una lámina de agua, o de algo así como agua, que casi se puso de pie. Estaban en la orilla de un lago o río...
C.S.Lewis. Fuera del Planeta Silencioso.
En primer lugar, pequeña parte de los datos donde ya se conoce la identidad (conjunto de datos de entrenamiento) se utiliza para desarrollar (ajustar) el modelo de clasificación (Figura\(\PageIndex{2}\)). En el siguiente paso, este modelo se utiliza para clasificar objetos con identidad desconocida (testing dataset). En la mayoría de estos métodos, es posible estimar la calidad de la clasificación y también evaluar la significancia de cada carácter.
Vamos a crear conjuntos de datos de entrenamiento y pruebas a partir de datos de iris:
Código\(\PageIndex{1}\) (R):
(iris.unknown es por supuesto el falso desconocido así que para usarlo correctamente, debemos especificar iris.unknown [, -5]. Por otro lado, la información de especies ayudará a crear tabla de clasificación errónea (matriz de confusión, ver más adelante).)
Aprendiendo con regresión
Análisis discriminante lineal
Uno de los métodos más simples de clasificación es el análisis discriminante lineal (LDA). La idea básica es crear el conjunto de funciones lineales que “decidan” cómo clasificar el objeto en particular.
Código\(\PageIndex{2}\) (R):
El entrenamiento resultó en la hipótesis que permitió que casi todas las plantas (con excepción de siete Iris virginica) fueran colocadas en el grupo adecuado. Tenga en cuenta que LDA no requiere escalado de variables.
Es posible verificar los resultados de LDA con métodos inferenciales. El análisis multidimensional de variación (MANOVA) permite comprender la relación entre datos y modelo (clasificación a partir de LDA):
Código\(\PageIndex{3}\) (R):
Aquí son importantes tanto el valor p basado en las estadísticas de Fisher, como también el valor de las estadísticas de Wilks que es la razón de verosimilitud (en nuestro caso, la probabilidad de que los grupos no sean diferentes).
Es posible verificar la importancia relativa de cada personaje en LDA con técnicas similares a Anova:
Código\(\PageIndex{4}\) (R):
(Esta idea también es aplicable a otros métodos de clasificación).
... y también visualizar los resultados de LDA (Figura\(\PageIndex{3}\)):
Código\(\PageIndex{5}\) (R):
(Tenga en cuenta elipses de confianza del 95% con centros.)
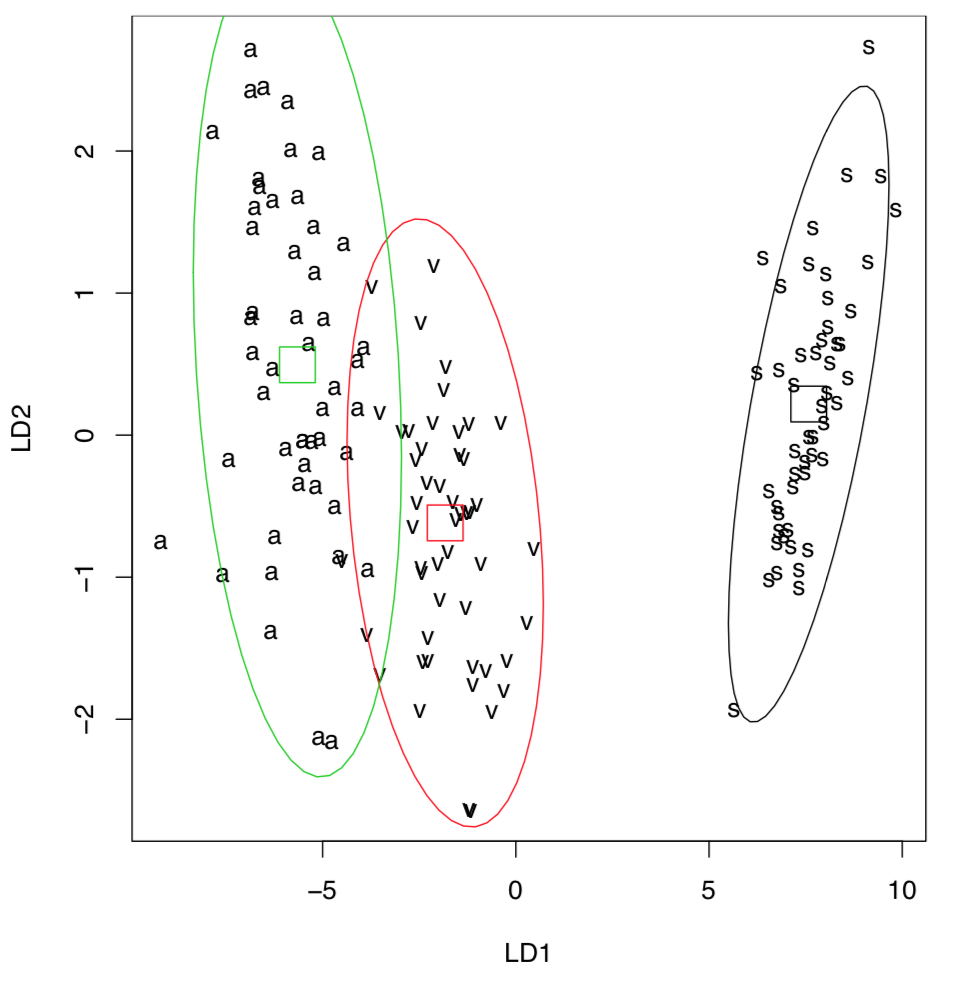
Para colocar todos los puntos en la parcela, simplemente usamos todos los datos como entrenamiento. Obsérvese la buena discriminación (mayor que en PCA, MDS o agrupamiento), incluso entre Iris versicolor cercano e I. virginica. Esto se debe a que la LDA frecuentemente sobreestima las diferencias entre grupos. Esta característica, y también la parametricidad y linealidad de LDA, la hicieron menos utilizada en los últimos años.
Con LDA, es fácil ilustrar un concepto más importante del aprendizaje automático: la calidad de la capacitación. Considera el siguiente ejemplo:
Código\(\PageIndex{6}\) (R):
Error de clasificación errónea aquí ¡casi dos veces más grande! ¿Por qué?
Código\(\PageIndex{7}\) (R):
Bueno, el uso de sample () (y particular set.seed () value) resultó en una muestra de entrenamiento sesgada, es por eso que nuestro segundo modelo se entrenó tan mal. Nuestra primera forma de muestrear (cada quinto iris) fue mejor, y si es necesario usar sample (), considere muestrear cada especie por separado.
Ahora, vuelva a la configuración predeterminada del generador de números aleatorios:
Código\(\PageIndex{8}\) (R):
Tenga en cuenta que es ampliamente conocido que si bien el LDA se desarrolló sobre material biológico, este tipo de datos rara vez cumple con dos supuestos clave de este método: (1) normalidad multivariada y (2) homocedasticidad multivariada. Sorprendentemente, ¡incluso los datos de Fisher's Iris con los que se inventó LDA, no cumplen con estos supuestos! Por lo tanto, no recomendamos usar LDA y mantenerla aquí principalmente con fines de enseñanza.
Partición recursiva
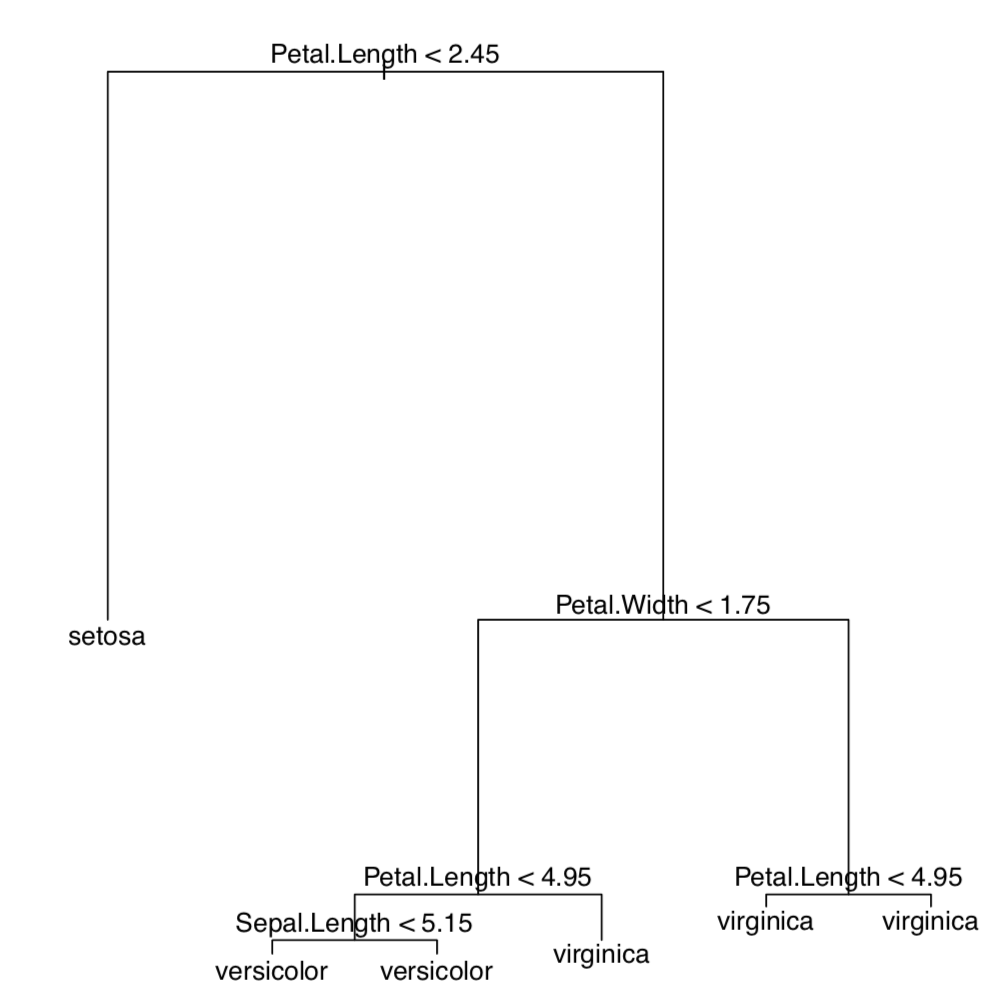
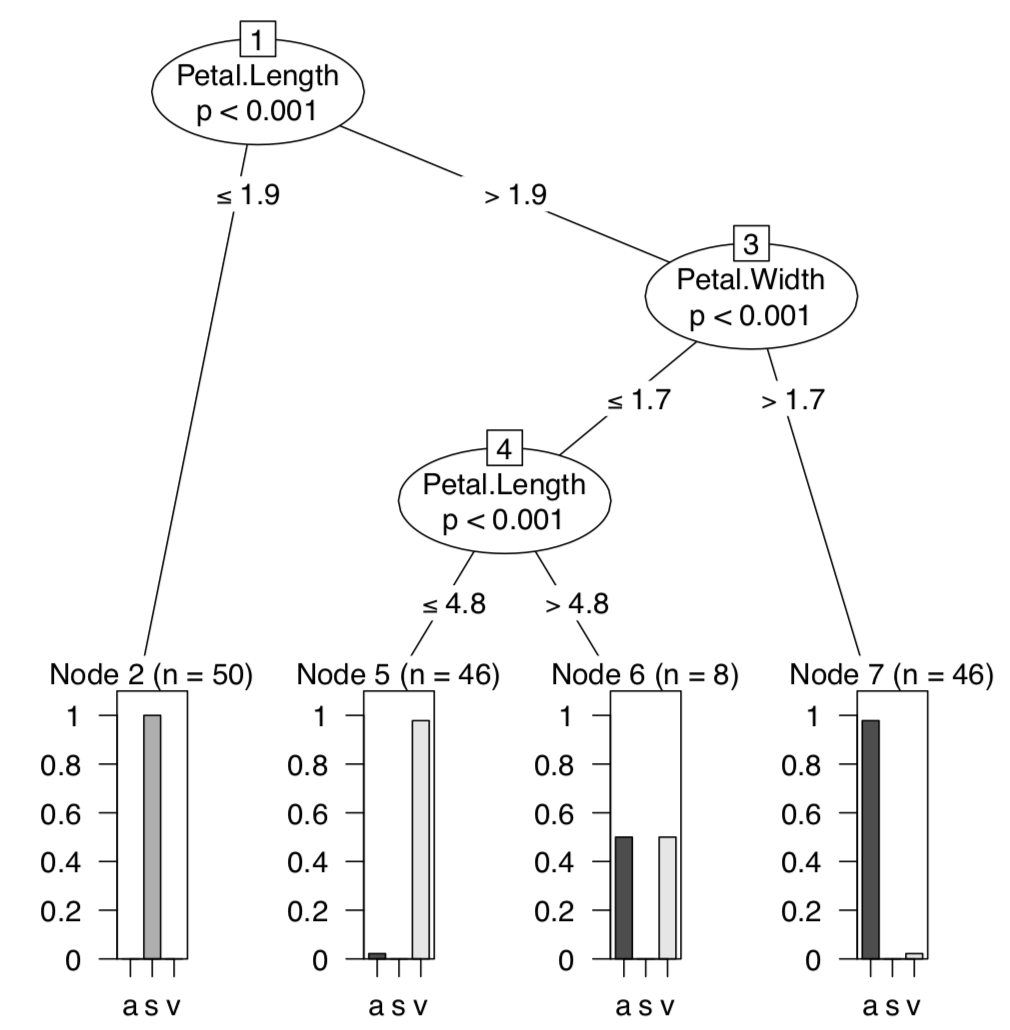
Para reemplazar el análisis discriminante lineal, se inventaron múltiples métodos con ideas de fondo similares. La partición recursiva, o árboles de decisión (árboles de regresión, árboles de clasificación), permiten, entre otras cosas, hacer y visualizar el tipo de clave de discriminación donde cada paso da como resultado la división de objetos en dos grupos (Figura\(\PageIndex{4}\)):
Código\(\PageIndex{9}\) (R):
Primero cargamos el paquete de árbol que contiene la función tree () (rpart es otro paquete que hace árboles de clasificación). Luego nuevamente usamos todo el conjunto de datos como datos de entrenamiento. La parcela muestra que todas las plantas con longitud de pétalo inferior a 2.45 cm pertenecen a Iris setosa, y del resto aquellas plantas que tienen un ancho de pétalo inferior a 1.75 cm y una longitud de pétalo superior a 4.95 cm, son I. versicolor; todos los demás iris pertenecen a I. virginica.
De hecho, estos árboles son resultado de algo similar al “análisis discriminante jerárquico”, y es posible utilizarlos para la clasificación supervisada:
Código\(\PageIndex{10}\) (R):
Trate de averiguar qué caracteres distinguen especies de colas de caballo descritas en el archivo de datos eq.txt. El archivo eq_c.txt contiene la descripción de los caracteres.
Package Party ofrece sofisticados métodos de partición recursiva junto con gráficas de árbol avanzadas (Figura\(\PageIndex{5}\)):
Código\(\PageIndex{11}\) (R):
(Para los nombres de especies, usamos abreviaturas de una letra.)
Ensamble learnig
Bosque Aleatorio
El otro método, internamente similar a los árboles de regresión, gana rápidamente popularidad. Este es el Bosque Aleatorio. Su nombre proviene de la capacidad de utilizar numerosos árboles de decisión y construir el complejo modelo de clasificación. Random Forest pertenece a los métodos de ensacado de conjuntos; utiliza bootstrap (ver en Apéndice) para multiplicar el número de árboles en el modelo (de ahí “bosque”). A continuación se muestra un ejemplo del clasificador Random Forest hecho a partir de los datos del iris:
Código\(\PageIndex{12}\) (R):
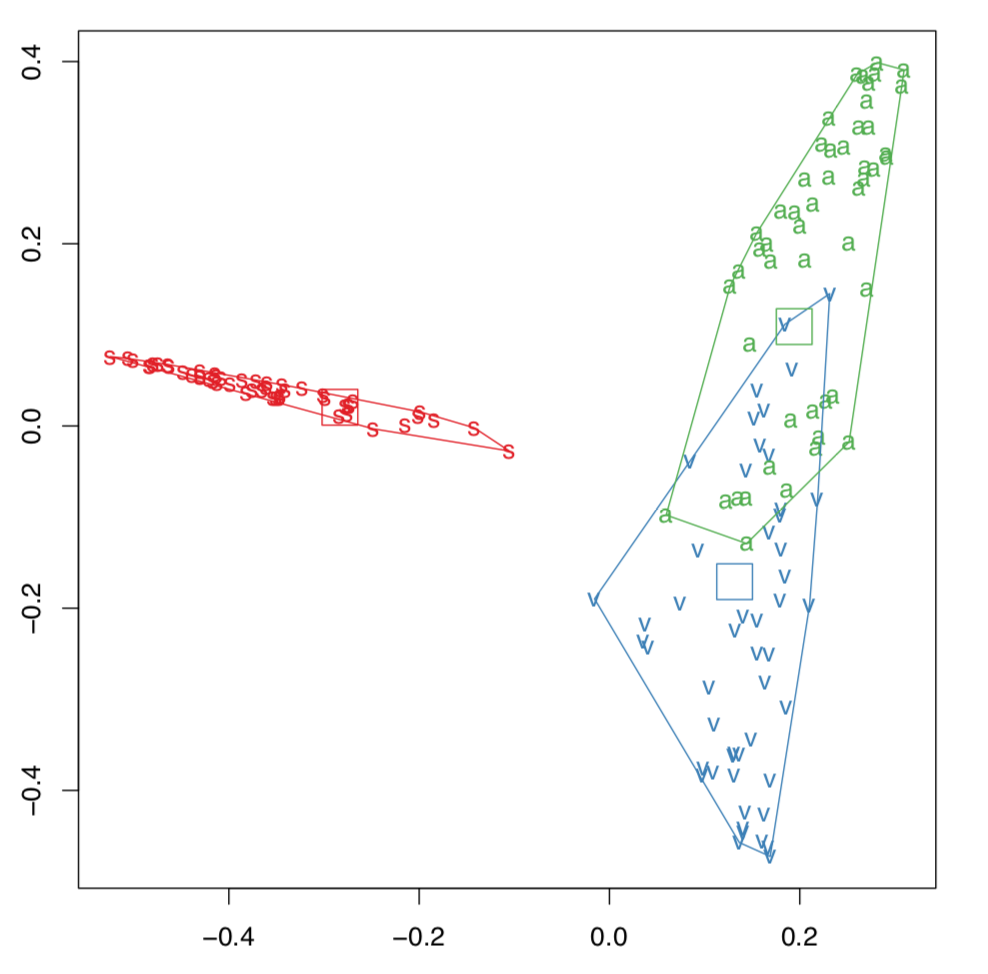
Aquí los resultados son similares a LDA pero Random Forest permite más. Por ejemplo, puede aclarar la importancia de cada personaje (con función importance ()), y revelar distancias de clasificación (proximidades) entre todos los objetos de subconjunto de entrenamiento (estas distancias podrían ser a su vez utilizadas para la agrupación). Random Forest también podría visualizar el dataset multidimensional (Figura\(\PageIndex{6}\)):
Código\(\PageIndex{13}\) (R):
(Aplicamos varios trucos para mostrar los cascos convexos y sus centroides).
Package ranger implementa una variante aún más rápida del algoritmo Random Forest, también puede emplear cálculos paralelos.
Impulso de gradiente
Hay muchos métodos de clasificación débiles que suelen cometer errores de clasificación errónea altos. Sin embargo, muchos de ellos también son ultrarrápidos. Entonces, ¿es posible combinar muchos aprendices débiles para hacer el fuerte? ¡Sí! Esto es lo que hacen los métodos de impulso. El impulso de gradiente emplea la optimización de varios pasos y ahora se encuentra entre las técnicas de aprendizaje más frecuentemente utilizadas. En R, hay varios paquetes de refuerzo de gradiente, por ejemplo, xgboost y gbm:
Código\(\PageIndex{14}\) (R):
(La trama es puramente técnica; en la forma anterior, mostrará el efecto marginal (efecto sobre la pertenencia) de la 1ª variable. Por favor, hágalo usted mismo. “Truco de membresía” selecciona la “mejor especie” de tres alternativas, ya que gbm () informa el resultado de clasificación en forma difusa.)
Aprendiendo con proximidad
El algoritmo K-Neighbors más cercano (o KnN) es el “clasificador perezoso” porque no funciona hasta que se suministran datos desconocidos:
Código\(\PageIndex{15}\) (R):
KnN se basa en el cálculo de distancia y el “voto”. Calcula distancias desde cada objeto desconocido hasta cada objeto del conjunto de entrenamiento. A continuación, considera varios (5 en el caso anterior) vecinos más cercanos con identidad conocida y encuentra cuál id prevalece. Esta identificación prevalente asignada al miembro desconocido. La función knn () usa distancias euclidianas pero en principio, cualquier distancia funcionaría para kNn.
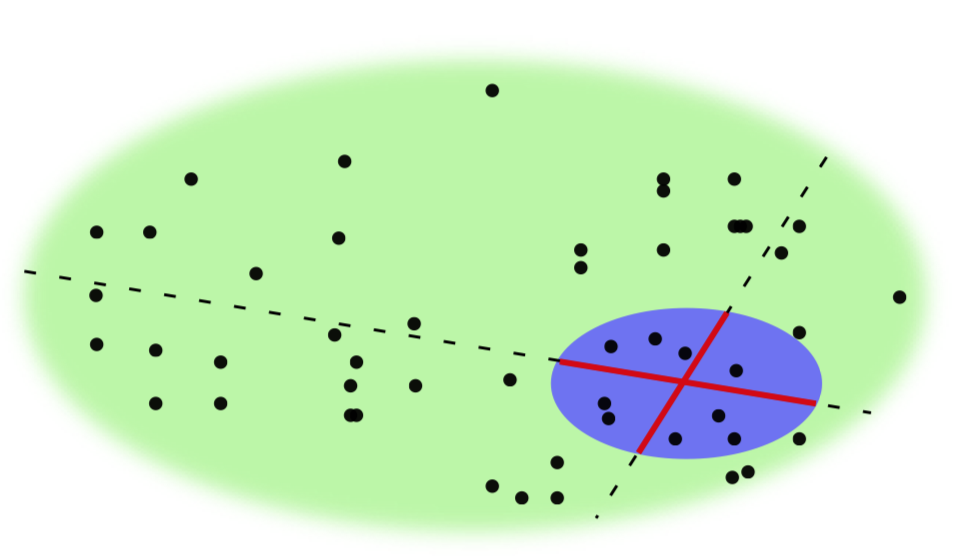
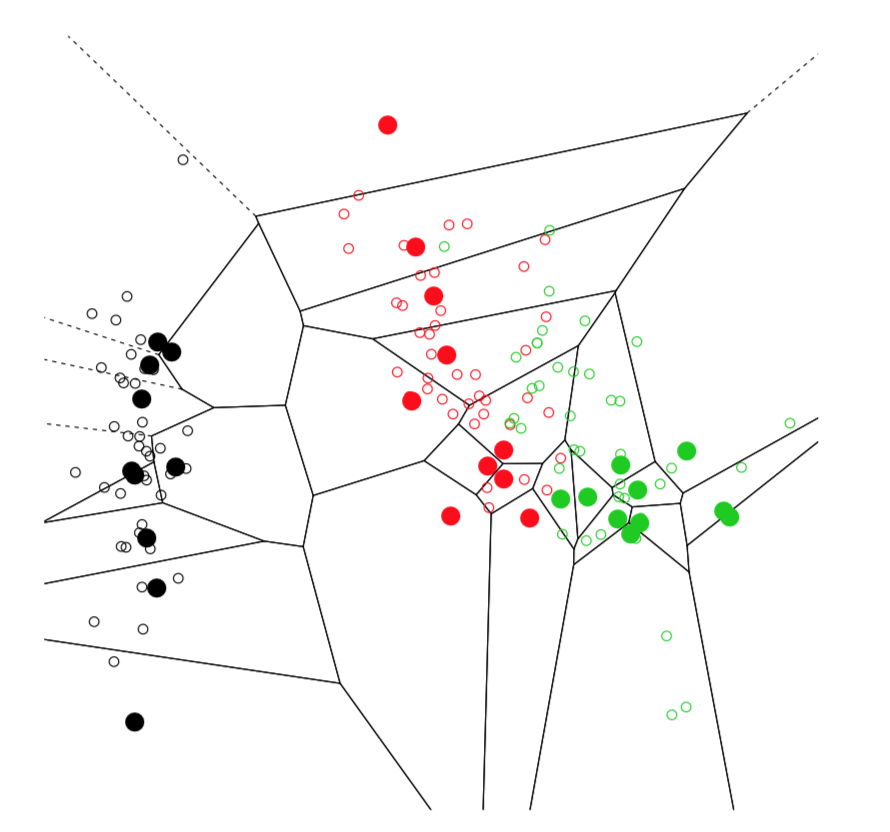
Para ilustrar la idea de los vecinos más cercanos, utilizamos la descomposición de Voronoi, la técnica que se aproxima tanto a KnN como al cálculo de distancia:
Código\(\PageIndex{16}\) (R):
La gráfica (Figura\(\PageIndex{7}\)) contiene múltiples celdas que representan barrios de muestra de entrenamiento (puntos grandes). Esto no es exactamente lo que hace KnN, pero la idea es igual. De hecho, la trama Voronoi es una buena herramienta para visualizar cualquier enfoque basado en la distancia.
Clasificación de profundidad basada en qué tan cerca se encuentra un punto arbitrario del espacio a un centro definido implícitamente de una nube de datos multidimensional:
Código\(\PageIndex{17}\) (R):
Aprendiendo con reglas
El clasificador Naïve Bayes es uno de los algoritmos de aprendizaje automático más simples que trata de clasificar objetos en función de las probabilidades de atributos previamente vistos. De manera inesperada, suele ser un buen clasificador:
Código\(\PageIndex{18}\) (R):
Tenga en cuenta que el clasificador Naïve Bayes podría usar no solo numéricos como los anteriores, sino también predictores nominales (que es similar al análisis de correspondencia).
El método Apriori es similar a los árboles de regresión pero en lugar de clasificar objetos, investiga reglas de asociación entre clases de objetos. Este método podría ser utilizado no sólo para encontrar estas reglas sino también para hacer clasificación. Tenga en cuenta que los datos de iris de medición son menos adecuados para las reglas de asociación que los datos nominales, y primero necesitan discretización:
Código\(\PageIndex{19}\) (R):
(Las reglas se explican por sí mismas. ¿Qué opinas, este método funciona mejor para los datos nominales? Por favor averiguarlo.)
Aprendiendo de la caja negra
Famoso SVM, Support Vector Machines es una técnica de kernel que calcula parámetros de los hiperplanos dividiendo múltiples grupos en el espacio multidimensional de caracteres:
Código\(\PageIndex{20}\) (R):
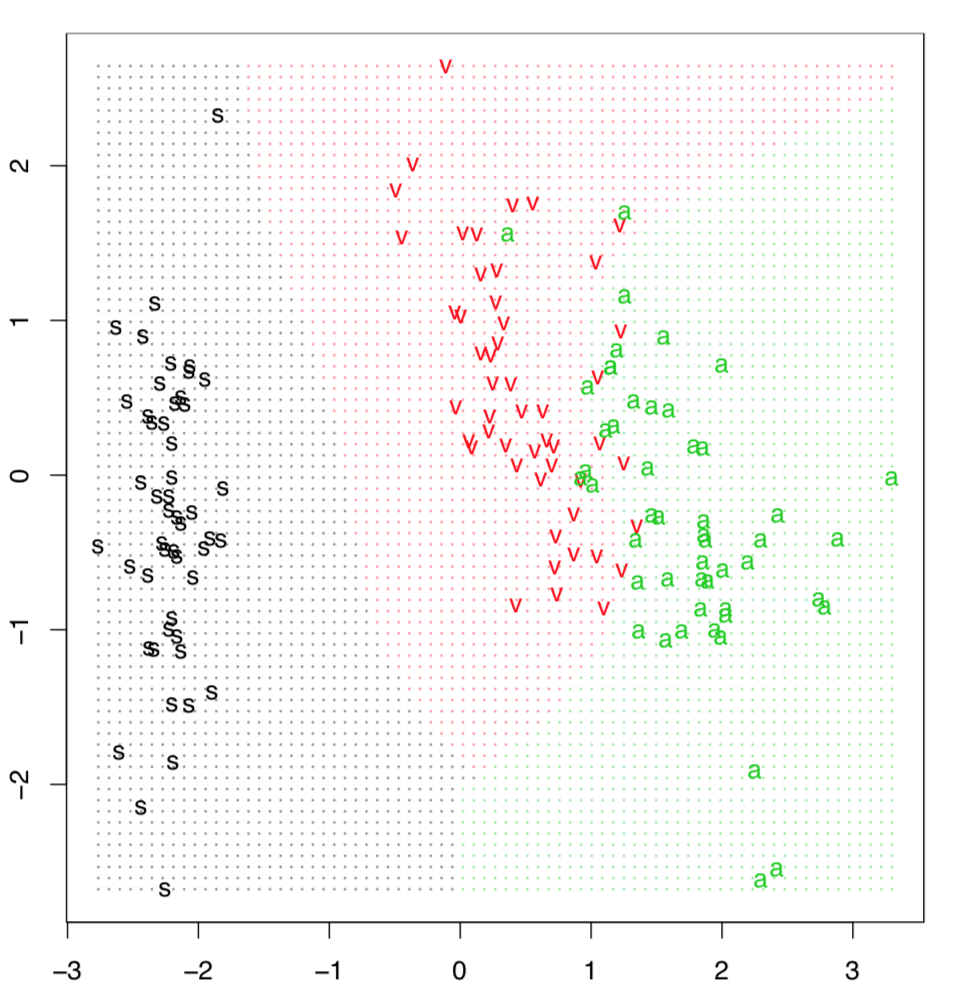
La clasificación o cuadrícula de predicción a menudo ayuda a ilustrar el método SVM. Los puntos de datos se disponen con PCA para reducir la dimensionalidad, y luego el clasificador predice la identidad para cada punto en la cuadrícula hecha artificialmente (Figura\(\PageIndex{8}\)). Esto es posible realizarlo manualmente pero la función Gradd () simplifica el trazado:
Código\(\PageIndex{21}\) (R):
Y finalmente, ¡redes neuronales! Este nombre se utiliza para la técnica estadística basada en algunas características de las células neuronales, las neuronas. Primero, necesitamos preparar datos y convertir la variable categórica Species en tres variables ficticias lógicas:
Código\(\PageIndex{22}\) (R):
Ahora, llamamos paquete neuralnet y procedemos al cálculo principal. El paquete “quiere” suministrar todos los términos del modelo explícitamente:
Código\(\PageIndex{23}\) (R):
(Tenga en cuenta el uso de set.seed (), esto es para que sus resultados sean similares a los que se presentan aquí.)
Ahora predice (con la función compute ()) y verifica la clasificación errónea:
Código\(\PageIndex{24}\) (R):
Los resultados de la predicción de redes neuronales son difusos, similares a los resultados de agrupamiento difuso o árboles de regresión, por lo que se aplicó qué.max () para cada fila. Como ve, este es uno de los errores de clasificación errónea más bajos.
Es posible trazar la red real:
Código\(\PageIndex{25}\) (R):
La gráfica (Figura 7.4.1) es un poco esotérica para el novato, pero ojalá se introduzca en el método porque existe una aparente estructura multicapa que se utiliza para las decisiones de redes neuronales.
Referencias
1. Énfasis mío.


