7.7: Respuestas a ejercicios
- Page ID
- 150006
Respuesta a la pregunta de las estrellas.
Primero, cargue los datos y como se sugirió anteriormente, convierta las coordenadas en decimales:
Código\(\PageIndex{1}\) (R):
A continuación, algunas parcelas preliminares (por favor hágalas tú mismo):
Código\(\PageIndex{2}\) (R):
Ahora, cargue el paquete dbscan e intente encontrar donde el número de “constelaciones” es máximo:
Código\(\PageIndex{3}\) (R):
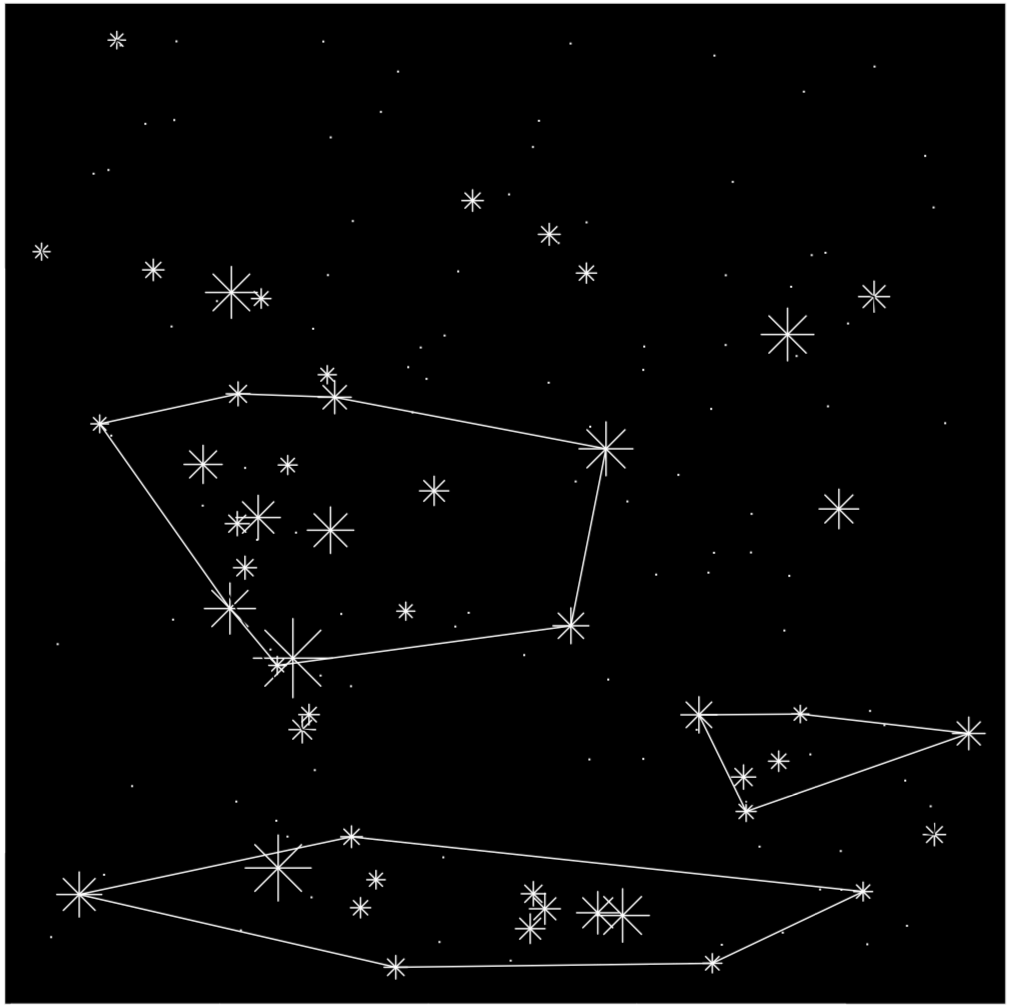
Trazar el “cielo nocturno” prettificado (Figura\(\PageIndex{1}\)) con constelaciones encontradas:
Código\(\PageIndex{4}\) (R):
dev.off ()
Para acceder a un acuerdo entre dos clasificaciones (dos sistemas de constelaciones) podríamos usar el índice Rand ajustado que cuenta correspondencias:
Código\(\PageIndex{5}\) (R):
(Por supuesto, es bajo.)
Respuesta al ejercicio de clasificación de cerveza. Para hacer la clasificación jerárquica, primero necesitamos hacer la matriz de distancia. Echemos un vistazo a los datos:
Código\(\PageIndex{6}\) (R):
Los datos son binarios y por lo tanto necesitamos el método específico de cálculo de distancia. Vamos a utilizar aquí Jaccard distancia implementado en función vegdist () desde el paquete vegano. También es posible usar aquí otros métodos como “binary” de la función core dist (). El siguiente paso sería la construcción del dendrograma (Figura\(\PageIndex{2}\)):
Código\(\PageIndex{7}\) (R):
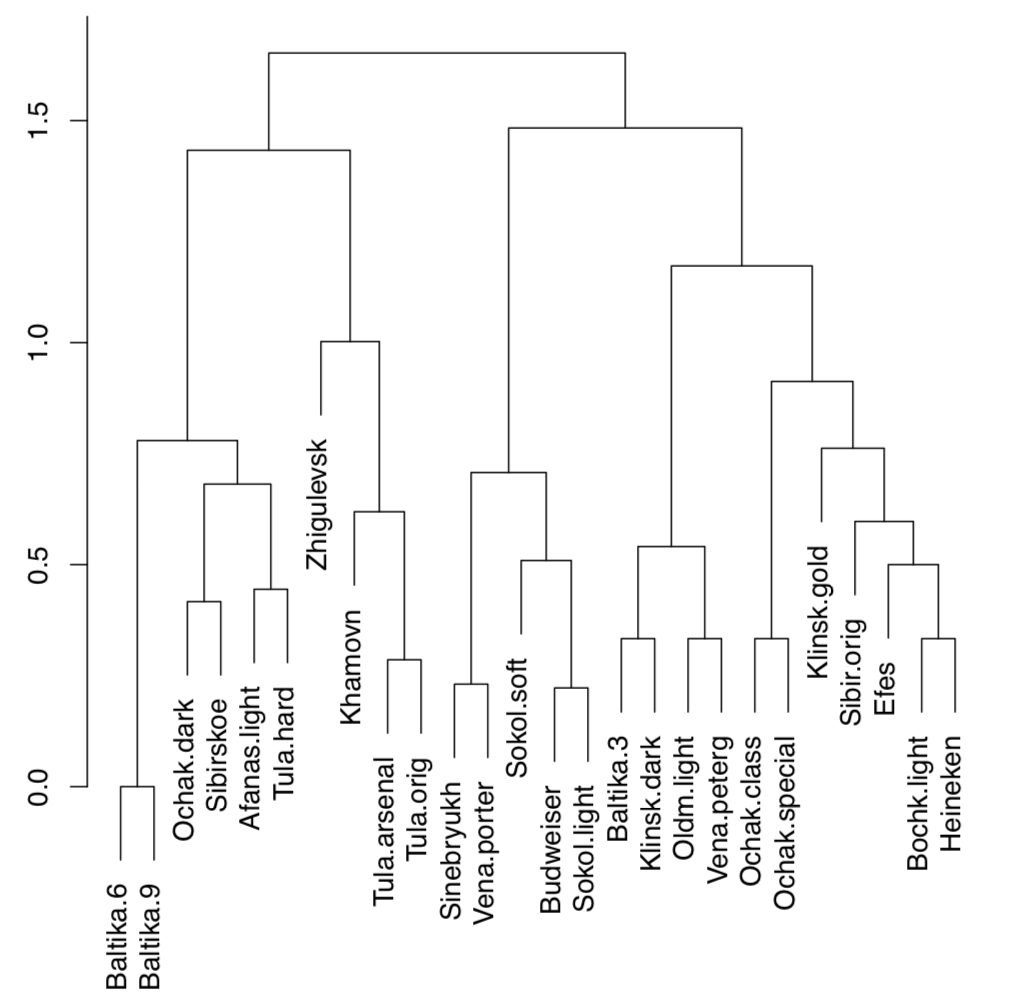
Hay dos grandes grupos (en aproximadamente el nivel de disimilitud 1.7), podemos llamarlos “Baltika” y “Budweiser”. En la siguiente división (aproximadamente en 1.4 disimilitud), hay dos subgrupos en cada grupo. Todas las demás divisiones son significativamente más profundas. Por lo tanto, es posible realizar la siguiente clasificación jerárquica:
- Grupo Baltika
- Subgrupo Baltika: Baltika.6, Baltika.9, ochak.Dark, afanas.Light, Sibirskoe, tula.Duro
- Subgrupo de Tula: Zhigulevsk, Khamovn, Tula.Arsenal, Tula.orig
- Grupo Budweiser
- Subgrupo Budweiser: Sinebryukh, Vena.porter, Sokol.soft, Budweiser, Sokol.light
- Subgrupo Ochak: Baltika.3, Klinsk.Dark, Oldm.Light, Vena.Peterg, Ochak.class, OChak.Special, Klinsk.Gold, Sibir.Orig, Efes, Bochk.Light, Heineken
También es una buena idea verificar la clasificación resultante con cualquier otro método de clasificación, como agrupación no jerárquica, escalado multidimensional o incluso PCA. Cuanto más consistente sea la clasificación anterior con este segundo enfoque, mejor.
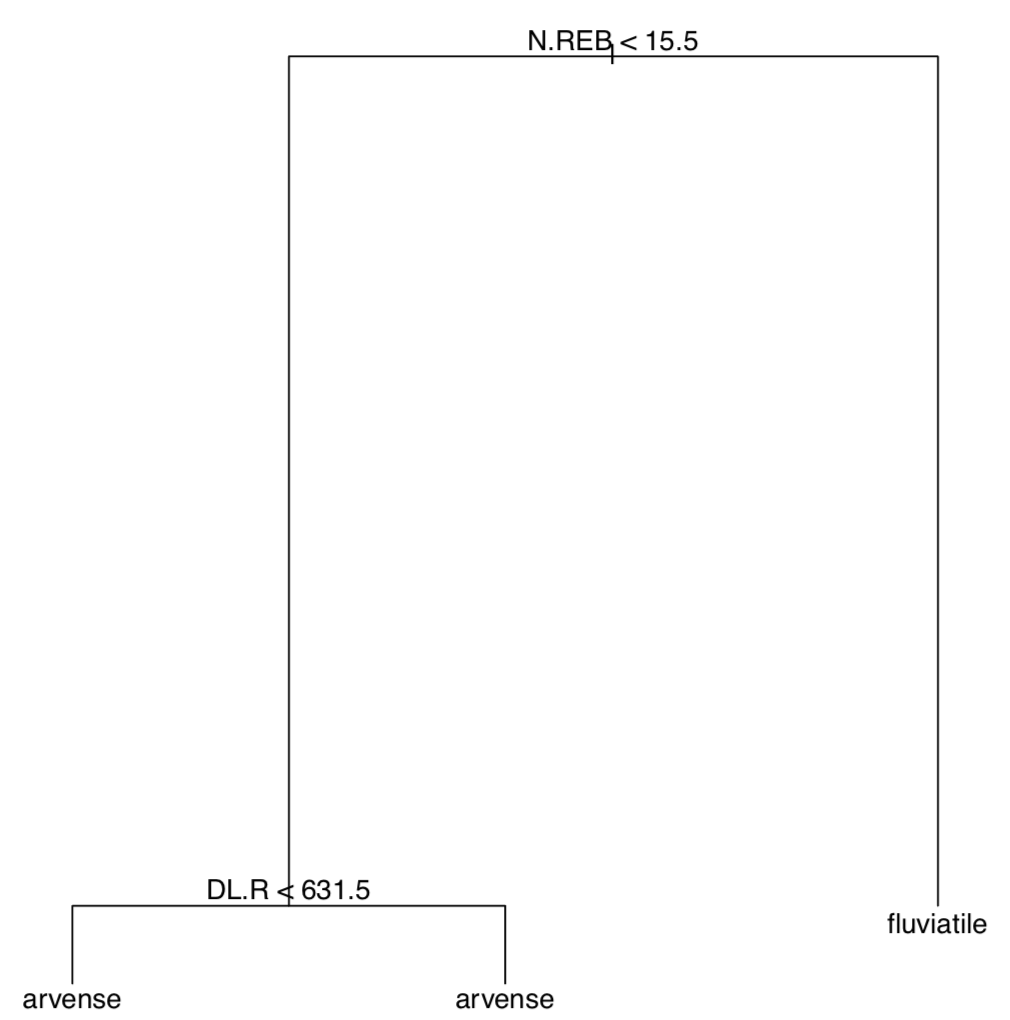
Respuesta al ejercicio de clasificación de especies vegetales de árboles. El árbol se explica por sí mismo pero tenemos que construirlo primero (Figura\(\PageIndex{3}\)):
Código\(\PageIndex{8}\) (R):
Respuesta a la pregunta de kubricks (Figura\(\PageIndex{3}\)). Esto es solo un plan, ya que aún necesitará realizar estos pasos individualmente:
1. Abra R, abra Excel o cualquier software de hoja de cálculo y cree el archivo de datos. Este archivo de datos debe ser la tabla donde las especies kubrick son filas y los caracteres son columnas (variables). Cada fila debe comenzar con un nombre de kubrick (es decir, letra), y cada columna debe tener un encabezado (nombre del carácter). Para los caracteres, son preferibles los nombres cortos en mayúsculas sin espacios.
La celda de Topleft podría permanecer vacía. En todas las demás celdas, debe haber 1 (carácter presente) o 0 (carácter ausente). Para el personaje, podrías usar “presencia de tallo” o “presencia de tres bocas”, o “habilidad para hacer fotosíntesis”, o algo parecido. Dado que hay 8 kubricks, se recomienda inventar\(N+1\) (en este caso, 9) caracteres.
2. Guarde su tabla como un archivo de texto, preferiblemente separado por tabulaciones (use enfoques descritos en el segundo capítulo), luego cárguela en R con read.table (.., h=true, fila.names=1).
3. Aplicar clustering jerárquico con el método distance aplicable para datos binarios (0/1), por ejemplo binario de dist () u otro método (como Jaccard) del vegan: :vegdist ().
4. Hacer el dendrograma con hclust () usando el algoritmo de clustering apropiado.
En el directorio de datos, hay un archivo de datos, kubricks.txt. Es solo un ejemplo por lo que no es necesariamente correcto y no contiene descripciones de caracteres. Dado que es bastante obvio cómo realizar la agrupación jerárquica (ver el ejemplo de “cerveza” anterior), presentamos a continuación otras dos posibilidades.
Primero, usamos MDS más el MST, árbol de expansión mínimo, el conjunto de líneas que muestran la ruta más corta conectando todos los objetos en la gráfica de ordenación (Figura\(\PageIndex{4}\)):
Código\(\PageIndex{9}\) (R):
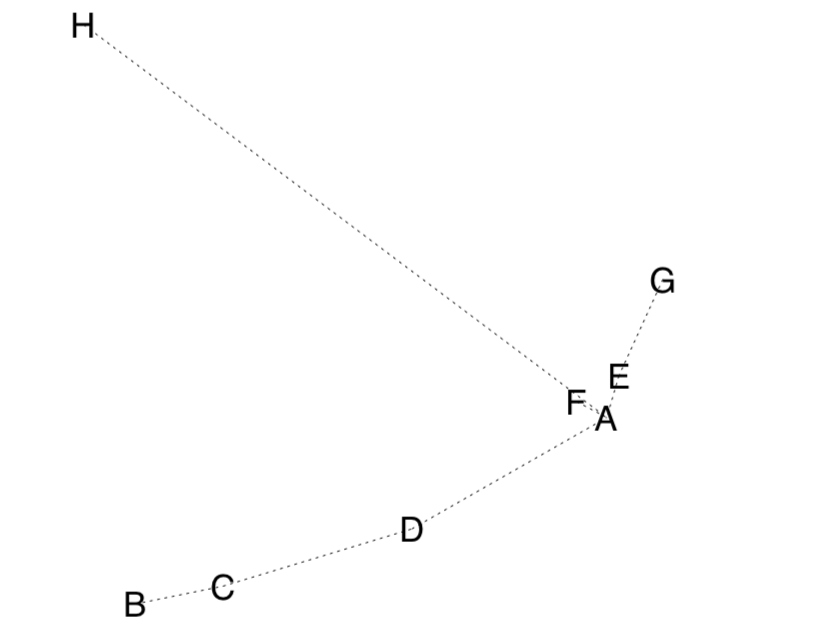
Segundo, podemos tomar en cuenta que los kubricks son objetos biológicos. Por lo tanto, con la ayuda de paquetes ape y phangorn podemos intentar construir el árbol de filogenia más parsimonioso (es decir, el más corto) para kubricks. Aceptemos que kubrick H es el grupo externo, el más primitivo:
Código\(\PageIndex{10}\) (R):
(Haz y revisa esta parcela tú mismo.)

