3.5: Ensayo de Epigenética
( \newcommand{\kernel}{\mathrm{null}\,}\)
- 1: Conocer diferentes tipos de ensayos epigenéticos.
- 2: Conocer cómo se utilizan diferentes ensayos epigenéticos cuando las células están expuestas a tóxicos o mutágenos.
5.1: Ensayo de metilación de ADN
Los ensayos de metilación del ADN son importantes para conocer la modificación epigenética que es una modificación heredable inducida por enzimas sin alteración de los pares de bases nucleotídicas. La transferencia de un grupo metil al carbono 5 en la citosina en un dinucleótido CpG ocurre en la metilación del ADN por ADN metiltransferasas (DNMT1, DNMT3A y DNMT3B). El alto nivel de metilación de la isla CpG del promotor da como resultado el silenciamiento génico. La técnica de inmunoprecipitación de ADN metilado (MediP) -chip se utiliza para el ensayo de metilación del ADN.
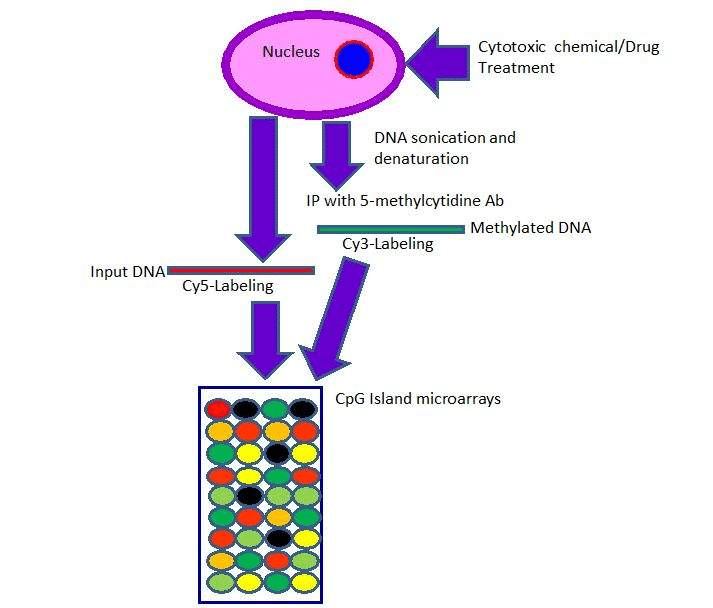
En resumen, el procedimiento de chip MEDIP se menciona de la siguiente manera. El ADN genómico es cizallado a fragmentos de bajo peso molecular (aproximadamente 400 pb) por sonicación. Luego, los ADN metilados se inmunoprecipitan con el anticuerpo anti-metil-citosina, y se amplifican con PCR, si el material fuente es menor. El ADN de entrada y metilado se etiquetan con colorantes fluorescentes Cy3 (verde) y Cy5 (rojo), se agrupan, se desnaturalizan y se hibridan con un portaobjetos de micromatriz que contiene todas las islas CpG humanas anotadas u otros diseños de micromatrices de genoma completo o promotor. Después se escanea la diapositiva usando un escáner y cada imagen se analiza con el software de análisis de imágenes (Figura 1).
5.2: Ensayo de modificación de histonas
Los ensayos de modificación de histonas son útiles para encontrar la modificación de proteínas histonas (por ejemplo, acetilación de lisina, metilación de lisina y arginina, fosforilación de serina y treonina, y ubiquitinación y sumoilación de lisina) que tienen papeles importantes en la herencia epigenética. El ensayo de inmunoprecipitación de cromatina (ChIP) seguido de hibridación a microarrays (ChIP-chip) (izquierda) o por secuenciación de alto rendimiento (ChIP-seq) (derecha) son técnicas poderosas para encontrar la modificación de histonas.
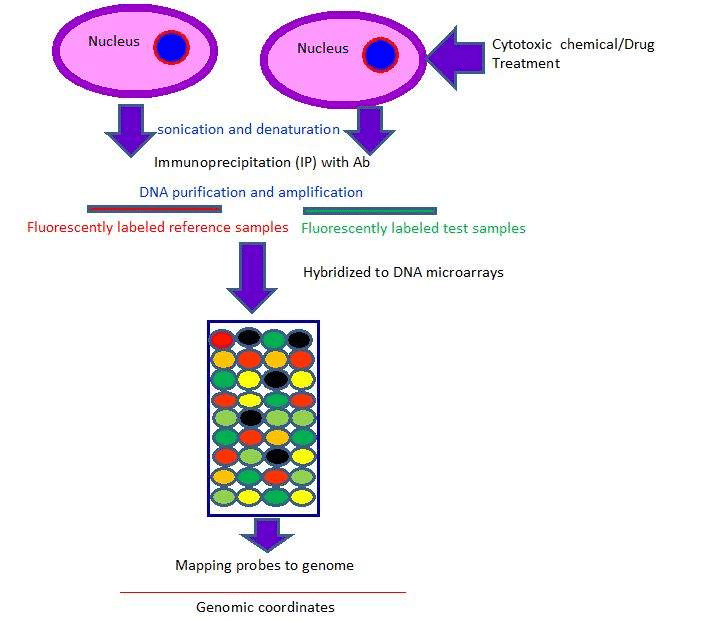
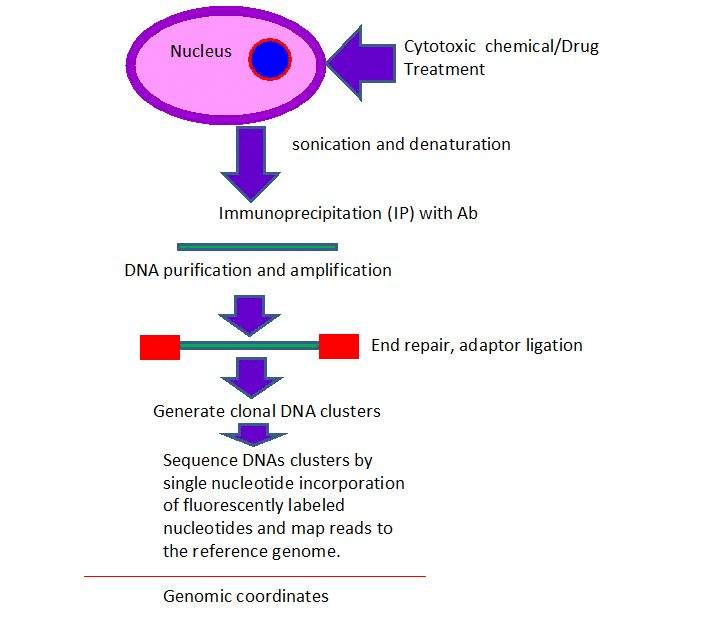
5.3: Ensayo de microARN
Los ensayos de microARN se utilizan para conocer los ARN no codificantes (17-25 nucleótidos) que se dirigen a los ARN mensajeros (ARNm) y descomponen los ARNm o se regulan por disminución a nivel de traducción en proteína. Casi el 60% de los genes codificantes de proteínas humanas están controlados por miARN y estos miARN están regulados epigenéticamente. Alrededor del 50% de los genes miARN están relacionados con islas CpG, las cuales pueden ser reprimidas por metilación epigenética. Otros miARN están controlados epigenéticamente por modificaciones de histonas o por metilación del ADN. La expresión de microARN se cuantifica mediante RT-PCR seguida de PCR cuantitativa (qPCR). Luego, los miARN se hibridan a micromatrices, portaobjetos o chips con sondas a cientos o miles de dianas de miARN. Los microARN pueden inventarse y perfilarse mediante métodos de secuenciación (secuenciación de microARN).
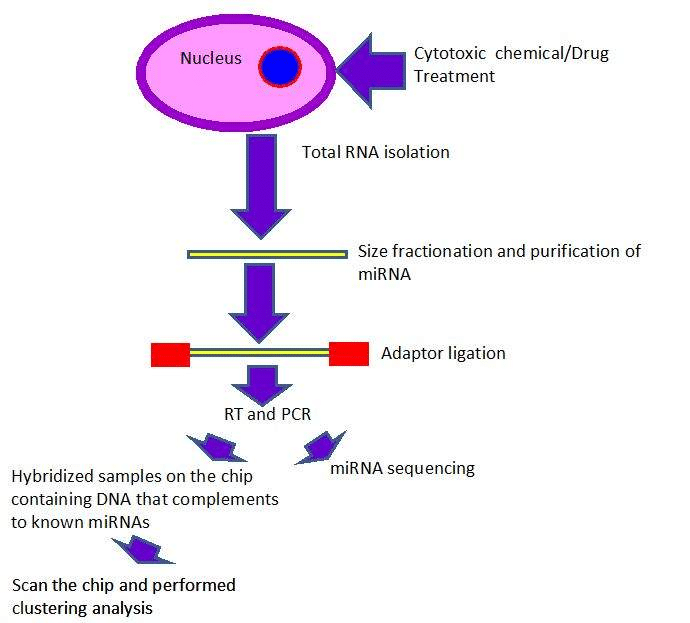
Tema 5: Puntos Clave
En esta sección, exploramos los siguientes puntos principales:
- 1: Diferentes tipos de ensayos epigenéticos.
- 2: Cómo se utilizan diferentes ensayos epigenéticos, a saber, el ensayo de metilación del ADN, el ensayo de modificación de histonas y el ensayo de microARN cuando las células se exponen a sustancias químicas o agentes tóxicos.
1. Los ensayos de metilación del ADN son importantes para conocer la modificación no epigenética.
Cierto
Falso
- Contestar
-
false
2. Se utilizan ensayos de microARN para conocer los ARN no codificantes. Estos ARN no codificantes son:
De 17 a 25 nucleótidos.
50 a 100 nucleótidos.
200 a 400 nucleótidos.
Ninguna de las anteriores.
- Contestar
-
De 17 a 25 nucleótidos.
3. Los ensayos de modificación de histonas son útiles para encontrar la modificación de proteínas histonas que tienen un papel importante en la herencia epigenética:
Cierto
Falso
- Contestar
-
true


