11.7: Uso de R para una Regresión Lineal Multivariada
- Page ID
- 69320
Para ilustrar cómo podemos usar R para completar una regresión lineal multivariada, use este enlace y guarde el archivo allSpec.csv en su directorio de trabajo. Los datos de este archivo constan de 80 filas y 642 columnas. Cada fila es una muestra independiente que contiene uno o más de los siguientes cationes de metales de transición: Cu 2 +, Co 2 +, Cr 3 + y Ni 2 +. Las primeras siete columnas proporcionan información sobre las muestras:
- un id de muestra (en la forma custd_1 para un único estándar de Cu 2 + o nicu_mix1 para una mezcla de Ni 2 + y Cu 2 +)
- una lista de los analitos en la muestra (en la forma cuco para una muestra que contiene Cu 2 + y Co 2 +)
- el número de analitos en la muestra (un número de 1 a 4 y etiquetado como dimensiones)
- la concentración molar de Cu 2 + en la muestra
- la concentración molar de Co 2 + en la muestra
- la concentración molar de Cr 3 + en la muestra
- la concentración molar de Ni 2 + en la muestra
Las columnas restantes contienen valores de absorbancia a 635 longitudes de onda entre 380.5 nm y 899.5 nm. Utilizaremos un subconjunto de estos datos que sea idéntico al utilizado para ilustrar un análisis de conglomerados y un análisis de componentes principales.
Primero, necesitamos leer los datos en R, lo que hacemos usando la función read.csv ()
spec_data <- read.csv (” allSpec.csv “, check.names = FALSO)
donde la opción check.names = FALSE anula el valor predeterminado de la función para no permitir que el nombre de una columna comience con un número. A continuación, crearemos objetos para mantener las concentraciones y absorbancias para soluciones estándar de Cu 2 +, Cr 3 + y Co 2 +, que son los tres analitos
wavelength_ids = seq (8, 642, 40)
abs_stds = spec_data [1:15, wavelength_ids]
conc_stds = data.frame (spec_data [1:15, 4], spec_data [1:15, 5], spec_data [1:15, 6])
abs_samples = spec_data [c (1, 6, 11, 21:25, 38:53), wavelength_ids]
donde wavelength_ids es un vector que identifica las 16 longitudes de onda igualmente espaciadas, abs_stds es una trama de datos que da los valores de absorbancia para 15 soluciones estándar de los tres analitos Cu 2 +, Cr 3 + y Co 2 + a las 16 longitudes de onda, conc_stds es un marco de datos que contiene las concentraciones de los tres analitos en las 15 soluciones estándar, y abs_samples es un marco de datos que contiene las absorbancias de las 24 muestras en las 16 longitudes de onda. Estos son los mismos datos utilizados para ilustrar el análisis de conglomerados y el análisis de componentes principales.
Para resolver para la\(\epsilon b\) matriz escribiremos y obtendremos la siguiente función que toma dos objetos, un marco de datos de valores de absorbancia y un marco de datos de concentraciones, y devuelve una matriz de\(\epsilon b\) valores.
findeb = función (abs, conc) {
abs.m = as.matrix (abs)
conc.m = as.matriz (conc)
ct = t (conc.m)
ctc = ct %*% conc.m
invctc = resolver (ctc)
eb = invctc %*% ct %*% abs.m
salida = eb
invisible ( salida)
}
Pasar abs_stds y conc_stds a la función
eb_pred = findeb (abs_stds, conc_stds)
devuelve los valores predichos para\(\epsilon b\) que componen nuestra calibración. Como vemos a continuación, una gráfica de los\(\epsilon b\) valores para Cu 2 + tiene la misma forma que una gráfica de los valores de absorbancia para uno de los estándares de Cu 2 +.
longitudes de onda = como.numeric (colnames (spec_data [8:642]))
old.par = par (mfrow = c (2,1))
plot (x = longitudes de onda [wavelength_ids], y = eb_pred [1,], type = “b”,
xlab = “longitud de onda (nm)”, ylab = “eb”, lwd = 2, col = “azul”)
plot (x = longitudes de onda, y = spec_ data [1, 8:642], type = “l”,
xlab = “longitud de onda (nm)”, ylab = “absorbancia”, lwd = 2, col = “azul”)
par (old.par)
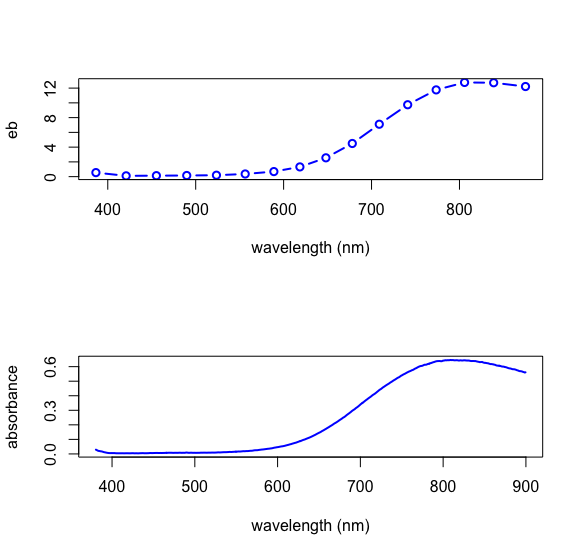
Una vez completada la calibración, podemos determinar las concentraciones de los tres analitos en las 24 muestras utilizando la siguiente función, que toma como entradas el marco de datos de valores de absobancia y la\(\epsilon b\) matriz devuelta por la función findeb
findconc = función (abs, eb) {
abs.m = as.matrix (abs)
eb.m = as.matrix (eb)
ebt = t (eb.m)
ebebt = eb %*% ebt
invebebt = resolver (ebebt)
pred_conc = redondo (abs.m %*% ebt %*% invebebt, dígitos = 5)
salida = pred_conc
invisible (salida)
}
pred_conc = findconc (abs_samples, eb_pred)
Para determinar el error en las concentraciones predichas, primero extraemos las concentraciones reales del conjunto de datos original como marco de datos, ajustando los nombres de las columnas para mayor claridad.
real_conc = data.frame (spec_data [c (1, 6, 11, 21:25, 38:53), 4],
spec_data [c (1, 6, 11, 21:25, 38:53), 5],
spec_data [c (1, 6, 11, 21:25, 38:53), 6])
colnames (real_conc) = c (“cobre”, “cobalto”, “cromo”)
y determinar la diferencia entre las concentraciones reales y las concentraciones predichas
conc_error = real_conc - pred_conc
Finalmente, podemos reportar el error medio, la desviación estándar y el intervalo de confianza del 95% para cada analito.
media = aplicar (conc_error, 2, media)
redondo (medias, dígitos = 6)
cobre cobalto cromo
-0.000280 -0.000153 -0.000210
sds = aplicar (conc_error, 2, sd)
redondo (sds, dígitos = 6)
cobre cobalto cromo
0.001037 0.000811 0.000688
conf.it = abs (qt (0.05/2, 20)) * sds
redondo (conf.it, dígitos = 6)
cobre cobalto cromo
0.002163 0.001693 0.001434
Comparado con los intervalos de concentraciones para los tres analitos en las 24 muestras
rango (real_conc$cobre)
[1] 0.00 0.05
rango (real_conc$cobalto)
[1] 0.0 0.1
rango (real_conc$cromo)
[1] 0.0000 0.0375
los errores medios y los intervalos de confianza son lo suficientemente pequeños como para tener confianza en los resultados.

