11.6: Uso de R para un análisis de componentes principales
- Page ID
- 69313
Para ilustrar cómo podemos usar R para completar un análisis de clúster: use este enlace y guarde el archivo allSpec.csv en su directorio de trabajo. Los datos de este archivo constan de 80 filas y 642 columnas. Cada fila es una muestra independiente que contiene uno o más de los siguientes cationes de metales de transición: Cu 2 +, Co 2 +, Cr 3 + y Ni 2 +. Las primeras siete columnas proporcionan información sobre las muestras:
- un id de muestra (en la forma custd_1 para un único estándar de Cu 2 + o nicu_mix1 para una mezcla de Ni 2 + y Cu 2 +)
- una lista de los analitos en la muestra (en la forma cuco para una muestra que contiene Cu 2 + y Co 2 +)
- el número de analitos en la muestra (un número de 1 a 4 y etiquetado como dimensiones)
- la concentración molar de Cu 2 + en la muestra
- la concentración molar de Co 2 + en la muestra
- la concentración molar de Cr 3 + en la muestra
- la concentración molar de Ni 2 + en la muestra
Las columnas restantes contienen valores de absorbancia a 635 longitudes de onda entre 380.5 nm y 899.5 nm.
Primero, necesitamos leer los datos en R, lo que hacemos usando la función read.csv ()
spec_data <- read.csv (” allSpec.csv “, check.names = FALSO)
donde la opción check.names = FALSE anula el valor predeterminado de la función para no permitir que el nombre de una columna comience con un número. A continuación, crearemos un subconjunto de este gran conjunto de datos para trabajar con
id_longitud de onda = seq (8, 642, 40)
id_muestra = c (1, 6, 11, 21:25, 38:53)
pca_data = spec_data [sample_ids, wavelength_ids]
donde wavelength_ids es un vector que identifica las 16 longitudes de onda igualmente espaciadas, sample_ids es un vector que identifica las 24 muestras que contienen uno o más de los cationes Cu 2 +, Co 2 + y Cr 3 +, y cluster_data es una trama de datos que contiene los valores de absorbancia para estas 24 muestras en estas 16 longitudes de onda.
Para completar el análisis de componentes principales utilizaremos la función prcomp () de R, que toma la forma general
prcomp (objeto, centro, escala)
donde object es un marco de datos o matriz que contiene nuestros datos, y el centro y la escala son valores lógicos que indican si primero debemos centrar y escalar los datos antes de completar el análisis. Cuando centramos y escalamos nuestros datos cada variable (en este caso, la absorbancia a cada longitud de onda) se ajusta para que su media sea cero y su varianza sea una. Esto tiene el efecto de colocar todas las variables en una escala común, lo que asegura que cualquier diferencia en la magnitud relativa de las variables no afecte al análisis de componentes principales.
pca_results = prcomp (pca_data, center = VERDADERO, escala = VERDADERO)
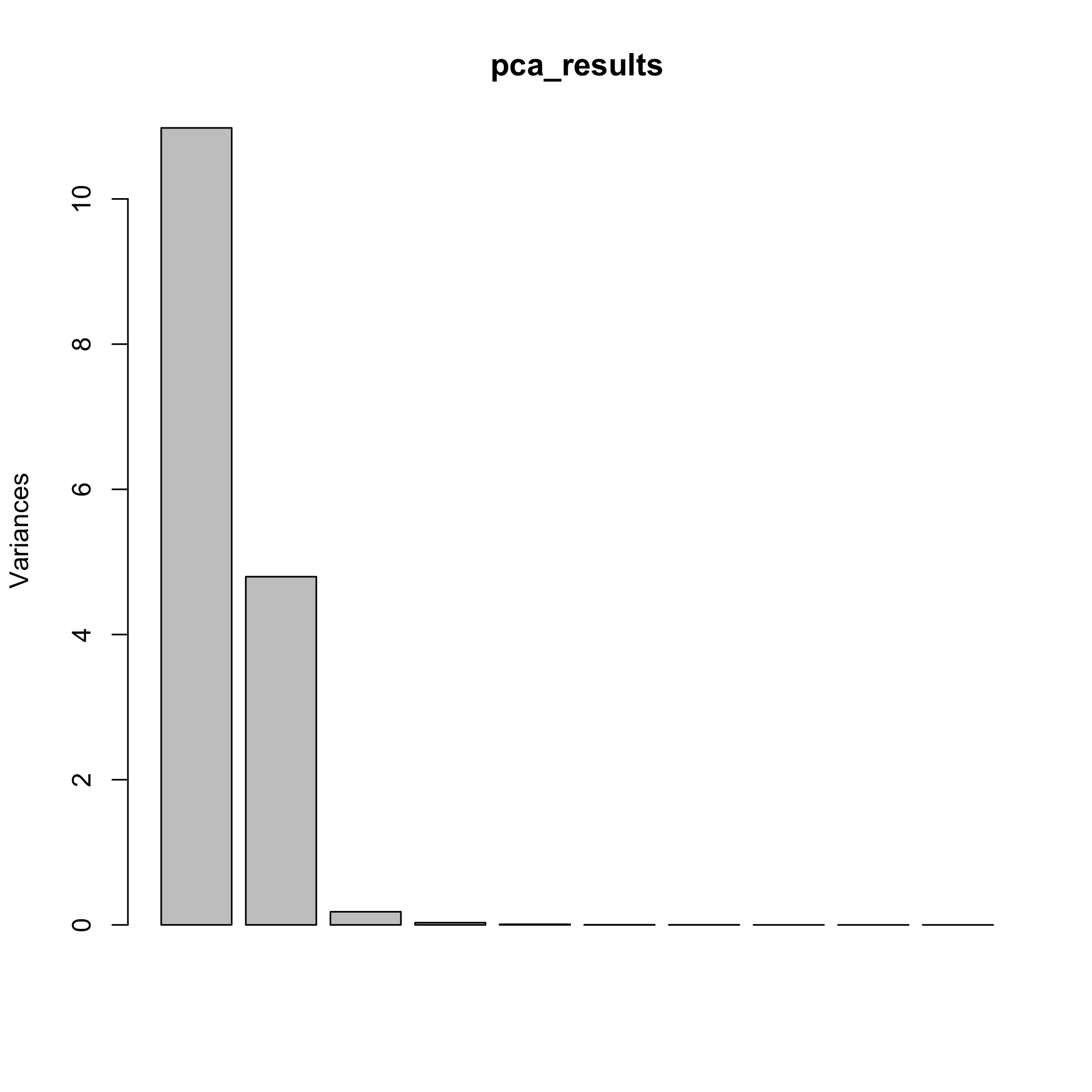
La función prcomp () devuelve una variedad de información que podemos usar para examinar los resultados, incluyendo la desviación estándar para cada componente principal, sdev, una matriz con las cargas, rotación, una matriz con las puntuaciones, x, y los valores utilizados para centrar y escalar los datos originales. La función summary (), por ejemplo, devuelve las desviaciones estándar para y la proporción de la varianza general explicada por cada componente principal, y la proporción acumulada de varianza explicada por los componentes principales.
resumen (pca_results)
Importancia de los componentes:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9
Desviación estándar 3.3134 2.1901 0.42561 0.17585 0.09384 0.04607 0.04026 0.01253 0.01049
Proporción de varianza 0.6862 0.2998 0.01132 0.00193 0.00055 0.00013 0.00010 0.00001 0.00001
Proporción Acumulada 0.6862 0.9859 0.99725 0.99919 0.99974 0.99987 0.99997 0.99998 0.99999
PC10 PC11 PC12 PC13 PC14 PC15 PC16
Desviación estándar 0.009211 0.007084 0.004478 0.00416 0.003039 0.002377 0.001504
Proporción de varianza 0.000010 0.000000 0.000000 0.00000 0.000000 0.000000 0.000000
Proporción Acumulada 0.999990 1.000000 1.000000 1.00000 1.000000 1.000000
También podemos examinar la varianza de cada componente principal (el cuadrado de su desviación estándar) en forma de gráfica de barras pasando los resultados del análisis de componentes principales a la función plot ().
plot (pca_results)
Como se señaló anteriormente, las 24 muestras incluyen uno, dos o tres de los cationes Cu 2 +, Co 2 + y Cr 3 +, lo que es consistente con nuestros resultados si se elaboran soluciones individuales combinando alícuotas de soluciones madre de Cu. 2 +, Co 2 + y Cr 3 + y diluir a un volumen común. En este caso, el volumen de solución madre para un catión pone límites a los volúmenes de los otros cationes de tal manera que una mezcla de tres componentes esencialmente tiene dos variables independientes.
Para examinar las puntuaciones para el análisis del componente principal, pasamos las puntuaciones a la función plot (), aquí usando pch = 19 para mostrarlas como puntos rellenos.
plot (pca_results$x, pch = 19)
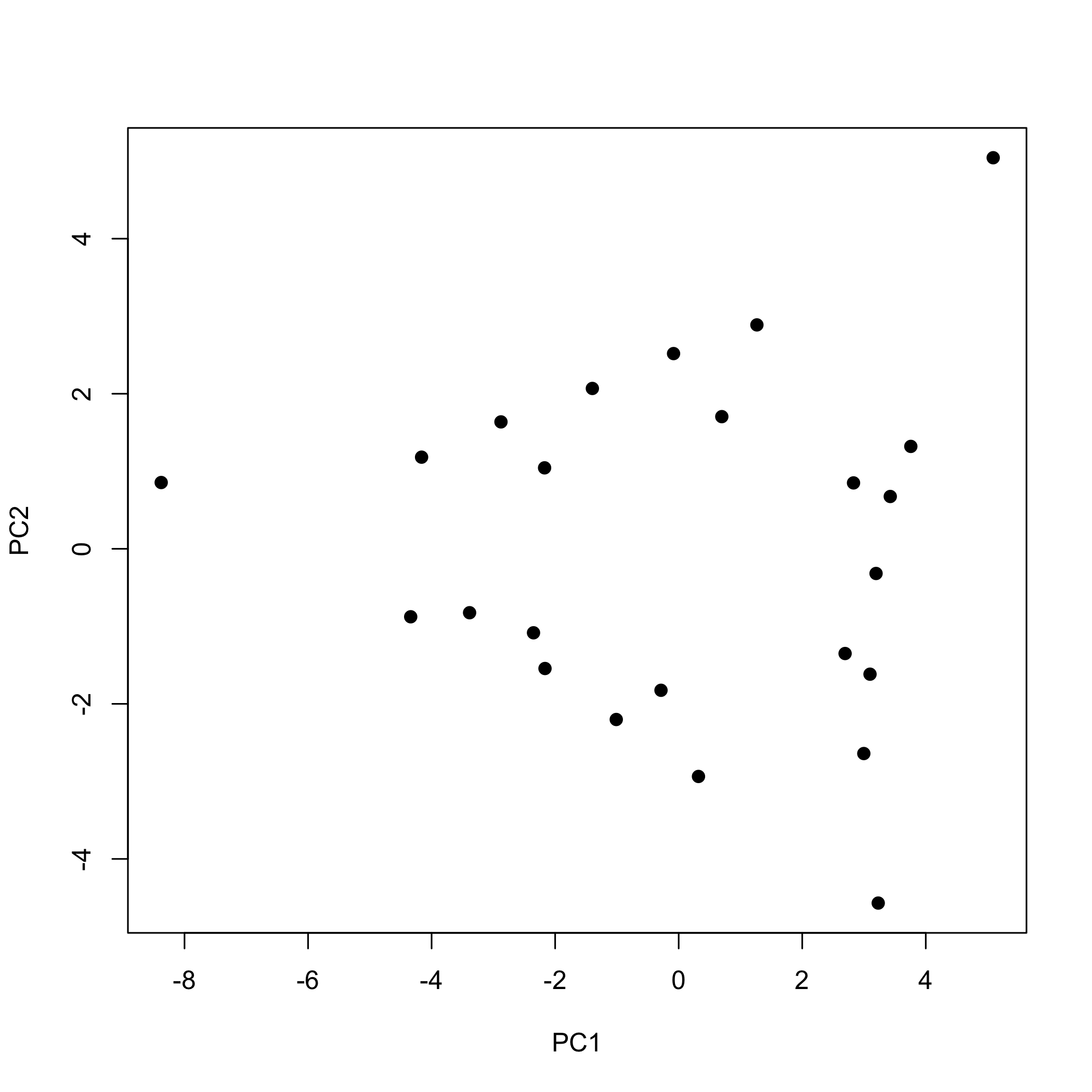
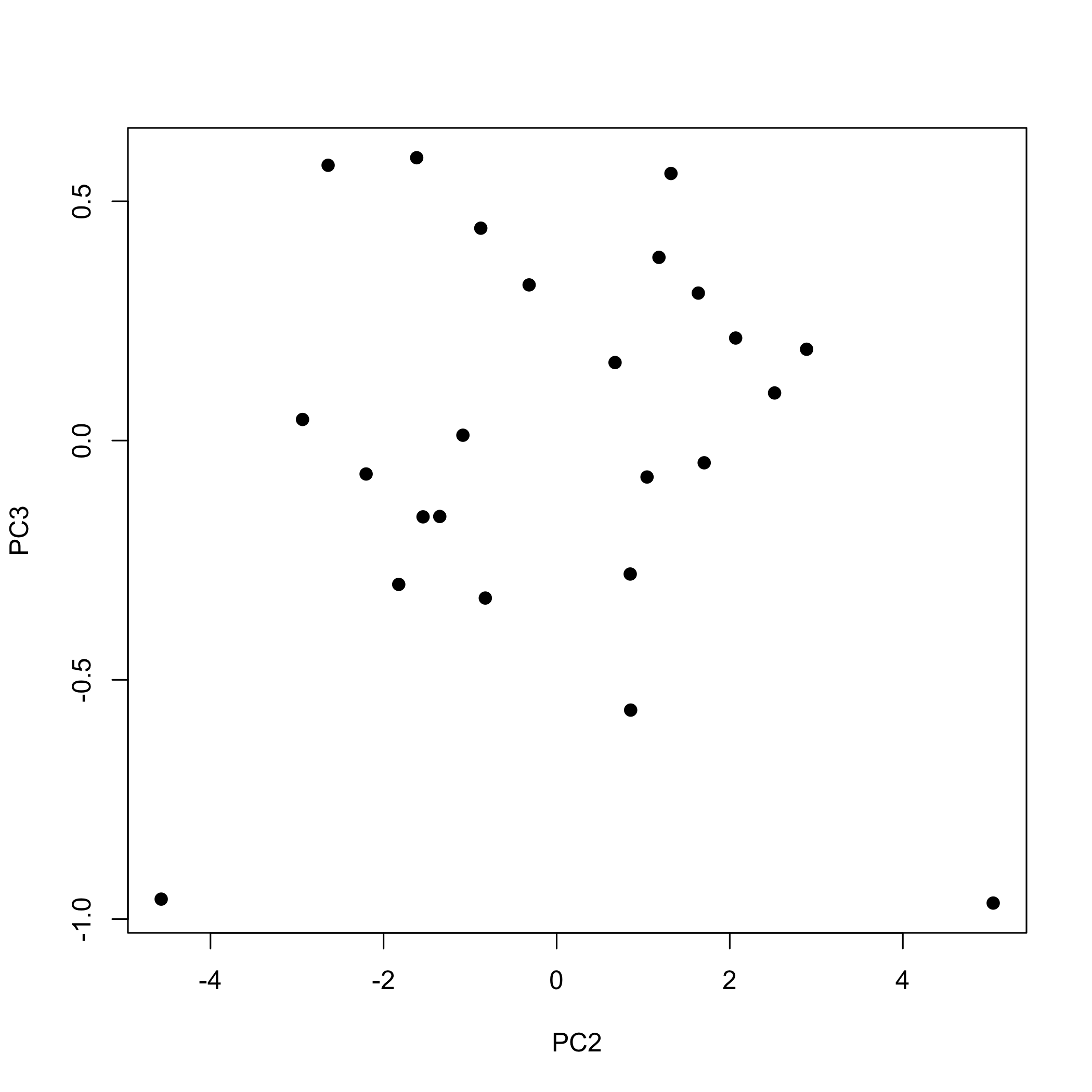
Por defecto, la función plot () muestra los valores de los dos primeros componentes principales, con el primero (PC1) colocado en el eje x y el segundo (PC2) colocado en el eje y. Si queremos examinar otros componentes principales, entonces debemos especificarlos al llamar a la función plot (); el siguiente comando, por ejemplo, usa las puntuaciones para el segundo y el tercer componente principal.
plot (x = pca_results$x [,2], y = pca_results$x [,3], pch = 19, xlab = “PC2", ylab = “PC3")
Si queremos mostrar los tres primeros componentes principales usando la misma gráfica, entonces podemos usar la función scatter3D () del paquete plot3D, que toma la forma general
biblioteca (Plot3D)
Scatter3D (x = pca_results$x [,1], y = pca_results$x [,2], z = pca_results$x [,3], pch = 19, type = “h”, theta = 25, phi = 20, ticktype = “detallado”, colvar = NULL)
donde usamos la función library () para cargar el paquete en nuestra sesión R (nota: esto supone que has instalado el paquete Plot3D). El tipo de opción = “h” deja caer una línea horizontal desde cada punto hacia abajo hasta el plano para PC1 y PC2, lo que nos ayuda a orientar los puntos en el espacio. Por defecto, la gráfica usa color para mostrar cada valor de puntos del tercer componente principal (mostrado en el eje z); aquí establecemos colvar = NULL para mostrar todos los puntos usando el mismo color.
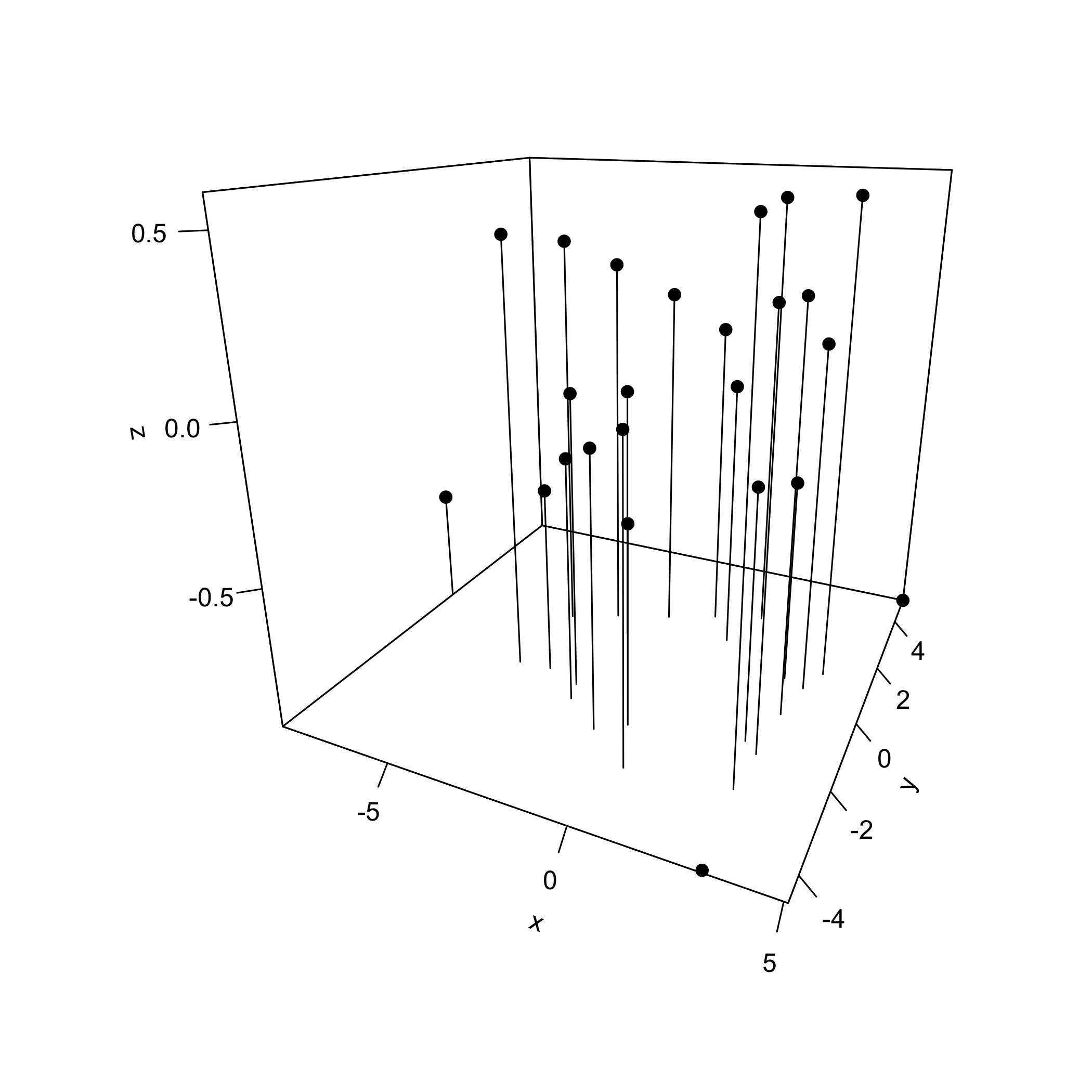
Aunque las gráficas no se muestran aquí, podemos usar los mismos comandos, reemplazando x con rotación, para mostrar las cargas.
plot (pca_results$rotación, pch = 19)
plot (x = pca_results$rotación [,2], y = pca_results$rotación [,3], pch = 19, xlab = “PC2", ylab = “PC3")
Scatter3D (x = pca_results$rotation [,1], y = pca_results$rotation [,2], z = pca_results$rotation [,3], pch = 19, type = “h”, theta = 25, phi = 20, ticktype = “detallado”, colvar = NULL)
Otra forma de ver los resultados de un análisis de componentes principales es mostrar las puntuaciones y las cargas en la misma parcela, lo que podemos hacer usando la función biplot ().
biplot (pca_results, cex = c (2, 0.6), xlabs = rep (“•”, 24))
donde la opción xlabs = rep (“•”, 24) anula el valor predeterminado de la función para mostrar las puntuaciones como números, reemplazándolos por puntos, y cex = c (2, 0.6) se usa para aumentar el tamaño de los puntos y disminuir el tamaño de las etiquetas para las cargas.
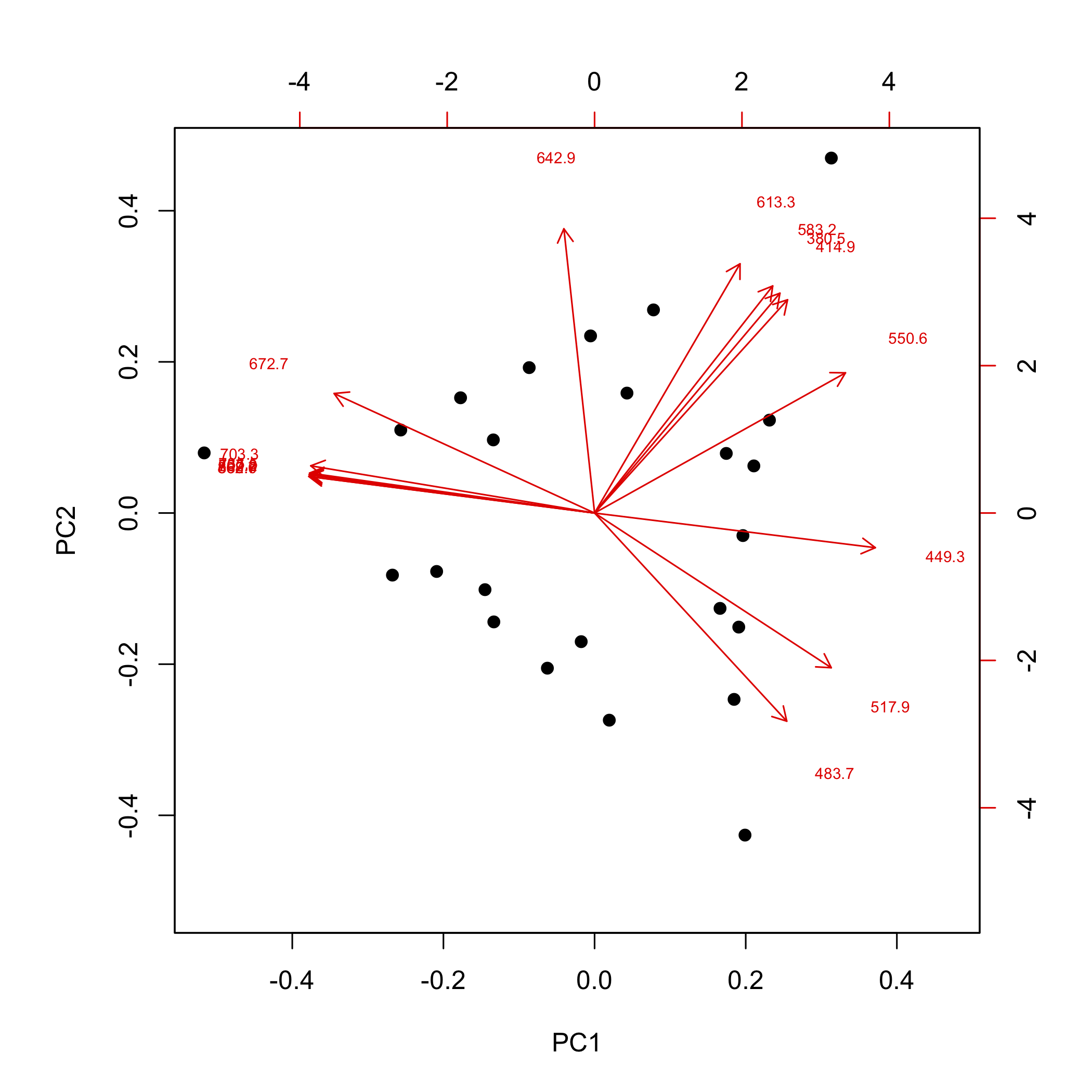
En este biplot, las puntuaciones se muestran como puntos y las cargas se muestran como flechas que comienzan en el origen y apuntan hacia las cargas individuales, las cuales son indicadas por las longitudes de onda asociadas a las cargas. Para este conjunto de datos, las puntuaciones y cargas que se ubican conjuntamente entre sí representan muestras y longitudes de onda que están fuertemente correlacionadas entre sí. Por ejemplo, la muestra cuya puntuación está en la esquina superior derecha está fuertemente asociada con la absorbancia de luz con longitudes de onda de 613.3 nm, 583.2 nm, 380.5 nm y 414.9 nm.
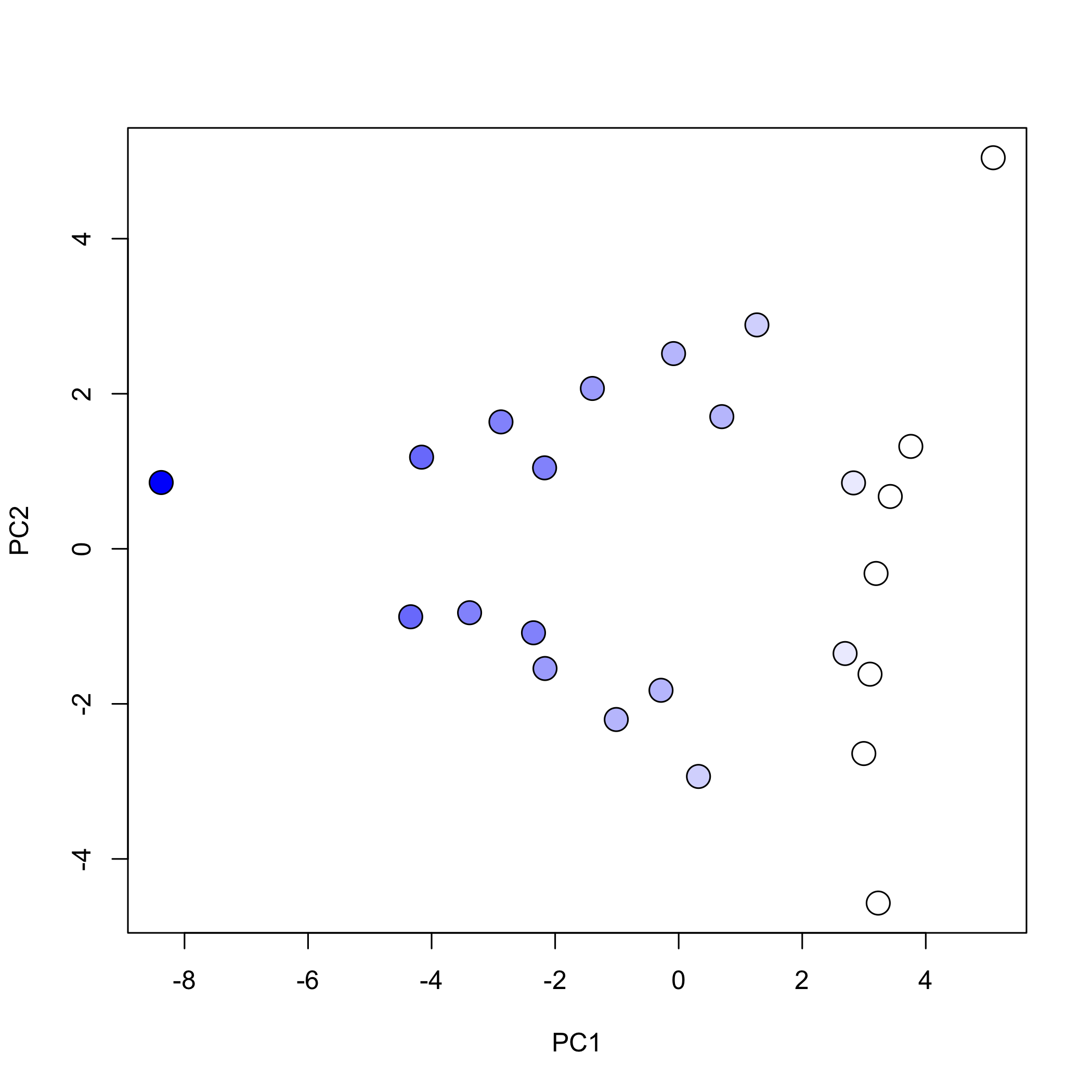
Finalmente, podemos usar el color para resaltar características de nuestro conjunto de datos. Por ejemplo, las siguientes líneas de código crean una gráfica de puntuaciones que utiliza una paleta de colores para indicar la concentración relativa de Cu 2 + en la muestra.
cu_palette = colorRampPalette (c (“blanco”, “azul”))
cu_color = cu_pallete (50) [como.numeric (cut (spec_data$conccu [sample_ids], breaks = 50))]
La función ColorRampPalette () toma un vector de colores, en este caso blanco y azul, y devuelve una función que podemos usar para crear una paleta de colores que va desde el blanco puro hasta el azul puro. Luego usamos esta función para crear 50 tonos de blanco y azul
cu_palette (50)
[1] "#FFFFFF" "#F9F9FF" "#F4F4FF" "#EFEFFF" "#EAEAFF" "#E4E4FF" "#DFDFFF" "#DADAFF"
[9] "#D5D5FF" "#D0D0FF" "#CACAFF" "#C5C5FF" "#C0C0FF" "#BBBBFF" "#B6B6FF" "#B0B0FF"
[17] "#ABABFF" "#A6A6FF" "#A1A1FF" "#9C9CFF" "#9696FF" "#9191FF" "#8C8CFF" "#8787FF"
[25] "#8282FF" "#7C7CFF" "#7777FF" "#7272FF" "#6D6DFF" "#6868FF" "#6262FF" "#5D5DFF"
[33] "#5858FF" "#5353FF" "#4E4EFF" "#4848FF" "#4343FF" "#3E3EFF" "#3939FF" "#3434FF"
[41] "#2E2EFF" "#2929FF" "#2424FF" "#1F1FFF" "#1A1AFF" "#1414FF" "#0F0FFF" "#0A0AFF"
[49] "#0505FF" "#0000FF"
donde #FFFFFF es el código hexadecimal para el blanco puro y #0000FF es el código hexadecimal para el azul puro. La última parte de esta línea de código
cu_color = cu_pallete (50) [como.numeric (cut (spec_data$conccu [sample_ids], breaks = 50))]
recupera las concentraciones de cobre en cada una de nuestras 24 muestras y asigna un código hexadecimal para un tono de azul que indica la concentración relativa de cobre en la muestra. Aquí vemos que la primera muestra tiene un código hexadecimal de #0000FF para azul puro, lo que significa que esta muestra tiene la mayor concentración de cobre y las muestras 2—8 tienen códigos hexademicales de #FFFFFF para blanco puro, lo que significa que estas muestras no contienen ningún cobre.
cu_color
[1] "#0000FF" "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF"
[9] "#D0D0FF" "#B6B6FF" "#9C9CFF" "#8282FF" "#6868FF" "#D0D0FF" "#B6B6FF" "#9C9CFF"
[17] "#8282FF" "#6868FF" "#EAEAFF" "#EAEAFF" "#B6B6FF" "#B6B6FF" "#8282FF" "#8282FF"
Finalmente, creamos la gráfica de partituras, usando pch = 21 para un círculo abierto cuyo color de fondo designamos usando bg = cu_color y donde usamos cex = 2 para aumentar el tamaño de los puntos.
plot (pca_results$x, pch = 21, bg = cu_color, cex = 2)