8.2: Proteínas
- Page ID
- 80614
Las proteínas son grandes moléculas biológicas que tienen pesos moleculares que van desde las mil hasta los millones. Los humanos tienen alrededor de 24,000 proteínas diferentes que catalizan reacciones químicas, reconocen moléculas extrañas y patógenos, permiten el movimiento celular y del organismo, y regulan la respuesta celular, incluyendo la división celular y la muerte.
Las proteínas son polímeros que consisten en mono meros llamados aminoácidos. Hay veinte aminoácidos diferentes de origen natural que difieren en uno de los cuatro grupos conectados a un átomo de carbono central. En un aminoácido, el carbono central (alfa) tiene un grupo amina (RNH 2, RNH 3 +), un grupo ácido carboxílico (RCOOH, RCOO -), un hidrógeno (H) y uno de veinte grupos R diferentes (también llamados cadenas laterales) unidos a él.
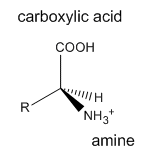
Los grupos R se clasifican como generalmente no polares, con carga polar o no cargados polares. El aminoácido más pequeño es la glicina (Gly) que tiene un átomo de hidrógeno como su grupo R. Todos los otros 19 aminoácidos naturales tienen un estereocentro (en el carbono que contiene los grupos amina y carboxilo) y pueden existir como dos posibles enantiómeros; solo el enantiómero L ocurre en las proteínas. Todos los aminoácidos en las proteínas tienen la configuración absoluta mostrada anteriormente. A excepción del aminoácido cisteína (Cys) con un -CH 2 SH para un grupo R (que pasa a tener un estereocentro R), todos los aminoácidos restantes que se encuentran en las proteínas tienen un estereocentro S.
Los aminoácidos forman polímeros cuando un grupo amino de un aminoácido se une covalentemente al carbono carbonilo (C=O) del grupo carboxilo del siguiente aminoácido. El vínculo resultante entre los aminoácidos es un enlace amida que los bioquímicos llaman enlace peptídico. En esta reacción, se libera agua (condensación). En una reacción inversa, el enlace peptídico puede ser escindido por agua (hidrólisis). Cuando dos aminoácidos se unen para formar un enlace amida, la estructura resultante se llama dipéptido. Asimismo, podemos tener tripéptidos, tetrapéptidos y otros polipéptidos. En algún momento, cuando la estructura es lo suficientemente larga, se le llama proteína. Hay muchas formas diferentes de representar la estructura de un polipéptido o proteína. Cada uno muestra diferentes cantidades de información. A continuación se muestra un heptapéptido, ácido aspártico-lisina-glutamina-histidina-cisteína-arginina-fenilalanina. Cada aminoácido se denota por un código de tres letras (Asp-Lys-Gln-His-Cys-Arg-Phe).
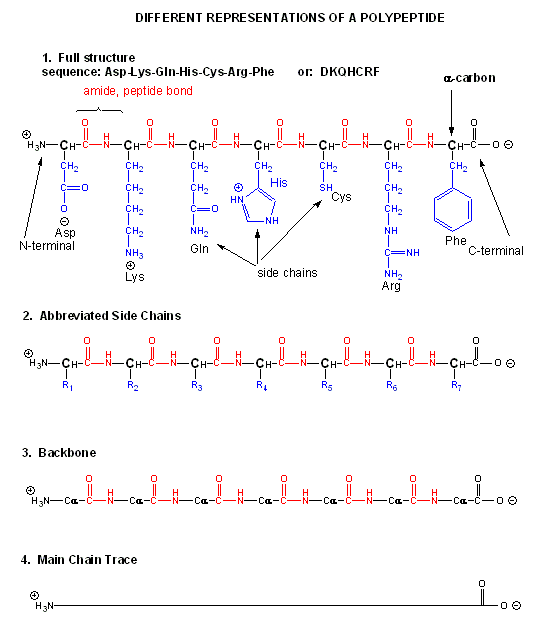
Observe que la cadena proteica tiene un principio (un extremo N con un grupo amino) y un extremo (un extremo C con un grupo carboxilo). También tenga en cuenta que cada átomo en la cadena principal tiene una ligera carga derivada de la presencia de los átomos electronegativos O y N. De ahí que la cadena principal sea polar. Los grupos R en cada aminoácido en el péptido también se denominan cadenas laterales.
La secuencia lineal real de aminoácidos que componen una proteína se llama su estructura primaria (1 o). Tanto la secuencia de aminoácidos como la longitud total de la cadena difieren de una proteína a otra. Solo por un octapéptido, hay más de 25 mil millones de posibles arreglos diferentes de aminoácidos. De ahí que la diversidad de posibles proteínas sea enorme.
La mayoría de las proteínas no forman una estructura alargada como lo implican las estructuras extendidas mostradas anteriormente. Más bien colapsan sobre sí mismos para formar estructuras compactas, en su mayoría globulares (aproximadamente esféricas). Lo hacen ya que los grupos locales y distantes de la cadena se atraen entre sí a través de IMF que ahora se ejercen dentro de la proteína grande y no entre diferentes proteínas. ¿Qué tipos de IMF están involucrados?
Para simplificar el proceso, consideremos primero solo la columna vertebral polar sin las cadenas laterales. La cadena principal puede formar claramente enlaces de hidrógeno consigo misma y con el agua. Si los enlaces de hidrógeno están entre la amida H (un enlace de hidrógeno “donante” y un carbonilo O (un “aceptor” de enlace de hidrógeno) se puede formar un número fijo de aminoácidos distantes de la amida H, se pueden formar estructuras secundarias regulares y repetitivas (2 o) llamadas hélice. Una hélice especialmente prevalente, la hélice alfa, se forma dentro de un tramo corto de aminoácidos cuando la amida H de un aminoácido (dado el número i th) en la cadena principal forma un enlace de hidrógeno al carbonilo C cuatro aminoácidos en la secuencia proteica (i th + 4). Hay 3.6 aminoácidos/giro de la hélice alfa.
Hélice Alfa (líneas amarillas punteadas representan enlaces de hidrógeno)
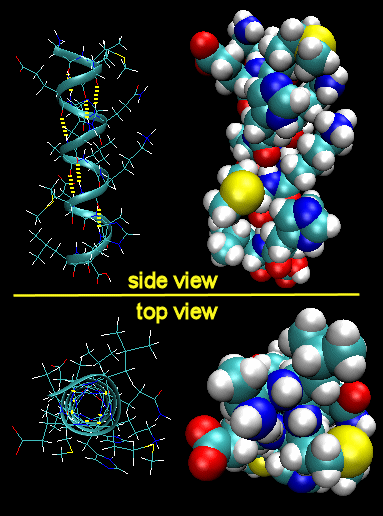
Tenga en cuenta que todas las cadenas laterales (grupos R) están apuntando lejos del eje de hélice. Evidencia del modelo de relleno de espacio, no hay abertura en la hélice mientras miras hacia abajo del eje porque los átomos reales están densamente empaquetados. El rastro de una hélice alfa en una proteína suele estar representado por una cinta rizada roja o morada.
 Jmol: Una hélice aislada de una Proteína Anticongelante Jmol14 (Java) | JSmol (HTML5)
Jmol: Una hélice aislada de una Proteína Anticongelante Jmol14 (Java) | JSmol (HTML5)Las cadenas/láminas beta, otro tipo de estructura secundaria, también se producen cuando se forman enlaces H entre tramos cortos adyacentes de aminoácidos en los que la cadena principal de los tramos cortos discurre ya sea en la misma dirección N-C (cadenas beta paralelas) o en direcciones opuestas (hebras beta antiparalelas). Los enlaces de hidrógeno en las estructuras secundarias están todos entre los átomos de la cadena principal en la cadena principal, no entre las cadenas laterales. La estructura beta generalmente se representa en forma de “caricatura” por cintas planas amarillas con una flecha que muestra la dirección de la cadena principal de la proteína desde el extremo N a la dirección del extremo C.
Hebras Beta Antiparalelas (las líneas amarillas representan enlaces de hidrógeno)
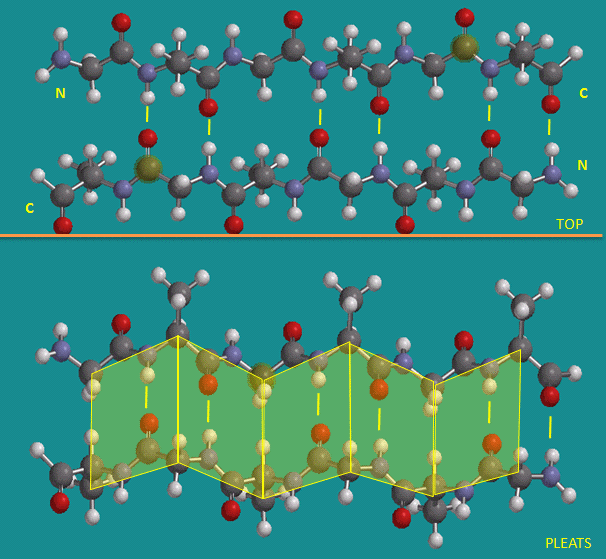
Filamentos beta paralelos (las líneas amarillas representan enlaces de hidrógeno)
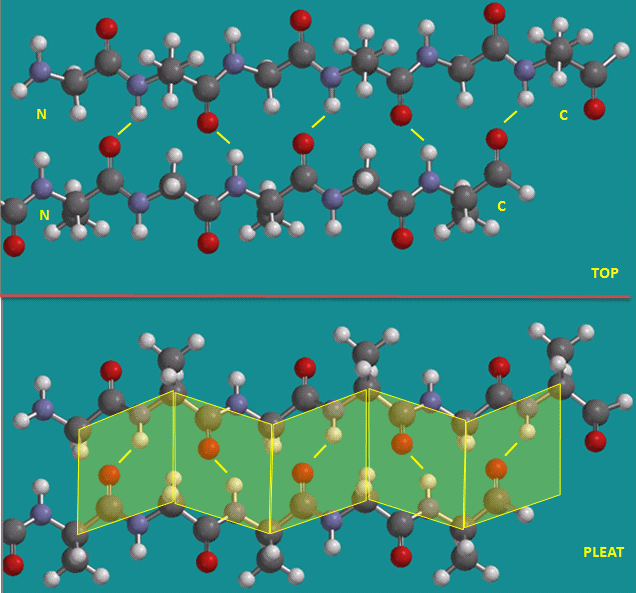
Las cadenas laterales en la lámina beta son perpendiculares al plano de la lámina, extendiéndose desde el plano en lados alternos. Las láminas paralelas distribuyen característicamente cadenas laterales no polares (o hidrófobas) en ambos lados de la lámina, mientras que las láminas antiparalelas suelen estar dispuestas con todos los residuos hidrófobos en un lado. Esto requiere una alternancia de cadenas laterales hidrófilas e hidrófobas en la secuencia primaria. Las láminas antiparalelas se encuentran en la seda con las hojas que corren paralelas a las fibras de seda. La siguiente repetición se encuentra en la secuencia primaria: (Ser-Gly-Ala-Gly) n), con Gly señalando desde una cara y Ser o Ala desde la otra.
 Jmol: Hoja beta retorcida de proteína de unión a arabinosa Jmol14 (Java) | JSmol (HTML5)
Jmol: Hoja beta retorcida de proteína de unión a arabinosa Jmol14 (Java) | JSmol (HTML5)El plegamiento de proteínas está determinado por mucho más que la formación de enlaces de hidrógeno entre donantes y aceptores de la cadena principal. Debemos considerar los efectos de los 20 grupos R diferentes (cadenas laterales) que complican el proceso de plegado. Una proteína finalmente se pliega en el espacio para formar una forma 3D única, que generalmente contiene algunas hélices alfa y láminas beta. La estructura general 3 D se denomina estructura terciaria (3 o) de la proteína. La estructura 3D de una proteína determina la función de la proteína. La forma de la proteína y las características de carga superficial determinan qué moléculas, tanto pequeñas como grandes, se unen a la proteína.
Aquí hay algunos modelos de proteínas que muestran estructuras secundarias y terciarias.
 Jmol: Met-mioglobina - predominantemente proteína alfa-helicoidal Jmol14 (Java) | JSmol (HTML5)
Jmol: Met-mioglobina - predominantemente proteína alfa-helicoidal Jmol14 (Java) | JSmol (HTML5) Jmol: Superóxido dismutasa - predominantemente estructura beta Jmol14 (Java) | JSmol (HTML5)
Jmol: Superóxido dismutasa - predominantemente estructura beta Jmol14 (Java) | JSmol (HTML5) Jmol: Triosa Fosfato Iomerasa - tiene estructura secundaria alfa y beta Jmol14 (Java) | JSmol (HTML5)
Jmol: Triosa Fosfato Iomerasa - tiene estructura secundaria alfa y beta Jmol14 (Java) | JSmol (HTML5)La estructura de las proteínas es mucho más complicada que las micelas y bicapas. A una primera aproximación, una proteína consiste en una cadena principal/cadena principal polar de la que cuelgan cadenas laterales de aminoácidos de polaridad y carga variables. Estas cadenas laterales pueden ser polares sin carga, con carga polar o no polares. En general las cadenas laterales no polares son más estables enterradas en el centro de la proteína, donde están rodeadas por otras cadenas laterales no polares y están orientadas alejadas del agua polar. Compárelo con la estructura de una micela. Sin embargo, dada la mayor complejidad de la estructura primaria y terciaria de la proteína, no todos los grupos laterales no polares pueden ser enterrados. Algunos están en la superficie expuestos al solvente. De igual manera, a las cadenas laterales polares y polares cargadas les gusta estar en la superficie expuestas al agua, pero algunas se encontrarán enterradas. Si lo están, estarán rodeados de cadenas laterales polares o interactuarán con donantes y aceptores de enlaces de hidrógeno enterrados en la columna vertebral que estabilizan el grupo polar enterrado. Aquí hay algunos hallazgos sobre proteínas derivadas de la conocida estructura 3D de miles de proteínas diferentes:
- En promedio, alrededor del 50% de los aminoácidos en una proteína se encuentran en estructura secundaria con un promedio de aproximadamente 27% de hélice alfa y 23% de estructura beta.
- La ubicación de la cadena lateral varía con la polaridad. El 83% de las cadenas laterales no polares (como Val, Leu, Ile, Met y Phe) están en el interior en la proteína plegada.
- Las cadenas laterales polares cargadas están divididas casi por igual entre ser enterradas o expuestas en la superficie.
- Los grupos polares no cargados como Asn, Gln, Ser, Thr y Tyr están en su mayoría (63%) enterrados, y no en la superficie.
- Las proteínas globulares (esféricas) son bastante compactas, con agua excluida. La densidad de empaque (V vdw /V tot) es de aproximadamente 0.74, que es como el cristal de NaCl y es igual a la densidad de empaque más cercana de 0.74. Esto se compara con los líquidos orgánicos, cuya densidad es de aproximadamente 0.6-0.7.
El empaquetamiento alrededor de una cadena lateral no polar enterrada del aminoácido fenilalanina (Phe) se muestra en el Jmol a continuación. Muestra la estructura de una proteína pequeña (proteína tirosina fosfatasa) y los grupos de aminoácidos que rodean la Phe enterrada.
 Jmol: Phe enterrado en proteína tirosina fosfatasa de bajo peso molecular (bovina) Jmol14 (Java) | JSmol (HTML5)
Jmol: Phe enterrado en proteína tirosina fosfatasa de bajo peso molecular (bovina) Jmol14 (Java) | JSmol (HTML5)Ejercicio\(\PageIndex{1}\)
La estructura secundaria en las proteínas implica
- enlaces de hidrógeno entre átomos de cadena lateral y átomos de cadena principal
- enlaces de hidrógeno entre los átomos de la cadena lateral y otros átomos de la cadena lateral
- Fuerzas de dispersión de Londres entre átomos de cadena lateral
- enlace de hidrógeno entre los átomos del esqueleto y otros átomos del esqueleto.
Ejercicio\(\PageIndex{2}\)
La estructura del aminoácido Ser se muestra a continuación:
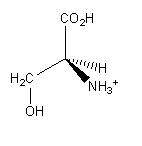
¿Qué grupos participan en la formación de enlaces covalentes con otros aminoácidos en una proteína?
- -CH 2 OH y -CO 2 H
- -CH 2 OH y -NH 3 +
- -CO 2 H y -NH 3 +
- -H y -CH 2 OH
Ejercicio\(\PageIndex{3}\)
La estructura del aminoácido Ser se muestra a continuación:
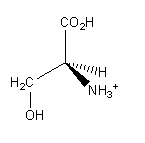
Si Ser estuviera en una hélice alfa, ¿qué grupo probablemente estaría proyectándose lejos del eje de la hélice?
- -CH 2 OH
- -NH 3 +
- -CO 2 H +
- -H
Ejercicio\(\PageIndex{4}\)
Si Ser fuera parte de una proteína, ¿cuál de las siguientes probablemente NO describiría el ambiente alrededor de la cadena lateral?
- podría ser enterrado en la proteína rodeada de cadenas laterales de aminoácidos no polares
- podría estar en la superficie rodeada de agua
- se enterraría adyacente a una cadena lateral de glutamina
- es polar por lo que no se pudo enterrar en la proteína
Ejercicio\(\PageIndex{5}\)
Las moléculas de una proteína dada tienen los siguientes rasgos:
- una secuencia de aminoácidos definida
- un peso molecular invariante
- una composición de aminoácidos fijos
- todo lo anterior
Ejercicio\(\PageIndex{6}\)
______ entre cadenas laterales de aminoácidos estrechamente empaquetadas en el interior de una proteína son una contribución importante a la estabilidad del estado nativo.
- Interacciones dipolo-dipolo
- Interacciones ion-ion
- Enlaces covalentes
- Fuerzas de dispersión de Londres
Ejercicio\(\PageIndex{7}\)
Si la siguiente sección de un polipéptido se pliega en una hélice alfa, ¿a qué aminoácido está unido el grupo carbonilo de Ala H?
Término N Ala-Leu-Ser-Asp-Glu-Val Término C
- Leu
- Ser
- Asp
- Glu
- Val
Ejercicio\(\PageIndex{8}\)
¿Qué forma de un péptido de 10 aminoácidos probablemente tendría la longitud más corta?
- el péptido está en solución y no forma parte de una estructura de proteínas
- el péptido es parte de una proteína y en una lámina beta antiparalela
- el péptido es parte de una proteína y en una lámina beta paralela
- el péptido es parte de una proteína y en una hélice alfa

