8.3: Ácidos nucleicos
- Page ID
- 80622
Los ácidos nucleicos ARN y ADN están involucrados en el almacenamiento y expresión de información genética en una célula. Ambos son polímeros de nucleótidos monoméricos. El ADN existe en la célula como hélices bicatenarias, mientras que el ARN normalmente es una molécula monocatenaria que puede plegarse en el espacio 3D para formar estructuras complejas secundarias (hélices bicatenarias) y terciarias de una manera similar a las proteínas. Las complejas estructuras 3D formadas por el ARN le permiten realizar funciones distintas al simple almacenamiento de información genética, como la catálisis. De ahí que la mayoría de los científicos creen que el ARN precedió tanto al ADN como a las proteínas en la evolución ya que puede almacenar información genética y catalizar reacciones químicas.
ADN
El ADN es un polímero, que consiste en mono meros llamados desoxinucleótidos. El monómero contiene un azúcar simple (desoxirribosa, que se muestra en negro a continuación), un grupo fosfato (en rojo) y un grupo R orgánico cíclico (en azul) que es análogo a la cadena lateral de un aminoácido.
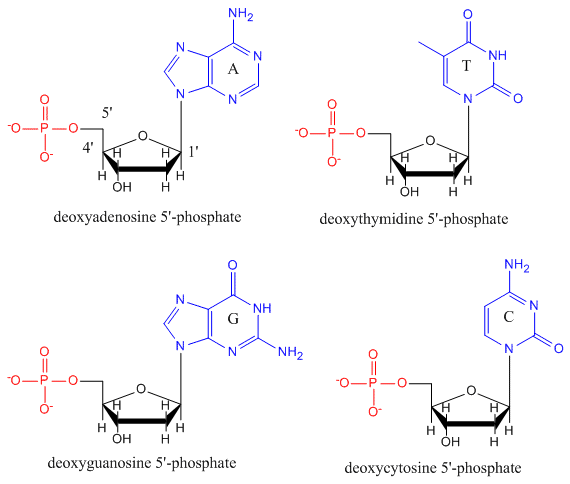
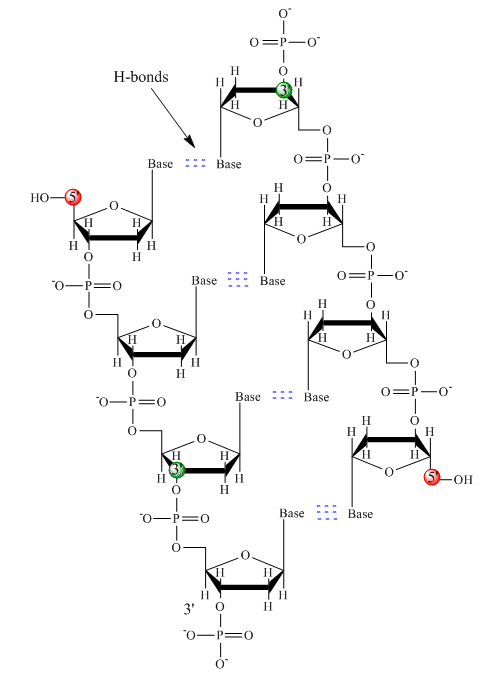
Solo se utilizan cuatro bases en el ADN (a diferencia de las 20 cadenas laterales diferentes en las proteínas) que abreviaremos, por simplicidad, como A, G, C y T. Son bases ya que contienen grupos amina que pueden aceptar protones. El polímero consiste en una cadena principal de azúcar - fosfato - azúcar - fosfato, con una base unida a cada molécula de azúcar. Al igual que con las proteínas, el esqueleto del ADN es polar pero también está cargado. Es un polianión. Las bases, análogas a las cadenas laterales de los aminoácidos, son predominantemente polares. Dada la naturaleza cargada de la cadena principal, es de esperar que el ADN no se pliegue a una forma globular compacta (esférica), incluso si los cationes cargados positivamente como el Mg se unen y estabilizan la carga en el polímero. En cambio, el ADN existe generalmente como una estructura bicatenaria (ds) con las cadenas principales de azúcar-fosfato de las dos cadenas diferentes que corren en direcciones opuestas (5'-3' y la otra 3'-5'). Las hebras se mantienen unidas por enlaces de hidrógeno entre bases en hebras complementarias. De ahí que al igual que las proteínas, el ADN tiene estructura secundaria; pero en este caso, en lugar de estar dentro de la cadena principal, los enlaces de hidrógeno se encuentran entre las bases de “cadena lateral” en cadenas opuestas. En realidad, es un nombre inapropiado llamar a ADNbc una molécula, ya que realmente consiste en dos cadenas diferentes y complementarias unidas por enlaces de hidrógeno. A continuación se incluye una estructura de dsADN que muestra la polaridad opuesta de las cadenas.
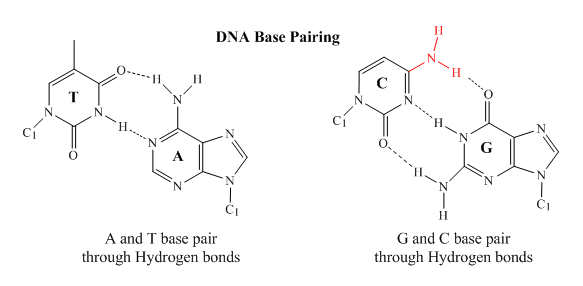
En el ADN bicatenario, la base de guanina (G) en una hebra puede formar tres enlaces H con una base de citosina (C) en otra hebra (esto se llama par de bases GC). La base de timina (T) en una hebra puede formar dos enlaces H con una base de adenina (A) en la otra hebra (esto se llama un par de bases AT). El ADN bicatenario tiene una estructura geométrica regular con una distancia fija entre las dos cadenas principales. Esto requiere que los pares de bases consten de una base con una estructura de dos anillos (bicíclica) (estas bases se llaman purinas) y una con una sola estructura de anillo (estas bases se llaman pirimidinas). De ahí que una G y A o una T y C no sean posibles parejas de pares base.
El ADN bicatenario varía en longitud (número de unidades de azúcar-fosfato conectadas), composición de bases (cuántas de cada conjunto de bases) y secuencia (el orden de las bases en la cadena principal). Los siguientes enlaces proporcionan modelos Jmol interactivos de dsDNA hechos por Angel Herráez, Univ. de Alcalá (España) y Eric Martz.
 JSmol externo:
JSmol externo:- Ds-ADN con pares de bases y enlaces H
- Hilos y espina dorsal helicoidal
- Finales, paralelismo
Los cromosomas consisten en un ADNbc con muchas proteínas unidas diferentes. El genoma humano tiene alrededor de 3 mil millones de pares de bases de ADN. Por lo tanto, en promedio, cada cromosoma individual de un par tiene alrededor de 150 millones de pares de bases y muchas proteínas unidas a él. dsDNA es una molécula altamente cargada, y puede verse, a una primera aproximación, como una molécula larga similar a varilla con una gran carga negativa. Es un polianión. Esta molécula muy grande debe de alguna manera ser empaquetada en un pequeño núcleo de una célula diminuta. En células complejas (eucariotas), este problema de empaquetamiento se resuelve enrollando ADN alrededor de un complejo central de cuatro pares diferentes (ocho proteínas en total) de proteínas histonas (H2A, H2B, H3 y H4) que tienen cargas positivas netas. El complejo central de histona con dsDNA enrollado alrededor de 2.5 veces se llama nucleosoma.
 Jmol: Nucleosoma Jmol14 (Java) | JSmol (HTML5)
Jmol: Nucleosoma Jmol14 (Java) | JSmol (HTML5)El ADN puede adoptar otros dos tipos de formas de doble hélice. El descubierto por Watson y Crick y encontrado en la mayoría de los libros de texto se llama B-ADN. Dependiendo de la secuencia de ADN real y del estado de hidratación del ADN, se puede convencer para formar otros dos tipos de hélices bicatenarias, ADN Z y A. La forma A es mucho más abierta que la forma B.
Los 3.2 mil millones de pares de bases de ADN en humanos contienen alrededor de 24,000 tramos cortos (genes) que codifican diferentes proteínas. Estos genes están intercalados entre ADN que ayuda a determinar si el gen se decodifica en moléculas de ARN (ver más abajo) y finalmente en proteínas. Para que un gen en particular se active (o “encienda”), las proteínas específicas deben unirse a la región de un gen en particular. ¿Cómo pueden las proteínas de unión encontrar dianas de unión específicas entre la gran cantidad de pares de bases que a una primera aproximación tienen una repetición repetitiva de azúcar-fosfato-base? El Jmol a continuación muestra cómo se puede lograr la especificidad. Cuando el ADN se enrolla en una doble hélice a través de pares de bases entre AT y GC, los donantes de enlaces de hidrógeno (amida H) y aceptores (O) en las bases que no se usan en el apareamiento de bases intracatenarias todavía están disponibles en la arboleda mayor y menor de la hélice ds-ADN (ver Jmol a continuación). Las secuencias de pares de bases únicas mostrarán patrones únicos de donantes y aceptores de enlaces H en la arboleda principal. Estos donadores/aceptores pueden ser reconocidos por proteínas de unión a ADN específicas que pueden conducir a la activación génica tras la unión.
 Jmol: ADNbc que muestra donantes y aceptores únicos de enlaces H en la arboleda mayor Jmol14 (Java) | JSmol (HTML5)
Jmol: ADNbc que muestra donantes y aceptores únicos de enlaces H en la arboleda mayor Jmol14 (Java) | JSmol (HTML5)RNA
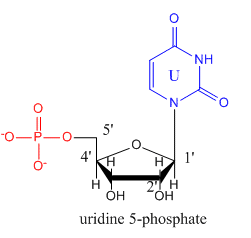
Los monómeros que componen el ARN son idénticos a los del ADN con dos diferencias. El 2'C del anillo de ribosa tiene un OH en lugar de H (como se encuentra en la desoxirribosa). Además, la base de timina (T) que se encuentra en el ADN es reemplazada por uracilo (U). A continuación se muestra la estructura de uridina-5-monofosfato.
Basándose en la analogía con el ADN, se podría esperar que el ARN adopte una conformación helicoidal bicatenaria. Puede hacer esto en tramos cortos, pero en cambio, incluso en presencia de cationes divalentes cargados positivamente, se encuentra predominantemente en forma monocatenaria. Esta cadena sencilla se pliega para formar regiones de estructura secundaria (hélices bicatenarias) embebidas en estructuras terciarias complejas (no muy diferente a una proteína).
La adición del 2'OH en el anillo de ribosa o ARN altera el frunce del anillo de ribosa y desestabiliza el dsRNA en comparación con el ss-RNA a través de interferencia estérica. El anillo de ribosa o desoxirribosa de cinco miembros puede adoptar una estructura similar a la envoltura en la que un átomo del anillo, que representa la punta de la envoltura, está por encima, o endo, a los otros cuatro átomos coplanares. En desoxirribosa, el átomo de carbono del anillo 2' prefiere la posición endo; en ribosa, el 3' C es endo (ver modelo Jmol a continuación). Cuando se forma ARN bicatenario, en realidad es muy similar en estructura a la forma de A-ADN más abierta (y menos abundantemente encontrada).
 Jmol: Modelo de Jmol que muestra el frunce del anillo de ribosa de ARN y el anillo de desoxirribosa de ADN (actualizado a HTML5 lo antes posible)
Jmol: Modelo de Jmol que muestra el frunce del anillo de ribosa de ARN y el anillo de desoxirribosa de ADN (actualizado a HTML5 lo antes posible) Jmol: Modelo Jmol comparando dsDNA y dsRNA (actualizado a HTML5 lo antes posible)
Jmol: Modelo Jmol comparando dsDNA y dsRNA (actualizado a HTML5 lo antes posible)Aquí hay algunos ejemplos de estructuras terciarias complejas formadas a partir de ARN monocatenario que se pliega para formar una estructura 3D compacta con algunas hélices de tipo A bicatenarias estabilizadas por enlaces H intramoleculares entre pares de bases G/C y A/U.
 Jmol: Modelo Jmol de ARNt (tenga en cuenta las regiones de estructura secundaria - ARNbc tipo A) (actualizado a HTML5 lo antes posible)
Jmol: Modelo Jmol de ARNt (tenga en cuenta las regiones de estructura secundaria - ARNbc tipo A) (actualizado a HTML5 lo antes posible) Jmol: Modelo Jmol de ribosoma 70 S de T. Thermophilus que muestra interacciones de ARNm y ARNt (actualizado a HTML5 lo antes posible)
Jmol: Modelo Jmol de ribosoma 70 S de T. Thermophilus que muestra interacciones de ARNm y ARNt (actualizado a HTML5 lo antes posible)Ejercicio\(\PageIndex{1}\)
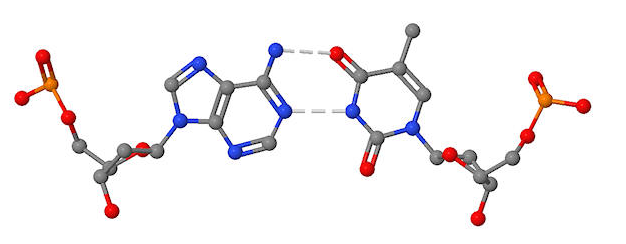
La siguiente figura muestra un par de bases real en un ADNbc.
- identificar el par de bases y los azúcares desoxirribosa
- colocar una carga negativa en cualquier átomo con una carga negativa
- marcar átomos distintos de aquellos entre las bases que podrían formar enlaces H con otras moléculas de unión a ADN
Ejercicio\(\PageIndex{2}\)
Haz lo mismo para el siguiente par de bases
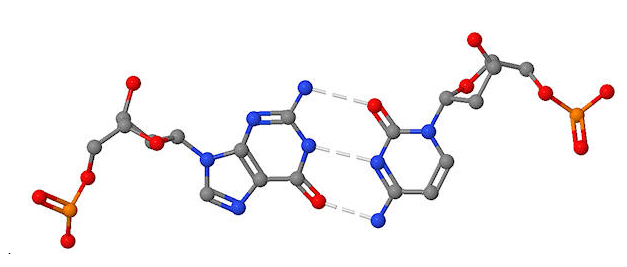
- identificar el par de bases y los azúcares desoxirribosa
- colocar una carga negativa en cualquier átomo con una carga negativa
- marcar átomos distintos de aquellos entre las bases que podrían formar enlaces H con otras moléculas de unión a ADN
Ejercicio\(\PageIndex{3}\)
La columna vertebral del ADNbc se destaca a continuación. Coloca un círculo en cada átomo con una carga negativa.
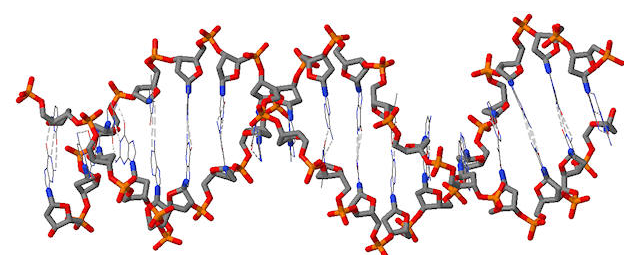
Ejercicio\(\PageIndex{4}\)
A continuación se muestra la estructura de un péptido completamente protonado, poli-Lys, compuesto solo por el aminoácido lisina.
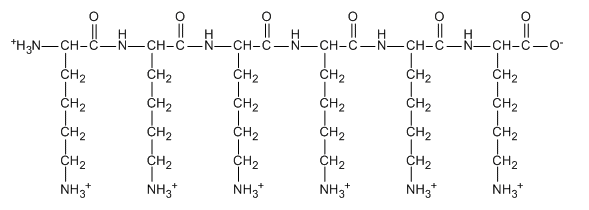
Representando esta molécula en forma de caricatura como una elipse alargada, muestra cómo podría interactuar con la hélice de ADN
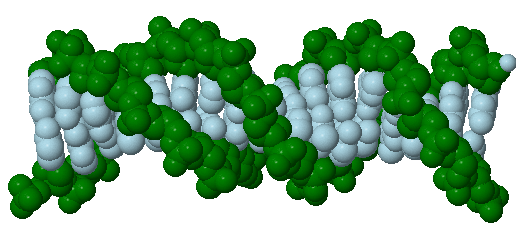
Ejercicio\(\PageIndex{5}\)
Con base en su respuesta a la pregunta anterior, ¿qué característica general podría estar presente en las proteínas de unión al ADN?

