9.7: Cosecha de los fotones- Fotosistema I
- Page ID
- 79392
Habiendo generado ya el gradiente de protones utilizado para la generación de ATP, ¿qué queda por hacer?
Recuerda volver a esa clase de biología de secundaria cuando viste por primera vez la ecuación para la fotosíntesis.
\[\ce{6 CO2_{(g)} + 6H2O_{(l)} -> C6H12O6_{(s)} + 6O2_{(g)}} \nonumber\]
Podemos romper eso en dos medias reacciones. El primero es la oxidación del agua.
\[\ce{2H2O_{(l)} -> O2_{(g)} + 4H^{+}_{(aq)} + 4e^{-}_{(aq)}} \nonumber\]
La otra mitad es la reducción del dióxido de carbono.
\[\ce{6CO2_{(g)} + 24H^{+}_{(aq)} -> 24e^{-}_{(aq)} -> C6H12O6_{(s)} + 6H2O_{(l)}} \nonumber\]
Si bien estas dos medias reacciones están físicamente separadas entre sí, los electrones aún deben pasar de uno a otro para completar la reacción completa. Ya hemos logrado la primera reacción y ahora tenemos electrones en la mano para llevar a cabo la segunda. Vamos a necesitar un agente reductor biológico, como el NADH, para llevar a cabo el segundo. En este caso, utilizaremos un compuesto estrechamente relacionado, el NADPH.
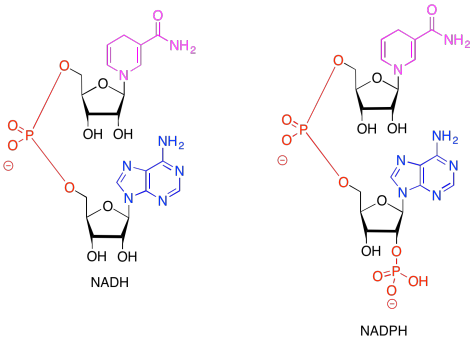
En ambos dibujos anteriores, la porción púrpura del compuesto es donde se aceptan los electrones. La porción negra es azúcar, la azul es una base de purina y la roja es la parte de fosfato.
Debido a que el NADH y el NADPH son portadores de electrones, es importante reconocer tanto las formas reducidas como las oxidadas. Dadas las formas reducidas de NADH y NADPH mostradas anteriormente, dibujar las formas oxidadas, y mostrar dónde se incorpora el:H - en la molécula en la forma reducida.
- Contestar
-
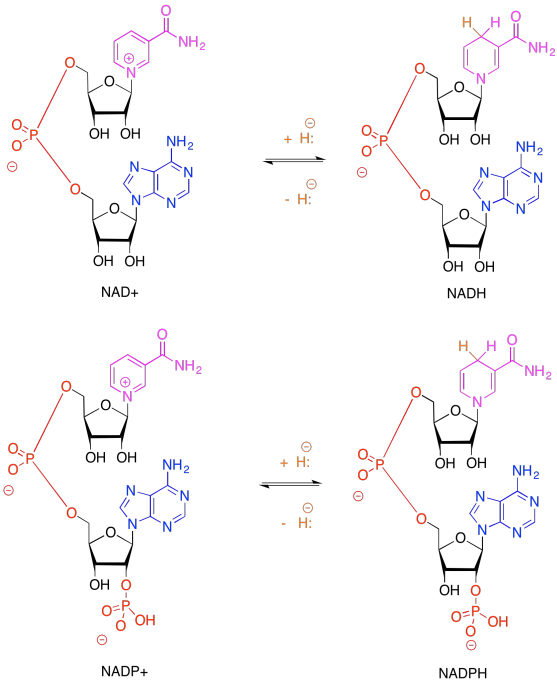
El problema es que el NADPH es un portador de electrones de relativamente alta energía —recuerden, el NADH estaba en el extremo cuesta arriba de la cadena de transporte de electrones en fosforilación oxidativa— y nuestros electrones acaban de montar su trineo hasta el fondo de la colina. Si vamos a hacer NADPH, tendremos que volver a tirar de nuevo los electrones cuesta arriba. Ese es esencialmente el trabajo del fotosistema I, que se muestra a continuación.
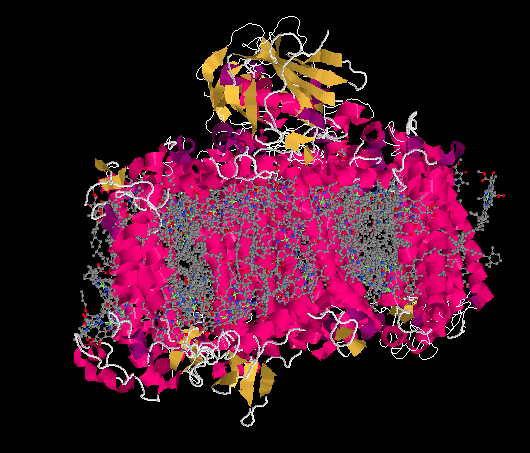
La vista de la estructura cristalina de solo ligando revela una imagen que ahora debería ser algo familiar: una gran cantidad de moléculas de antenas que forman un complejo de recolección de luz. Una vez más, las moléculas de antenas incluyen principalmente clorofilas, así como carotenoides como caroteno y licopeno, además de algunas xantinas como la violaxantina; las moléculas exactas varían dependiendo de si estamos mirando plantas, algas o bacterias.
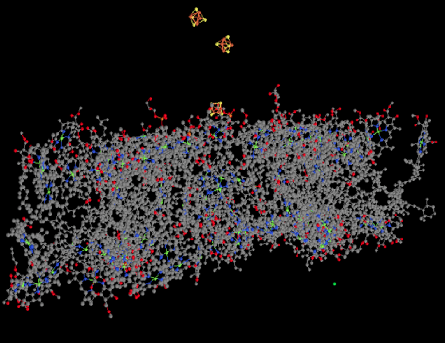
También es visible en la vista de solo ligando un grupo de clústeres de FE. Estos clústeres juegan un papel en la cadena de transporte de electrones del fotosistema I, conduciendo el electrón hacia el estroma, donde serán utilizados para reducir el NADP+ a NADPH. Ese agente reductor biológico, estrechamente relacionado con el NADH, se utilizará para ayudar a reducir el dióxido de carbono.
Hagamos una pausa por un momento. La mayoría de las estructuras cristalinas de rayos X que hemos visto provienen de bacterias. Por supuesto, estamos mucho más familiarizados con la idea de la fotosíntesis en las plantas. ¿Las proteínas y los procesos involucrados en la fotosíntesis en una son algo como las otras? A modo de comparación, aquí está el fotosistema I de una planta de espinacas.
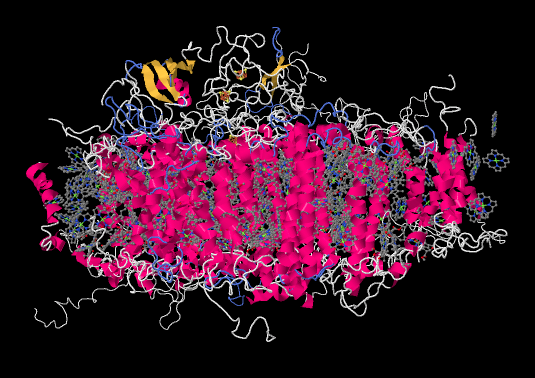
Es aproximadamente similar, aunque no idéntico, al de la bacteria. Hay una amplia franja de hélices alfa a través del medio, rematadas con un haz de hojas beta. En todo caso, la proteína de la espinaca se ve superficialmente un poco más simple que la de la bacteria. La vista de solo ligando, a continuación, es sorprendentemente similar, mostrando una pulverización de cromóforos y una serie de racimos de hierro-azufre.

Vamos a retomar la historia donde la dejamos cuando estábamos discutiendo el citocromo b 6 f. Un solo electrón estaba siendo transportado por el portador de electrones móvil soluble, plastocianina. Ese único electrón se entrega directamente al centro de reacción del fotosistema I. Ese centro de reacción es bastante similar al del fotosistema II, aunque absorbe fotones de una longitud de onda ligeramente mayor, 700 nm. El centro P700 contiene un dímero excitónico, un par de moléculas de clorofila muy espaciadas que absorben el fotón y experimentan separación electrón/agujero en las dos moléculas diferentes.
Al igual que en el P680, el estado excitado P700* puede aceptar un electrón sobre el socio clorfilo que tiene el agujero, P D2 + *. Ahí es donde la plastocianina entrega su electrón.
En tanto, la parte del dímero con el electrón extra, P D1 - *, pasa el electrón a otra estación en la cadena de transporte de electrones. Parte del objetivo aquí es hacer que los electrones vuelvan al estroma donde está esperando el NADP+. Si lo piensas bien, los electrones empezaron en el lumen, en una molécula de agua. Viajaron a través de la membrana tilacoidea en el fotosistema II hasta que se unieron a una plastoquinona y recogieron un par de protones; esos protones procedían del estroma. Las plastoquinonas viajaron de regreso a través de la membrana, una migración sigilosa de H + polar en la forma en PQH2 no polar, y entregaron sus protones a la luz. Los electrones de patada lateral saltaron a lo largo del borde de la luz en un portador de plastocianina, y ahora están viajando de regreso hacia el estroma a ese NADP+ en espera. El NADPH se producirá en el estroma porque ahí es donde se necesitará para la captura de CO 2.
Nuevamente, al igual que en P680, el primer aceptor de electrones después de P700 es una sola clorofila. Este anión clorofila que se forma es ligeramente descendente en energía del que está en el dímero, P D1 - *. A partir de ahí, el electrón pasa a una filoquinona, que es solo otra variación de la familia de quinonas familar.
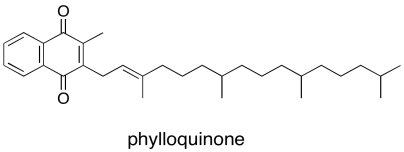
La filoquinona envía el electrón a esa serie de tres cúmulos de FeS visibles en la estructura cristalina. A partir de ahí, el electrón sale del fotosistema I y se deposita sobre una ferredoxina, una pequeña proteína que se une a otro cúmulo de hierro y azufre. La ferredoxina se localiza en el estroma, donde puede unirse con una enzima llamada ferredoxina NADP+ reductasa. Como la mayoría de las enzimas, el nombre dice exactamente lo que hace: cataliza la reacción en la que la ferredoxina reduce el NADP+, resultando en NADPH.
A continuación se muestra la estructura cristalina de una ferredoxina. Este en particular no es del fotosistema I (se encuentra en una bacteria fijadora de nitrógeno), sino que te da la idea. La proteína es lo suficientemente simple como para que puedas ver que es un dímero; la mitad superior es la misma que la mitad inferior, volteada. También se puede ver claramente un cúmulo de 2Fe2S en el borde de cada subunidad. La ubicación de ese cúmulo de FeS cerca del borde de la proteína es probablemente importante para facilitar la transferencia de electrones con otra proteína.
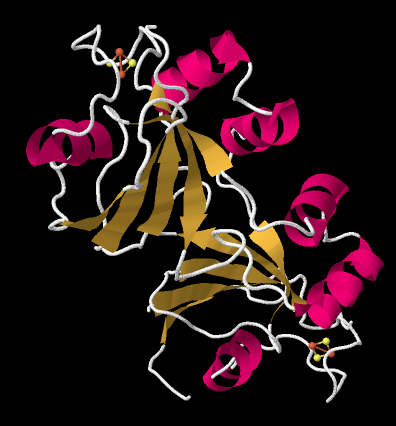
La ferredoxina es una proteína portadora de electrones. Su trabajo es entregar electrones a la ferredoxina NADP+ reductasa en el estroma, que luego utilizará los electrones para hacer NADPH. El NADPH es una fuente biológica de iones hidruro (formalmente, H-, o un protón más dos electrones) que en este contexto se utilizará para ayudar a reducir el dióxido de carbono en carbohidratos. A continuación, podemos ver un ejemplo. Esta es otra proteína dimérica con simetría rotacional: el lado izquierdo es el mismo que el derecho, pero volteado.

Si miramos dentro, podemos ver las moléculas NADP+ esperando en cualquiera de los extremos de la estructura, tanto en el extremo izquierdo como en el extremo derecho. Además, hay un par de moléculas que parecen vagamente relacionadas con el NADP+ hacia la mitad de la imagen. Esas son dos moléculas FAD. Las moléculas de FAD están ahí para conducir electrones de la ferredoxina t el NADP+.

En la imagen de abajo, la parte púrpura de la molécula es la región redox activa. Es capaz de aceptar electrones. La parte negra es azúcar, la azul es una base de purina y la roja es fosfato.

Mostrar la forma reducida de FAD. Recuerde, la forma reducida se llama FADH2.
- Contestar
-

¿Qué papel crees que juega el FAD en la ferredoxina NADP+ reductasa? ¿Por qué la ferredoxina no reduce el NADP+ directamente?
- Contestar
-
Recuerde, NADP+, como NAD+, es únicamente un agente oxidante de 2 electrones. Sólo puede recibir un par de electrones asociados a un hidruro. La ferredoxina, a través de un cúmulo de FeS, solo puede dar un electrón a la vez. Se necesita un adaptador entre estos dos portadores. El FAD puede aceptar 1 o 2 electrones, procediendo a cualquiera de los FADH . radical (estabilizado mediante deslocalización por resonancia extensa) o a FADH2.
Debido a que el fotosistema II y el fotosistema I forman parte de la misma cadena de transporte de electrones, es importante que el centro de reacción en uno se excita con la misma frecuencia que el centro de reacción en el otro. La regulación se logra en parte a través de la modificación covalente. Por ejemplo, la actividad insuficiente en el fotosistema I da como resultado la activación de una quinasa, lo que da como resultado la liberación de algunas de las moléculas de antenas del complejo de captación de luz en el fotosistema II.
- ¿Cómo ayudaría esta situación a remediar la falta de paridad en la actividad entre el fotosistema I y el fotosistema II?
- Explique cómo la quinasa lograría este resultado.
- Supongamos que ocurrió la situación opuesta: actividad insuficiente en el fotosistema II. ¿Qué enzima probablemente se activaría para remediar la situación?
- Contestar a
-
a) La liberación de moléculas de antenas del complejo de captación de luz en el fotosistema II disminuiría tanto el número de fotones capturados como el número de electrones enviados a la cadena de transporte de electrones. Por el contrario, en un modelo de “charco de cromóforos”, la liberación de estas moléculas del fotosistema II las haría más disponibles para su captación por el fotosistema I, resultando en una mayor absorción de fotones allí, y una mayor capacidad para aceptar electrones entrantes. Si están regulados, estos dos sistemas deberían poder nivelarse a una tasa idéntica de absorción de fotones.
- Respuesta b
-
b) Las quinasas fosforilan las cadenas laterales hidroxi en residuos de serina (y a veces treoninas y tirosinas). Esa modificación covalente da como resultado un cambio en la carga de la cadena lateral de neutra a aniónica. Se puede esperar que se produzcan cambios conformacionales significativos en la proteína. Es probable que este cambio conformacional resulte en una unión más floja de las moléculas de antenas en el complejo de recolección de luz del fotosistema II.
Además, la quinasa parece promover una unión más estrecha de las moléculas de antenas en el fotosistema I, presumiblemente a través de un cambio conformacional allí. El resultado es un desplazamiento de estas moléculas de la piscina disponible, de tal manera que cromóforos adicionales están disponibles en el fotosistema I y menos se retienen en el fotosistema II, hasta que la absorción de fotones en los dos sitios es igual.
- Respuesta c
-
c) De hecho, la fosfatasa, que es la enzima complementaria a la quinasa, se activa cuando el fotosistema I tiene alta actividad en comparación con el fotosistema I. Las fosfatasas eliminan los grupos fosfato de las serinas y residuos relacionados. El resultado sería una unión más estrecha de las moléculas de antenas en el fotosistema I y una unión más floja de las moléculas de antenas en el fotosistema II, de manera que la absorción de luz podría igualarse entre los dos sitios.
El uso de fosfatasas y quinasas para lograr objetivos opuestos en la regulación es común en varios sistemas bioquímicos.
Podemos resumir con un boceto del fotosistema I:
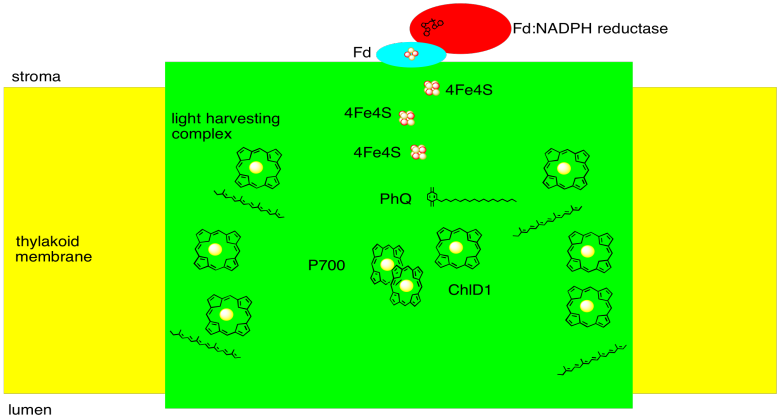
1. Estructuras cristalinas de rayos X: Jordan, P.; Fromme, P.; Witt, H.T.; Klukas, O.; Saenger, W.; Krauss, N. Estructura tridimensional del fotosistema cianobacteriano I a una resolución de 2.5 A. Naturaleza 2001, 411, 909-917. Imágenes obtenidas a través del Banco de Datos de Proteínas RCSB (1JB0).
2. Estructuras cristalinas de rayos X: Amunts, A., Drory, O., Nelson, N. La estructura de un supercomplejo de fotosistema I de planta a una resolución de 3.4 A. Naturaleza 2007, 447, 58-63. Imágenes obtenidas a través del Banco de Datos de Proteínas RCSB (2o01).
3. Estructuras cristalinas de rayos X: Schlesier, J., Rohde, M., Gerhardt, S., Einsle, O. Un interruptor conformacional desencadena la protección de nitrogenasa contra daños por oxígeno por la proteína II de Shethna (FeSiI). J. Am. Chem. Soc. 2016, 138, 239-247. Imágenes obtenidas a través del Banco de Datos de Proteínas RCSB (5ffi).
4. Estructuras cristalinas de rayos X: Komori, H., Seo, D., Sakurai, T., Higuchi, Y. Análisis de la estructura cristalina de Bacillus subtilis Ferredoxina-NADP (+) oxidorreductasa y la base estructural de su selectividad de sustrato. Proteína Sci. 2010, 19, 2279-2290. Imágenes obtenidas a través del Banco de Datos de Proteínas RCSB (3Lzw).

