2.7: Día 15- Polímeros de condensación, proteínas
- Page ID
- 78971
14
Día 15: Polímeros de condensación, proteínas
Polímeros de condensación D15.1
Un polímero de condensación es un polímero formado a través de una reacción de condensación (ver Sección D13.5). La molécula muy pequeña producida en cada condensación puede ser H 2 O, HCl o alguna otra molécula simple. Las polimerizaciones por condensación a menudo, pero no siempre, combinan dos monómeros diferentes en una estructura alterna.
Los polímeros de condensación generalmente crecen formando enlaces éster o amida, donde se forman nuevos enlaces C-O o C-N σ para unir monómeros. Un monómero típico tiene un grupo funcional en cada extremo de la molécula. Cuando un extremo del monómero reacciona y se añade a una cadena de polímero, el grupo funcional en el otro extremo permanece y permite una reacción adicional para alargar la cadena polimérica.
Poliésteres D15.2
Un poliéster es un polímero donde las unidades individuales se mantienen unidas por enlaces éster. Por ejemplo, el polietilen tereftalato común (abreviado como PET o PETE; tiene marcas como Dacron o Mylar) consiste en cadenas poliméricas formadas a partir de monómeros de ácido tereftálico (ácido benceno-1,4-dicarboxílico) y etilenglicol (etano-1,2-diol).
La reacción de condensación que da como resultado la formación del enlace éster en PET se muestra en la Figura 1. Reacciones de condensación repetidas dan como resultado el crecimiento escalonado de una cadena de polímero.
La unidad repetitiva de PET es
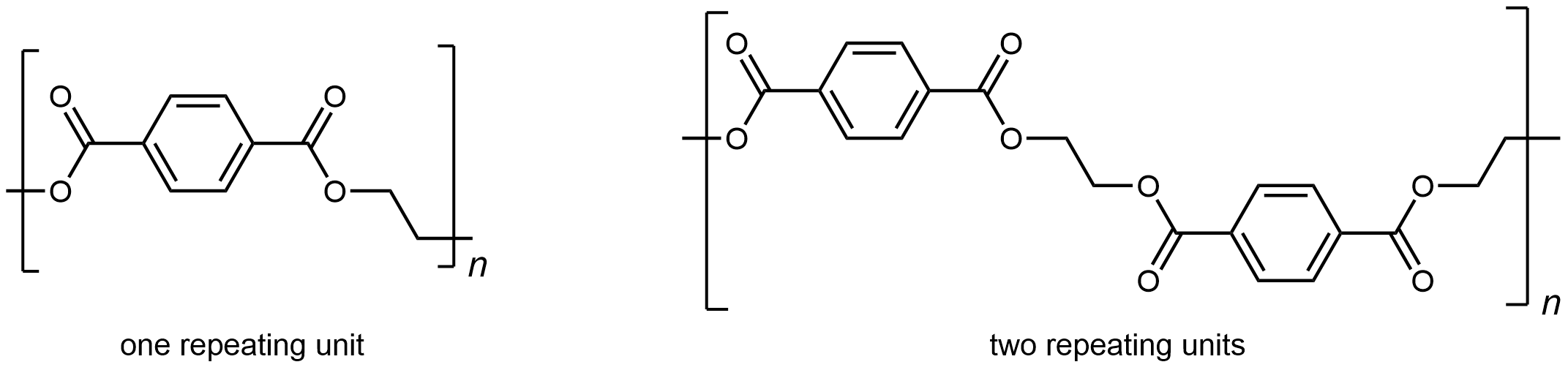
La presencia de grupos éster, un grupo funcional polar, en el polímero aumenta las fuerzas intermoleculares entre las cadenas poliméricas, y con ello aumenta la cristalinidad y la resistencia a la tracción del polímero. El PET hace que las fibras sean excelentes y se utilizan en muchas telas. También se utiliza para hacer botellas para refrescos y otras bebidas. El poliéster es biológicamente inerte, por lo que un tubo de poliéster tejido puede usarse en cirugía para reparar o reemplazar secciones enfermas de vasos sanguíneos.
Poliamidas D15.3
Una poliamida es un polímero en el que las unidades individuales se mantienen unidas por enlaces amida. Por ejemplo, el nylon 66 se obtiene de los monómeros 1,6-hexanodiamina y ácido hexanodioico. La reacción de condensación entre estos dos monómeros forma un enlace amida (resaltado):
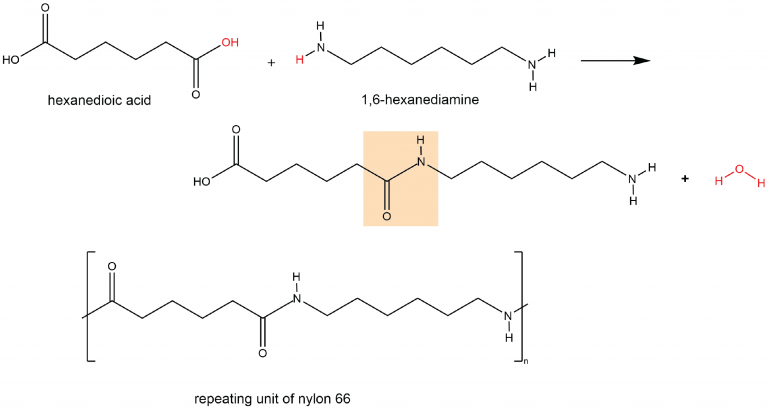
En un extremo, la molécula producto tiene un grupo ácido carboxílico, el cual puede sufrir una reacción de condensación con otra molécula de 1,6-hexanodiamina. En el otro extremo hay un grupo amina, que puede reaccionar con otra molécula de ácido hexanodioico. Tales reacciones de condensación continuas conducen a la formación de una hebra de polímero de nylon 66, cuya unidad repetitiva también se muestra anteriormente.
El “66” en nylon 66 representa los seis átomos de carbono en cada una de las moléculas monómeras. Otros nilones tienen diferentes números de átomos de carbono en las moléculas monoméricas, como el nylon 510, que tiene 5 átomos de carbono en la diamina (1,5-pentanodiamina) y 10 átomos de carbono en el diácido (ácido decanodioico).
El nailon produce hilos y fibras extremadamente fuertes porque además de las fuerzas de dispersión de Londres y las atracciones dipolo-dipolo, existen enlaces de hidrógeno entre las cadenas de polímero. Específicamente, se puede formar un enlace de hidrógeno entre un N-H en una cadena y un par solitario de carbonilo O en una cadena vecina.
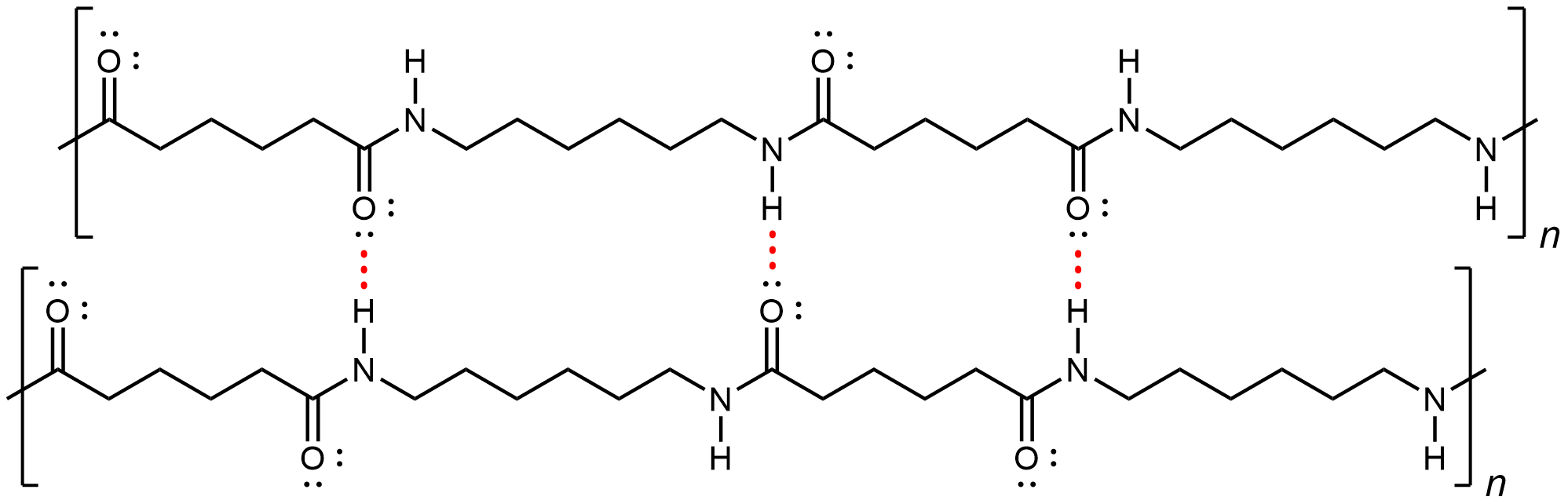
Si tira de ambos extremos de un hilo de nylon, después de un ligero estiramiento, resistirá romperse debido a los fuertes IMF que mantienen unidas las cadenas vecinas.
El kevlar es un polímero sintético hecho de 1,4-fenilendiamina y monómeros de cloruro de tereftaloilo. El subproducto de esta reacción de condensación es HCl.

El material tiene una alta relación tensión-resistencia a peso (es aproximadamente 5 veces más fuerte que un peso igual de acero), lo que lo hace útil para muchas aplicaciones, desde llantas de bicicleta hasta velas y armaduras corporales.
Similar al nylon, parte de la resistencia del Kevlar se debe a interacciones dipolo-dipolo y enlaces de hidrógeno (Figura 3) que aumentan las fuerzas intermoleculares entre las hebras de polímero.
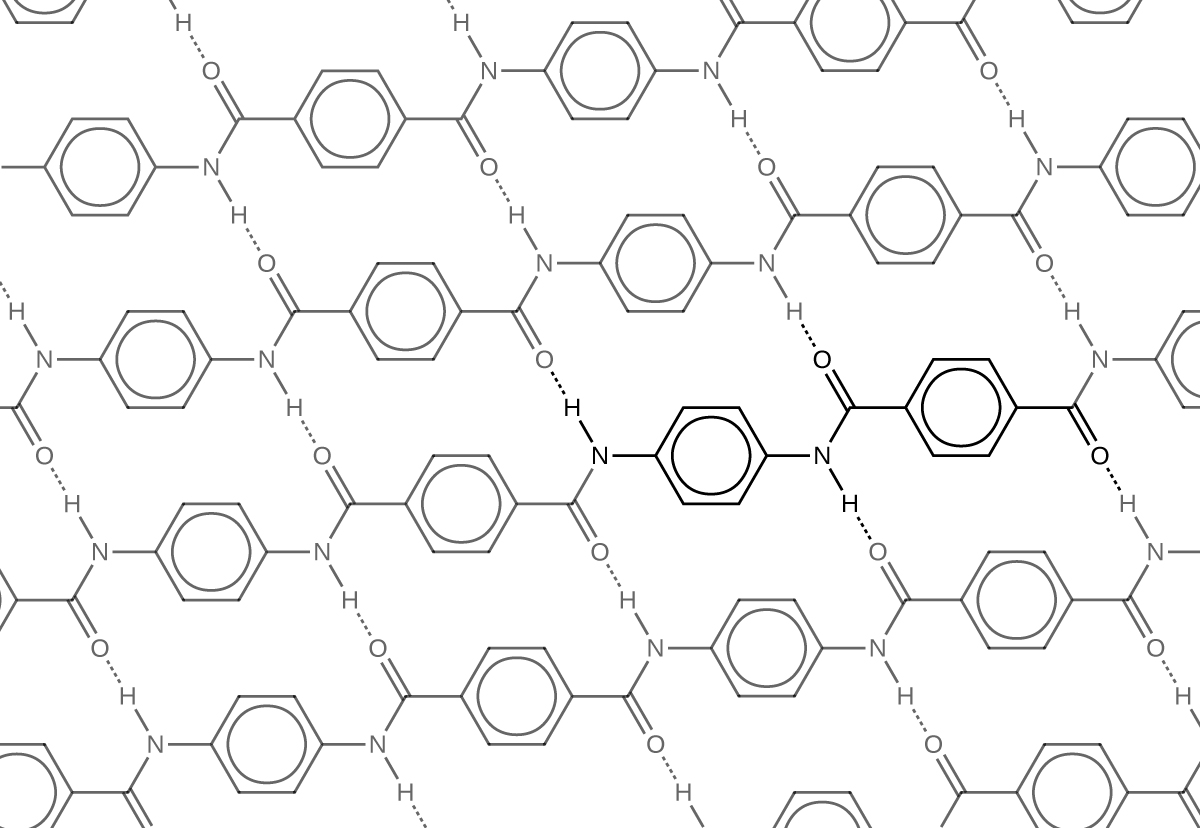
Observe que en comparación con el nylon 66, la densidad de los enlaces de hidrógeno en Kevlar es mayor, impartiendo fuerzas intermoleculares más fuertes.
Además, Kevlar también tiene fuerzas de dispersión más fuertes en Londres. Considera la unidad repetitiva de Kevlar resaltada en la Figura 3. Es una estructura plana, con anillos de benceno planos conectados por grupos amida planos. De ahí que una sola capa de Kevlar, como se muestra en la Figura 3, es plana. Cuando las capas de Kevlar se colocan una encima de la otra, se pueden apilar de manera muy eficiente, permitiendo las máximas fuerzas de dispersión de Londres.
Además de sus usos más conocidos, el Kevlar se utiliza en aplicaciones criogénicas debido a su muy baja conductividad térmica. El Kevlar mantiene su alta resistencia cuando se enfría a temperaturas de nitrógeno líquido (—196 °C). Muchos otros plásticos se vuelven quebradizos y se rompen a esa temperatura.
Proteínas D15.4
Las proteínas son polímeros de condensación elaborados a partir de aminoácidos. Un aminoácido consiste en un átomo de carbono (llamado carbono α, que se muestra en rojo a continuación) unido a un átomo de hidrógeno, un grupo amina, un grupo ácido carboxílico y un grupo R, a menudo llamado cadena lateral.

Observe que el carbono α es quiral cuando el grupo “R” difiere de los otros tres grupos. Se conocen cerca de 500 aminoácidos naturales, aunque solo veinte se encuentran en las moléculas proteicas en humanos. De estos veinte aminoácidos, diecinueve tienen un carbono α quiral.
Los aminoácidos se unen entre sí en proteínas mediante enlaces amida formados a través de reacciones de condensación. Un enlace amida en una proteína a menudo se llama enlace peptídico.
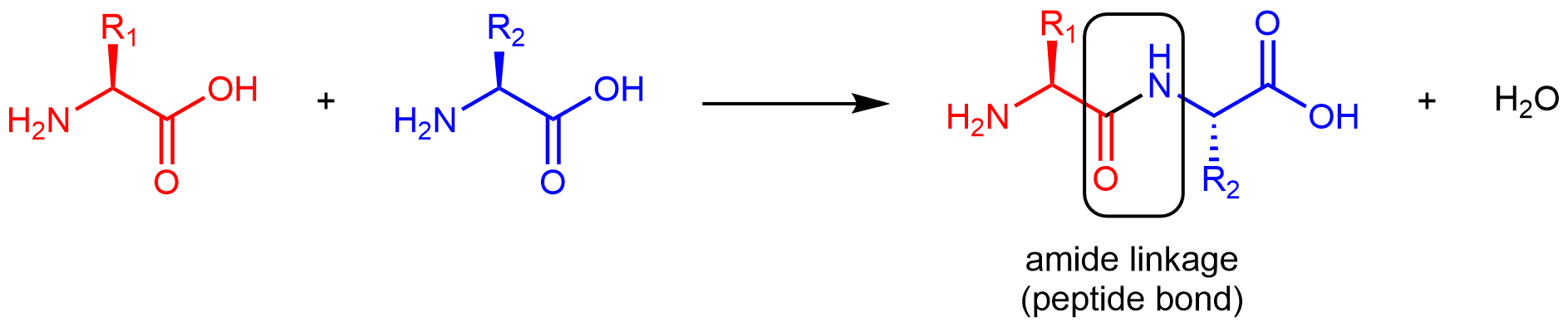
Tenga en cuenta que la reacción no involucra el átomo de carbono α, y la quiralidad en el carbono α es la misma después de la reacción que antes. (En la ecuación anterior, R2 se representa con un enlace discontinuo porque el aminoácido mostrado en azul ha sido volteado, girado 180° alrededor de un eje horizontal, desde el reactivo hasta el producto).
Como todos los grupos funcionales amida, los enlaces amida en una cadena de proteína son planos. Los átomos de carbono α, sin embargo, tienen geometría local tetraédrica. Esta cadena molecular de carbonos α conectados por grupos amida, se conoce como la cadena principal de la proteína. Es la cadena principal de una hebra de proteína. Los grupos de aminoácidos R se denominan cadenas laterales porque están unidos a, pero se ramifica de, la cadena principal.
15.5 Aminoácidos
Los veinte aminoácidos a menudo se clasifican en cuatro grupos: hidrófobos, polares pero sin carga, básicos y ácidos, dependiendo de la naturaleza de sus cadenas laterales. Los nueve aminoácidos que tienen cadenas laterales hidrófobas se muestran en la Figura 4:
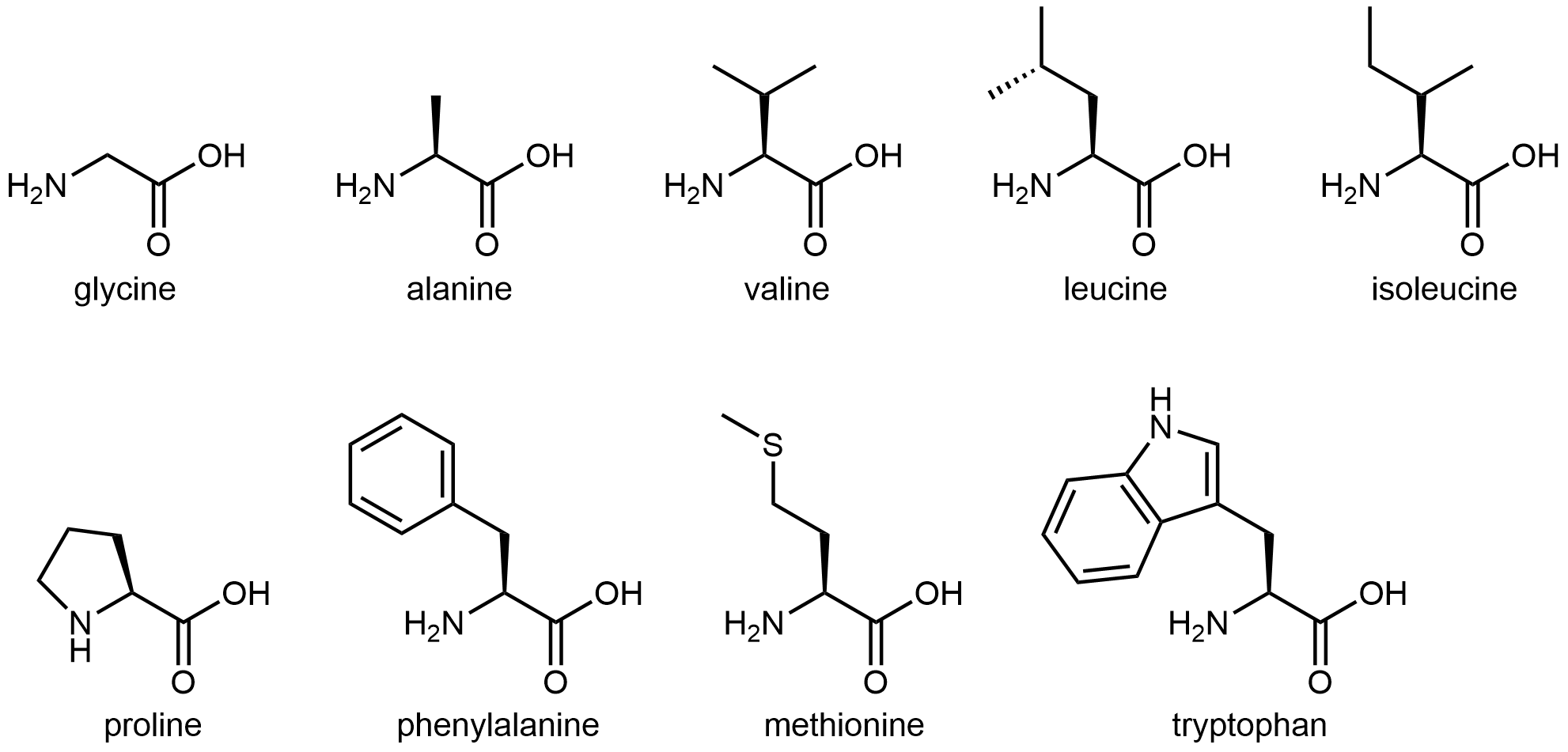
Estas cadenas laterales hidrófobas están compuestas principalmente por carbono e hidrógeno, tienen momentos dipolares muy pequeños y tienden a no mezclarse con agua. Como veremos, esta característica tiene implicaciones importantes para la estructura de una proteína.
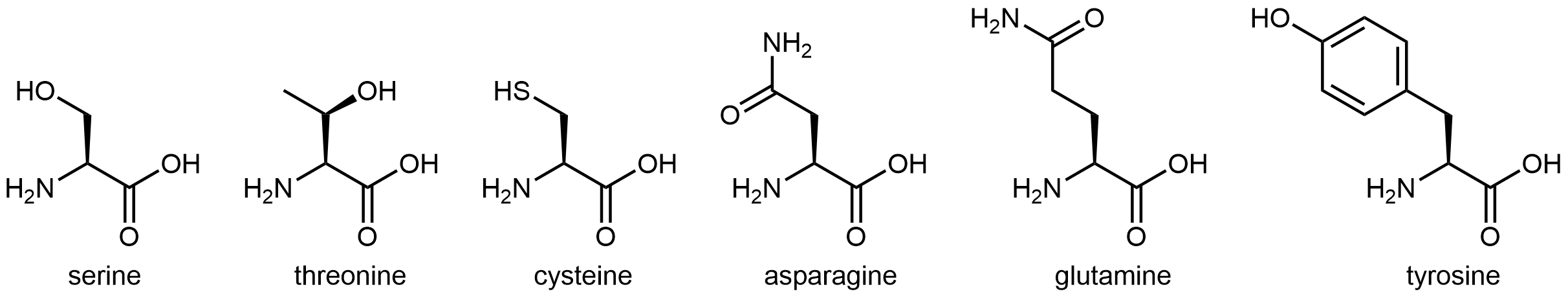
Los seis aminoácidos mostrados en la Figura 5 tienen cadenas laterales que son polares pero no ácidas ni básicas.
Los tres aminoácidos en la Figura 6 tienen cadenas laterales que contienen grupos funcionales que son básicos a pH neutro:
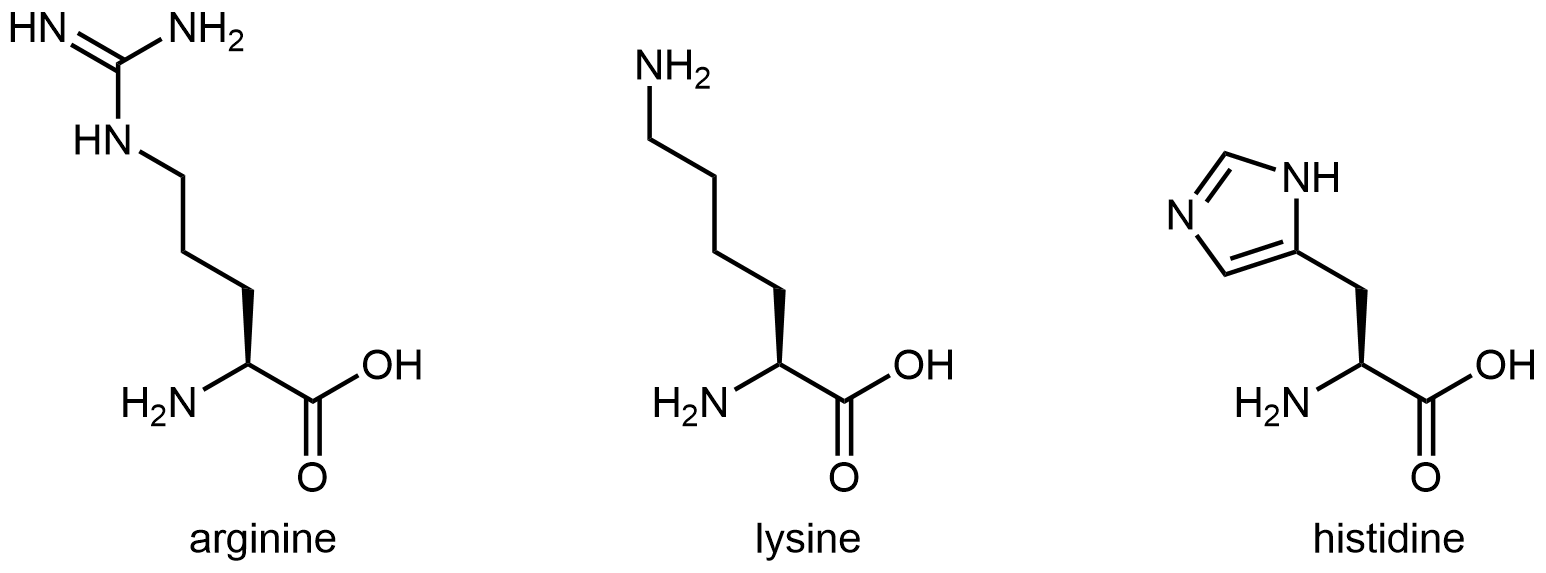
Actividad 1: ¿Qué Parte de la Cadena Lateral de Histidina es la Más Básica?
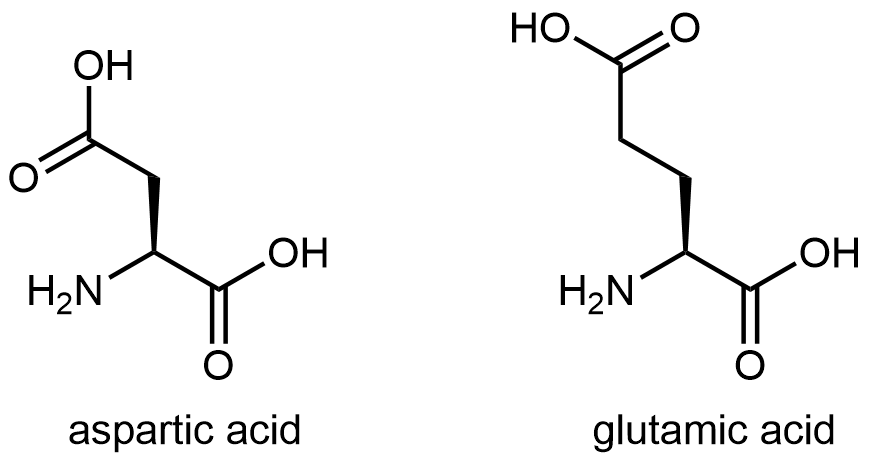
Y finalmente, la Figura 7 muestra dos aminoácidos que tienen cadenas laterales ácidas que contienen el grupo funcional ácido carboxílico:
Estructura de la proteína D15.6
Las moléculas proteicas son complicadas: hay 20 tipos diferentes de monómeros que se pueden unir para formar una gran cantidad de cadenas poliméricas diferentes. Así, las estructuras proteicas se clasifican de varias maneras. Fundamental es el orden en que se enlazan los monómeros: la estructura primaria. Las cadenas principales de proteínas largas pueden adoptar estructuras secundarias y terciarias tridimensionales únicas, las cuales dependen de la estructura primaria. Cuando dos o más cadenas proteicas de proteínas se mantienen unidas por fuerzas intermoleculares, también pueden adoptar estructuras cuaternarias específicas.
Estructura primaria
Una molécula de proteína es una poliamida (polipéptido), pero debido a que hay 20 monómeros de aminoácidos diferentes, una proteína es un copolímero sobre esteroides. Aunque la unidad repetitiva es la estructura de aminoácidos, hay 20 posibilidades para cada cadena lateral (resaltada en rojo a continuación):
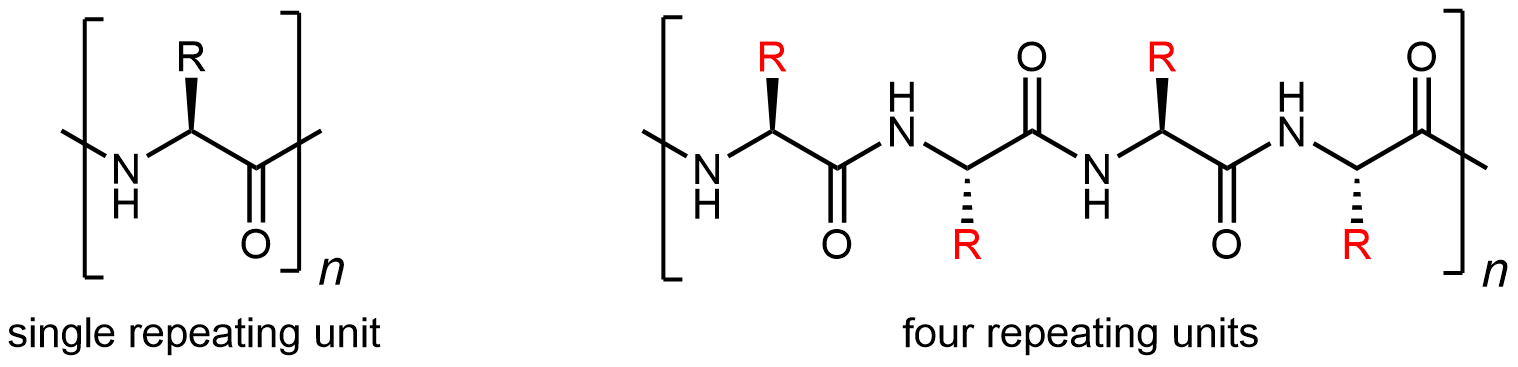
Por lo tanto, es posible un gran número de diferentes estructuras proteína-molécula. Cada estructura de copolímero posible tiene una secuencia única de cadenas laterales. Esta secuencia específica de aminoácidos se llama estructura primaria de la proteína.
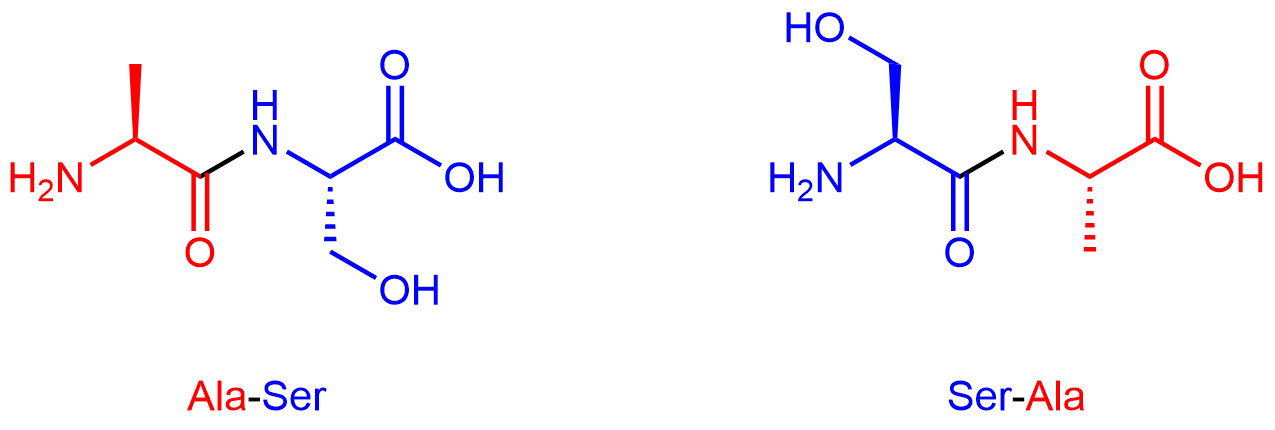
Por convención, las secuencias de proteínas se escriben desde el extremo con el grupo amina libre (el extremo N) hasta el extremo con el grupo carboxilo libre (el extremo C). Por ejemplo, Ala-Ser (alanina-serina) difiere de Ser-Ala (serina-alanina).
Ejercicio 2: Aminoácidos en un Tripéptido
Una tabla de estructuras de aminoácidos se puede encontrar en este enlace.
Estructura secundaria
La estructura secundaria de una proteína es la forma tridimensional de pequeños segmentos de proteínas. En las estructuras secundarias, los aminoácidos que no están uno al lado del otro a lo largo de la cadena principal están lo suficientemente cerca entre sí para formar enlaces de hidrógeno. El tipo de estructura secundaria se define por el patrón de enlaces de hidrógeno entre grupos amida que no son adyacentes en la cadena principal de la proteína. Dos elementos comunes de estructura secundaria, que están presentes en más del 60% de las proteínas conocidas, son la hélice α y la lámina beta.
La hélice α tiene una estructura helicoidal donde hay, en promedio, 3.6 aminoácidos por giro de la hélice. Todas las cadenas laterales se proyectan desde el eje helicoidal.
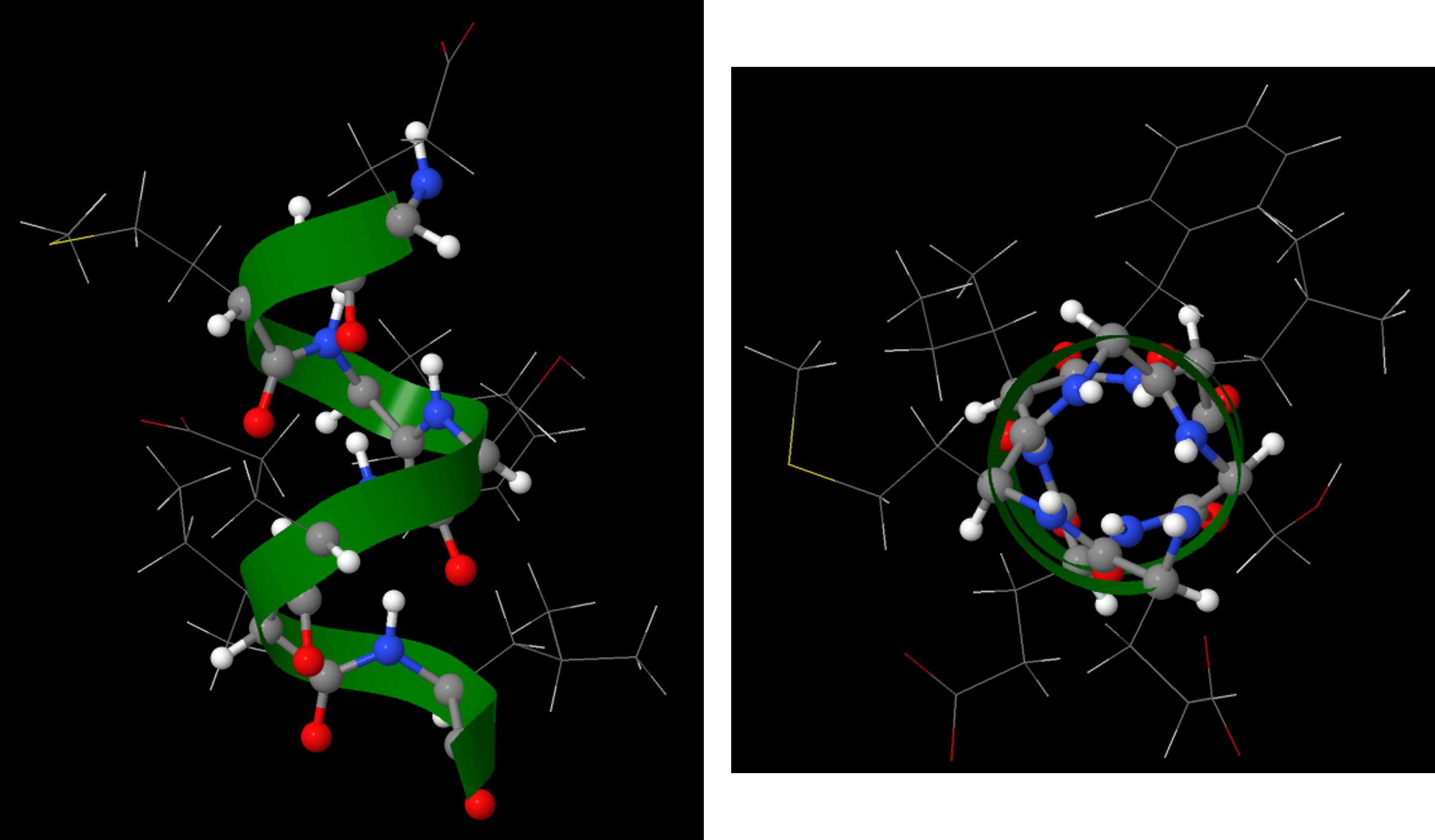
Una hélice α tiene un patrón regular de enlaces de hidrógeno entre los grupos amida de la cadena principal, donde el aminoácido n está unido por hidrógeno al aminoácido (n+4) th. Por ejemplo, como se muestra en la Figura 9, el C=O del aminoácido 1 forma un enlace de hidrógeno con N-H del aminoácido 5; el C=O del aminoácido 2 forma un enlace de hidrógeno con N-H del aminoácido 6; etc. Este enlace de hidrógeno que se repite regularmente es una característica prominente de una hélice α.
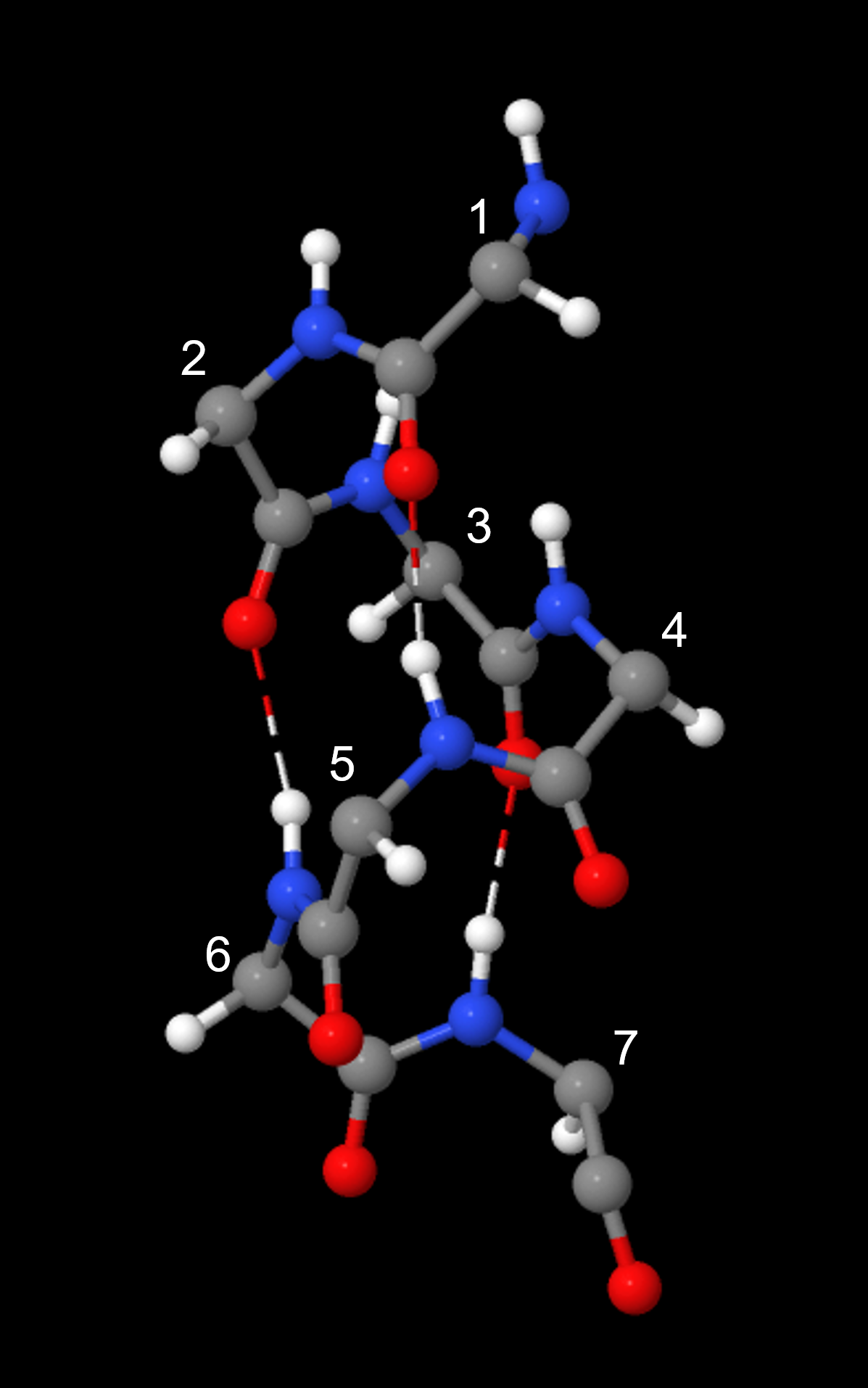
La lámina β o lámina plisada beta consiste en tramos de la cadena proteica conectados lado a lado por enlaces de hidrógeno entre los grupos amida de la cadena principal.
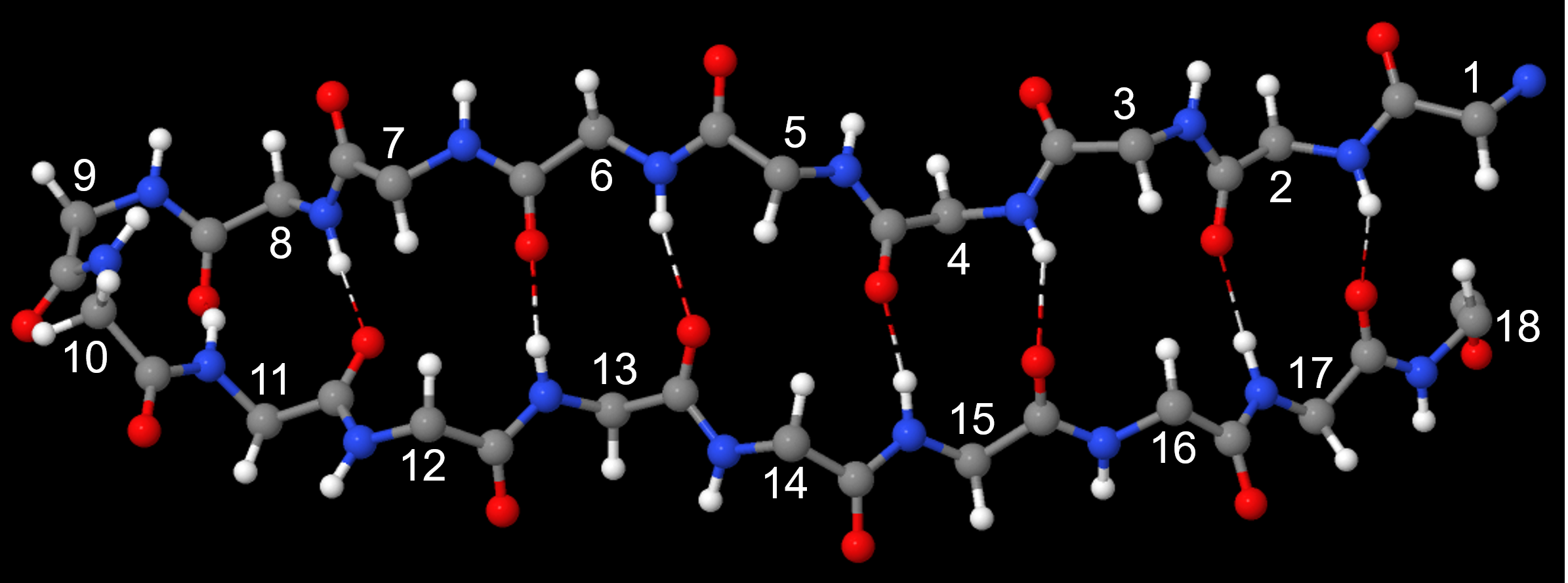
Dicho enlace de hidrógeno de la cadena principal da lugar a una apariencia general de lámina plisada, de ahí el nombre. Las cadenas laterales de los aminoácidos se alternan por encima y por debajo de la lámina β (Figura 11).
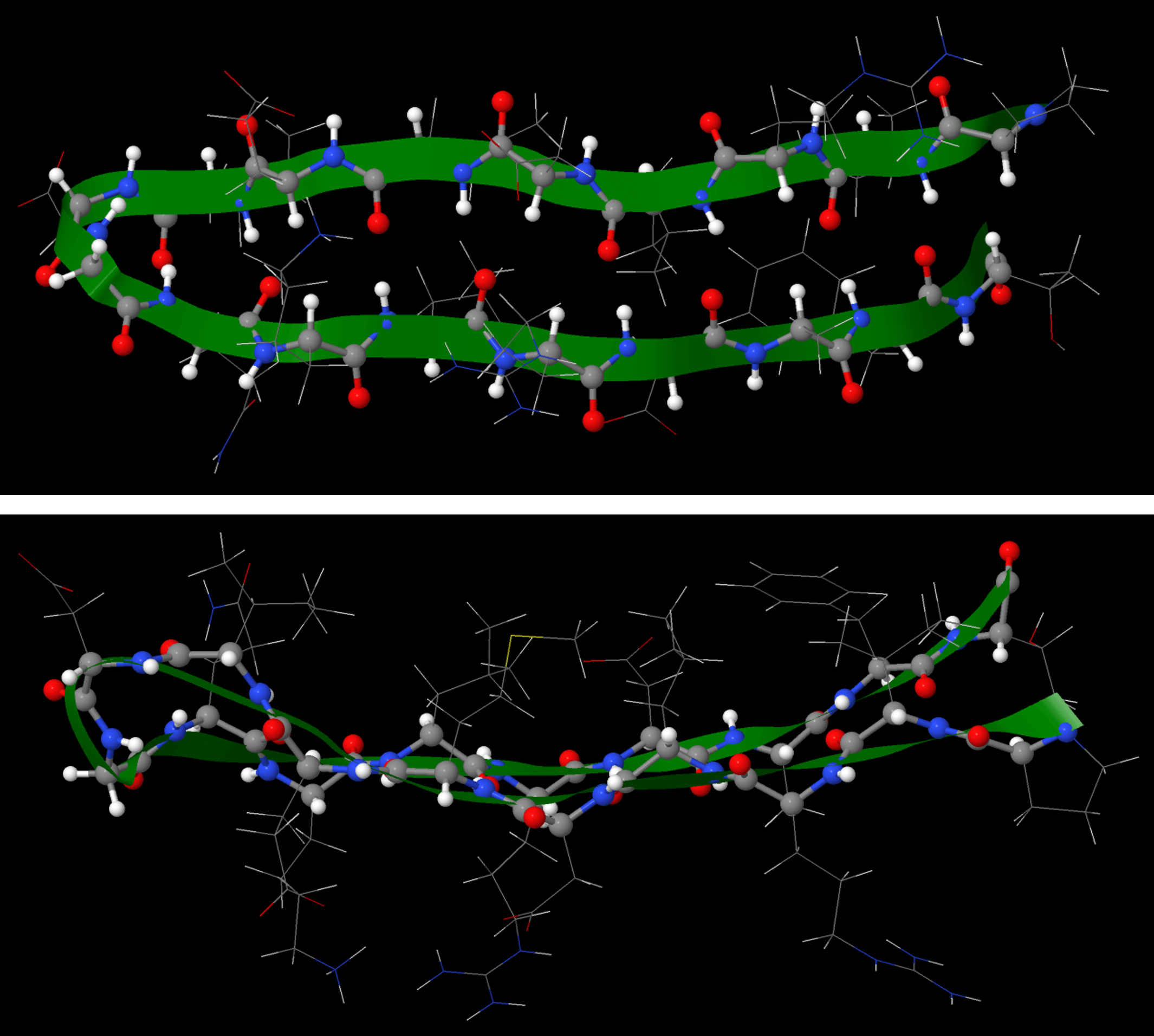
Los numerosos enlaces de hidrógeno de la cadena principal estabilizan tanto la hélice α como las estructuras de lámina β. Estas estructuras pueden combinarse para formar varias estructuras más grandes, llamadas motivos, que se encuentran en más de una estructura proteica.
Estructura Terciaria
La estructura terciaria de una proteína es la forma tridimensional general de la proteína. Es como α-hélices, β-láminas y motivos se unen para formar la estructura general. Las estructuras terciarias a menudo se definen por diversas interacciones intermoleculares que involucran las cadenas laterales de la proteína, así como el entorno en el que se encuentra la proteína.
Dos tipos generales de proteínas se encuentran en las células, las proteínas solubles en agua e insolubles en agua. Las proteínas solubles en agua, que incluyen enzimas y proteínas de transporte, se encuentran libres en compartimentos celulares como el citoplasma, núcleo o retículo endoplásmico.
En la estructura terciaria de las proteínas solubles en agua, la mayoría de los grupos de cadena lateral hidrófobos se encuentran en el interior de la proteína y lejos del agua, mientras que la mayoría de los grupos polares de cadena lateral se mantienen principalmente en el exterior y por lo tanto expuestos al (Los grupos amida de la cadena principal, aunque son polares y capaces de formar enlaces de hidrógeno, no interactúan mucho con el agua porque ya están comprometidos en enlaces de hidrógeno entre sí en hélices α y láminas β).
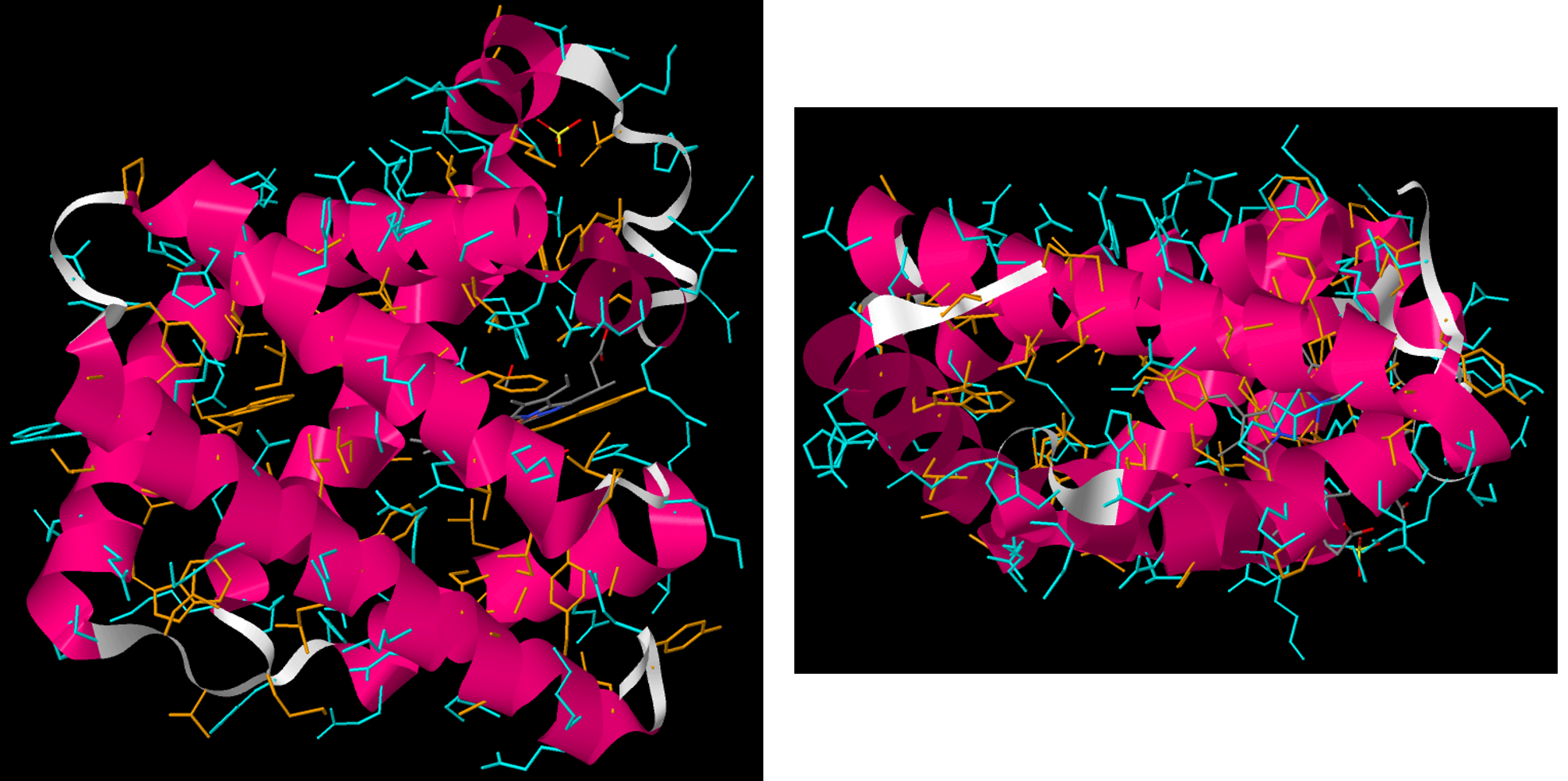
Las proteínas insolubles en agua incluyen canales de membrana, bombas y receptores. Estas proteínas se encuentran en bicapas lipídicas. Una descripción de cómo y por qué estas proteínas están asociadas con las membranas se encuentra en la Sección D16.7.
Un determinante importante de la estructura terciaria en algunas proteínas es el enlace disulfuro. Es similar a la reticulación que hemos discutido para los polímeros de adición. Una proteína puede formar enlaces disulfuro cuando las cisteínas en diferentes partes de su cadena están unidas por un enlace covalente entre los átomos de azufre en sus cadenas laterales (Figura 13).
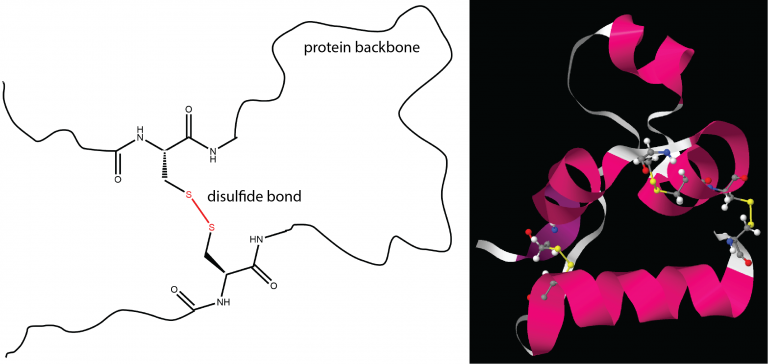 Figura 13. Enlaces disulfuro. Izquierda: Un enlace disulfuro (rojo) conecta dos cadenas laterales de cisteína a lo largo de la cadena principal de la proteína. Derecha: Tres enlaces disulfuro se muestran en amarillo. La columna vertebral se muestra como cintas magenta y las cisteínas se muestran como modelos de bola y varilla. Las seis cisteínas de esta proteína forman tres enlaces S-S. (PDB: 6RVA)
Figura 13. Enlaces disulfuro. Izquierda: Un enlace disulfuro (rojo) conecta dos cadenas laterales de cisteína a lo largo de la cadena principal de la proteína. Derecha: Tres enlaces disulfuro se muestran en amarillo. La columna vertebral se muestra como cintas magenta y las cisteínas se muestran como modelos de bola y varilla. Las seis cisteínas de esta proteína forman tres enlaces S-S. (PDB: 6RVA)
Estructura Cuaternaria
La estructura cuaternaria surge cuando hay más de una cadena proteica presente y las cadenas forman una estructura más grande. Por ejemplo, la hemoglobina en tu sangre está compuesta por cuatro cadenas (coloreadas magenta, azul, aguamarina y tan abajo) que forman dos subunidades.
Cuando el oxígeno se une a una subunidad de la hemoglobina, las interacciones entre las subunidades cambian. En otras palabras, la unión al oxígeno afecta la estructura cuaternaria. La estructura cuaternaria es el mecanismo habitual para los procesos conocidos como alosterio (en el que la unión de una molécula en un sitio sobre una proteína tiene efectos en otros sitios alejados de ese sitio) y cooperatividad (en la que la unión de una molécula en un sitio en una proteína de múltiples subunidades aumenta o disminuye la probabilidad de que otra molécula se una a otro sitio de la proteína).
D15.7 Plegamiento y desnaturalización de proteínas
¿Cómo se pliegan las proteínas en las complicadas estructuras tridimensionales? Se trata de un área muy activa de investigación en química y bioquímica en estos momentos. El primer indicio vino del trabajo de Christian Anfinsen sobre la proteína ribonucleasa, que descompone las moléculas de ARN. Anfinsen descubrió que después de tratar ribonucleasa con altas concentraciones de ciertos químicos que hacen que las proteínas se desplieguen y pierdan su estructura terciaria y secundaria, la ribonucleasa ya no descomponía el ARN. Además, si se eliminaran los productos químicos, la ribonucleasa recuperaría espontáneamente casi toda su actividad hidrolizante de ARN, sin necesidad de ningún otro componente celular. Anfinsen concluyó que la estructura primaria de una proteína determina completamente su estructura tridimensional en los niveles secundario, terciario y cuaternario.
Los científicos todavía están tratando de aprender cómo la estructura primaria de una proteína determina sus otros niveles de estructura. Han determinado que las fuerzas primarias que estabilizan la estructura tridimensional de una proteína son:
- Secuestro de cadenas laterales hidrófobas lejos del agua (por ejemplo, en el interior de proteínas solubles en agua)
- Maximizar las interacciones de dispersión de Londres (minimizando los espacios abiertos) en el interior de las proteínas
- Maximizar los enlaces de hidrógeno (por ejemplo, en hélices α-o β-láminas)
- Las atracciones entre los sitios con carga negativa y positiva se formaron cuando las cadenas laterales ácidas y básicas pierden y ganan iones H +
El proceso que utilizó Anfinsen se denomina desnaturalización, en el que las proteínas pierden las estructuras cuaternarias, terciarias y secundarias que están presentes en su estado nativo. Las proteínas pueden desnaturalizarse mediante la aplicación de algún estrés externo o compuesto tal como un ácido o base fuerte, una sal inorgánica concentrada, un disolvente orgánico (por ejemplo, alcohol o cloroformo), radiación o calor. Las proteínas desnaturalizadas pueden presentar una amplia gama de características, desde el cambio conformacional y la pérdida de solubilidad hasta la agregación debido a la exposición de grupos hidrófobos.
El plegamiento de proteínas es clave para saber si una proteína puede hacer su trabajo correctamente; debe doblarse en la forma correcta para funcionar. Sin embargo, los enlaces de hidrógeno, que juegan un papel importante en el plegamiento, son débiles en comparación con los enlaces covalentes y, por lo tanto, pueden verse afectados fácilmente por el calor, la acidez, las concentraciones variables de sal y otras tensiones. Esta es una de las razones por las que la homeostasis es fisiológicamente necesaria en muchas formas de vida.
https://mediaspace.wisc.edu/id/0_o4v8ja01?playerId=25717641
Día 15 Pre-clase Podia Problema: Nylon
La unidad repetitiva de nylon-6,10 tiene esta estructura.

Responde estas preguntas:
- Escribir las estructuras de dos monómeros que podrían combinarse para formar nylon-6,10.
- Enumere todas las fuerzas intermoleculares que existen entre las cadenas poliméricas en nylon-6,10.
- Dependiendo de la orientación de las cadenas poliméricas adyacentes, las fuerzas intermoleculares entre las cadenas de nylon-6,10 podrían ser más fuertes o más débiles. Dibujar estructuras de dos nylon-6,10 unidades repetitivas, una arriba y otra debajo de la estructura dada. Orienta las estructuras que dibujas para maximizar las fuerzas intermoleculares.
- Indique en su dibujo qué tipos de fuerzas intermoleculares son importantes entre qué partes de las moléculas. Además, escribe una explicación en palabras describiendo qué fuerzas son importantes dónde y por qué.
Dos días antes de la próxima sesión de toda la clase, esta pregunta de Podia se pondrá en vivo en Podia, donde podrás enviar tu respuesta.
Comentarios. Si encontró alguna inconsistencia, error u otras cosas que le gustaría informar sobre este módulo, utilice este enlace para reportarlos. Se incluirá un enlace similar en el material de cada día. Agradecemos sus comentarios.


