3.2: Niveles (Órdenes) de Estructura Proteínica
- Page ID
- 54622
A. Estructura primaria
1. L aminoácidos y la cadena principal polipeptídica C-N-C-N-...
La estructura primaria de una proteína se refiere a la secuencia de aminoácidos de su (s) cadena (s) de polipéptido (s). Las células utilizan solo 20 aminoácidos para elaborar polipéptidos y proteínas, aunque sí utilizan algunos aminoácidos adicionales para otros fines. Los enlaces peptídicos entre aminoácidos en polipéptidos se forman en reacciones de condensación en células durante la síntesis de proteínas (es decir, traducción). Los enlaces implican múltiples enlaces covalentes. Se rompen y reordenan entre los grupos carboxilo y amino de los aminoácidos durante la formación del enlace.
A continuación se muestran los 20 aminoácidos que se encuentran en las proteínas.
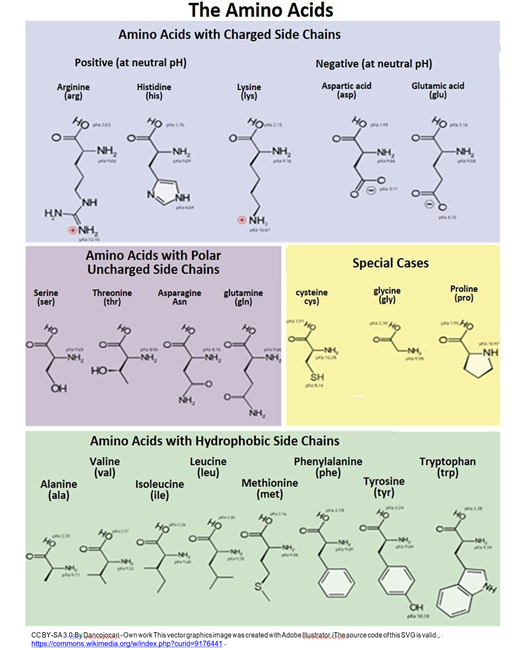
A excepción de la glicina, el carbono a en los otros 19 aminoácidos está unido a cuatro grupos diferentes, haciéndolos quirales u ópticamente activos.
Recordemos que los carbonos quirales permiten la imagen especular D y L o los isómeros ópticos d y l. Recordemos también que sólo la minúscula d o l define las propiedades ópticas de los isómeros. Solo para hacer la vida interesante, los aminoácidos L son en realidad dextrorrotarios en un polarímetro, ¡convirtiéndolos en d-aminoácidos! Si bien ambos enantiómeros existen en las células, solo los aminoácidos dextrorrotarios d (es decir, L) (junto con la glicina) son utilizados por las células para construir polipéptidos y proteínas. A continuación se ilustra un polipéptido parcial.
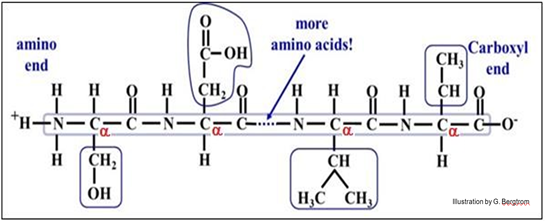
El resultado de la traducción en una célula es una cadena polipeptídica con un extremo carboxilo y un extremo amino. Las cadenas laterales de aminoácidos (en círculo arriba) terminan alternando en lados opuestos de una cadena principal polipeptídica C-N-C-N-... debido a los ángulos de enlace covalente a lo largo de la cadena principal. Podrías probarlo a ti mismo ensamblando un polipéptido corto con un kit de modelado molecular, ¡del tipo que podrías haber usado en una clase de química! La cadena principal C-N-C-N-... es la base subyacente de órdenes superiores, o niveles de estructura proteica (ver más adelante).
132 Secuencia de Aminoácidos y Estructura Primaria de Proteína
2. Determinación de la estructura primaria de proteínas - Secuenciación
Frederick Sanger fue el primero en demostrar un método práctico para secuenciar proteínas cuando reportó la secuencia de aminoácidos de los dos polipéptidos de la insulina bovina (vaca). Brevemente, la técnica implica la hidrólisis escalonada de fragmentos polipeptídicos, llamada Degradación de Edman. Cada hidrólisis deja atrás un fragmento de polipéptido acortado por un aminoácido que puede ser identificado. Sanger recibió un Premio Nobel en 1958 por esta hazaña. Por convención, la presentación y recuento de aminoácidos siempre comienza en el extremo amino o N-terminal (el extremo con un grupo NH2 libre). La estructura primaria es dictada directamente por el gen que codifica la proteína. Después de la transcripción de un gen), un ribosoma traduce el ARNm resultante en un polipéptido.
Desde hace algún tiempo, la secuenciación del ADN ha reemplazado a la mayoría de las secuencias proteicas directas. El método de secuenciación del ADN, coloquialmente conocido como el método didesoxi de Sanger, se generalizó rápidamente y finalmente se automatizó, permitiendo una secuenciación rápida de genes (e incluso del genoma completo). Ahora, en lugar de secuenciar directamente polipéptidos, podemos inferir secuencias de aminoácidos a partir de secuencias génicas aisladas por clonación o reveladas después de proyectos completos de secuenciación del genoma. Este es el mismo Sanger que primero secuenció proteínas, y sí..., ¡ganó un segundo Premio Nobel por el trabajo de secuenciación de ADN en 1980!
Las diferentes propiedades físicas y químicas de los propios aminoácidos resultan de las cadenas laterales en sus a-carbonos. Las propiedades físicas y químicas únicas de los polipéptidos y proteínas están determinadas por su combinación única de cadenas laterales de aminoácidos y sus interacciones dentro y entre polipéptidos. De esta manera, la estructura primaria refleja los fundamentos genéticos de la función polipeptídica y proteica. Las estructuras de orden superior que dan cuenta de los motivos funcionales y dominios de una proteína madura derivan de su estructura primaria. Christian Anfinsen ganó la mitad del Premio Nobel de Química de 1972 por demostrar que este fue el caso de la enzima ribonucleasa (Stanford Moore y William H. Stein obtuvieron su parte del premio por relacionar la estructura del sitio activo de la enzima con su función catalítica). Ver 1972 Premio Nobel de Química para más.
B. Estructura secundaria
La estructura secundaria se refiere a estructuras locales altamente regulares dentro de un polipéptido (por ejemplo, una hélice) y dentro o entre polipéptidos (láminas b-plisadas). Linus Pauling y sus compañeros de trabajo sugirieron estos dos tipos de estructura secundaria en 1951. ¡Un poco de historia de Linus Pauling sería relevante aquí! Para 1932, Pauling había desarrollado su Escala de Electronegatividad de los elementos que podían predecir la fuerza de los enlaces atómicos en las moléculas. Contribuyó mucho a nuestra comprensión de los orbitales atómicos y más tarde a la estructura de las moléculas biológicas. Obtuvo el Premio Nobel de Química 1954 por esta obra. Él y sus colegas descubrieron más tarde que la anemia falciforme se debía a una hemoglobina anormal, y pasaron a predecir la estructura secundaria alfa helicoidal y plisada de las proteínas. Si bien no obtuvo un segundo Nobel por estos novedosos estudios de genética molecular, sí ganó el premio Nobel de la Paz de 1962 por convencer a casi 10 mil científicos para que solicitaran a las Naciones Unidas que votaran para prohibir las pruebas de bombas nucleares atmosféricas. ¡Vale la pena leer una reseña más detallada de su extraordinaria vida (por ejemplo, en Linus Pauling-Short Biography)!
Las conformaciones de la estructura secundaria ocurren debido a la formación espontánea de enlaces de hidrógeno entre grupos amino y oxígenos a lo largo de la cadena principal del polipéptido, como se muestra en los dos paneles de la izquierda en el siguiente dibujo Tenga en cuenta que las cadenas laterales de aminoácidos no juegan un papel significativo en la estructura secundaria.
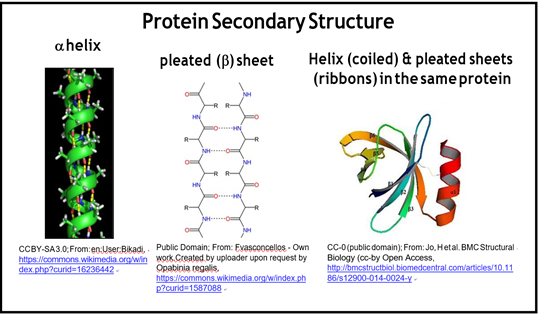
133 Estructura secundaria de proteínas
La hélice a o las láminas b son una disposición más estable de enlaces H en la (s) cadena (s). Estas regiones de estructura secundaria ordenada en un polipéptido pueden separarse por longitudes variables de péptidos menos estructurados llamados bobinas aleatorias. Los tres elementos de estructura secundaria pueden ocurrir en un solo polipéptido o proteína que se ha plegado en su estructura terciaria, como se muestra a la derecha en la ilustración anterior. Las hojas plisadas se muestran como cintas con puntas de flecha que representan la polaridad de N a C o C a N de las hojas. Como puedes ver, un par de regiones peptídicas que forman una lámina plisada pueden hacerlo ya sea en las direcciones paralelas o antiparalelas (mira las puntas de flecha de las cintas), lo que dependerá de otras influencias que dicten el plegamiento de proteínas para formar una estructura terciaria. Algunos polipéptidos nunca van más allá de su estructura secundaria, permaneciendo fibrosos e insolubles. La queratina es quizás el ejemplo más conocido de una proteína fibrosa, que compone el pelo, las uñas de las manos, las plumas de los pájaros e incluso los filamentos del citoesqueleto. Sin embargo, la mayoría de los polipéptidos y proteínas se pliegan y asumen estructura terciaria, convirtiéndose en proteínas globulares solubles.
C. Estructura terciaria
Los polipéptidos adquieren su estructura terciaria cuando las cadenas laterales hidrófobas y no polares se unen espontáneamente para excluir el agua, ayudados por la formación de puentes salinos y enlaces H entre cadenas laterales polares que se encuentran dentro del polipéptido globular. De esta manera, las hélices a o b se pliegan y se incorporan en formas globulares. Las fuerzas que cooperan para formar y estabilizar estructuras tridimensionales de polipéptidos y proteínas se ilustran a continuación.
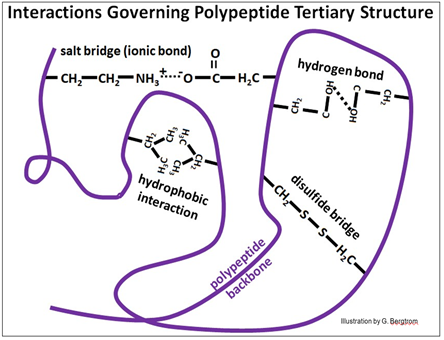
Las cadenas laterales polares (hidrófilas) que no pueden encontrar otras parejas de cadena lateral se encuentran típicamente en la superficie externa del '' glóbulo ', donde interactúan con el agua y así disuelven la proteína (recuerda el agua de hidratación). Basadas en enlaces no covalentes, las estructuras terciarias son fuertes simplemente por el gran número de interacciones que de otro modo serían débiles que las forman. No obstante, los enlaces disulfuro covalentes entre los aminoácidos de cisteína en el polipéptido (mostrado anteriormente) pueden estabilizar aún más la estructura terciaria. Los enlaces disulfuro (puentes) se forman cuando cisteínas muy separadas en la estructura primaria de la molécula terminan cerca una de la otra en un polipéptido plegado. Luego se oxidan los grupos -SH (sulfhidrilo) en las cadenas laterales de cisteína, formando los enlaces disulfuro (—S-S-).
La reacción de oxidación de sulfhidrilo se muestra a continuación.
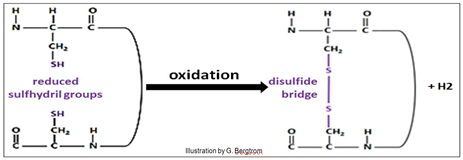
134 Proteína Terciaria (30) Estructura
135 Puentes Disulfuro Estabilizan 30 Estructura
Para comprender mejor cómo los puentes disulfuro pueden soportar la estructura tridimensional de una proteína, solo imagine su entorno físico y químico. Cambiar la temperatura o la concentración de sal que rodea a una proteína podría alterar los enlaces no covalentes involucrados en la forma 3D de la proteína activa. No afectados por estos cambios, los puentes disulfuro limitan la interrupción y permiten que la proteína se vuelva a plegar correcta y rápidamente cuando las condiciones vuelvan a la normalidad (¡piense en la homeostasis!).


