8.3: Memoria de codificación en un HMM- Detección de islas CpG
- Page ID
- 54719
Las islas CpG se definen como regiones dentro de un genoma que están enriquecidas con pares de nucleótidos C y G en la misma cadena. Por lo general, cuando este dinucleótido está presente dentro de un genoma, éste se metila, y cuando se produce la desaminación de la citosina, como lo hace con alguna frecuencia base, se convierte en una timina, otro nucleótido natural, y así no puede ser reconocido tan fácilmente por la célula como una mutación, provocando una mutación de C a T. Esta mayor frecuencia de mutación en las islas CpG agota las islas CpG a lo largo del tiempo evolutivo y las vuelve relativamente raras. Debido a que la metilación puede ocurrir en cualquiera de las cadenas, los CpG generalmente mutan en un TpG o un CpA. Sin embargo, cuando se sitúa dentro de un promotor activo, se suprime la metilación y los dinucleótidos CpG pueden persistir. De manera similar, los CPG en regiones importantes para la función celular se conservan debido a la presión evolutiva. Como resultado, la detección de islas CpG puede resaltar regiones promotoras, otras regiones transcripcionalmente activas o sitios de selección purificadora dentro de un genoma.
¿Sabías?
CpG significa [C] ytosina - [p] columna vertebral de hofosfato - [G] uanina. La 'p' implica que nos estamos refiriendo a la misma hebra de la doble hélice, en lugar de un par de bases G-C que ocurre a través de la hélice.
Dada su importancia biológica, las islas CpG son las principales candidatas para modelar. Inicialmente, se puede intentar identificar estas islas explorando el genoma para intervalos fijos ricos en GC. La eficacia de este enfoque se ve socavada por la selección de un tamaño de ventana apropiado; mientras que una ventana demasiado pequeña puede no capturar toda una isla CpG en particular, una ventana demasiado grande daría como resultado que faltaran muchas islas CpG más pequeñas pero genuinas. Examinar el genoma sobre una base por codón también conduce a dificultades porque los pares CpG no necesariamente codifican aminoácidos y, por lo tanto, pueden no estar dentro de un solo codón. En cambio, los HMM son mucho más adecuados para modelar este escenario porque, como veremos en breve en la sección sobre aprendizaje no supervisado, los HMM pueden adaptar sus parámetros subyacentes para maximizar su probabilidad.
No todos los HMM, sin embargo, son adecuados para esta tarea en particular. Un modelo HMM que solo considera las frecuencias de nucleótidos individuales de C y G no logrará capturar la naturaleza de las islas CpG. Considere uno de esos HMM con los dos siguientes estados ocultos:
• Estado '+' que representa islas CpG
• Estado '-': representando no islas
Cada uno de estos dos estados emite entonces bases A, C, G y T con cierta probabilidad. Aunque las islas CpG en este modelo pueden enriquecerse con C y G al aumentar sus respectivas probabilidades de emisión, este modelo no logrará capturar el hecho de que las C y G ocurren predominantemente en pares.
Debido a la propiedad de Markov que gobierna los HMM, la única información disponible en cada paso de tiempo debe estar contenida dentro del estado actual. Por lo tanto, para codificar la memoria dentro de una cadena de Markov, necesitamos aumentar el espacio estatal. Para ello, los estados individuales '+' y '-' pueden ser reemplazados por 4 estados '+' y 4 estados '-': A+, C+, G+, T+, A-, C-, G-, T- (Figura 8.4). Específicamente, hay 2 formas de modelar esto, y esta elección dará como resultado diferentes probabilidades de emisión:
- Un modelo sugiere que el estado A+, por ejemplo, implica que actualmente estamos en una isla CpG y el carácter anterior era un A. Las probabilidades de emisión aquí llevarán la mayor parte de la información y las transiciones serán bastante degeneradas.
- Otro modelo sugiere que el estado A+, por ejemplo, implica que actualmente estamos en una isla CpG y el carácter actual es un A. La probabilidad de emisión aquí será de 1 para A y 0 para todas las demás letras y las probabilidades de transición llevarán la mayor parte de la información en el modelo y las emisiones serán ser bastante degenerados. Vamos a asumir este modelo a partir de ahora.
¿Sabías?
El número de transiciones es el cuadrado del número de estados. Esto da una idea aproximada de cómo aumentar la escala de la “memoria” HMM (y por lo tanto los estados).
- La memoria de este sistema deriva del hecho de que cada estado sólo puede emitir un carácter y por lo tanto “recuerda” su carácter emitido. Además, la naturaleza dinucleotídica de las islas CpG se incorpora dentro de las matrices de transición. En particular, la frecuencia de transición de los estados C+ a G+ es significativamente mayor que de los estados C− a G−, lo que demuestra que estos pares ocurren con mayor frecuencia dentro de las islas.
FAQ
P: Dado que cada estado emite solo un personaje, ¿podemos decir entonces que esto se reduce a una Cadena Markov en lugar de una HMM?
A: No. A pesar de que las emisiones indican la letra del estado oculto, no indican si el estado es una isla CpG o no: tanto un estado A- como uno A+ emiten solo el A observable.
FAQ
P: ¿Cómo incorporamos nuestros conocimientos sobre el sistema mientras entrenamos modelos HMM, por ejemplo, algunas probabilidades de emisión de 0 en el caso de detección de isla CpG?
R: Podríamos forzar nuestro conocimiento sobre el modelo estableciendo algunos parámetros y dejando que otros varíen o podríamos dejar que el HMM se suelte en el modelo y dejar que descubra esas relaciones. De hecho, incluso hay métodos que simplifican el modelo al forzar que un subconjunto de parámetros sea 0 pero permitiendo que el HMM elija qué subconjunto.
Dado el marco anterior, podemos usar la decodificación posterior para analizar cada base dentro de un genoma y determinar si es muy probable que sea un constituyente de una isla CpG o no. Pero después de haber construido el modelo HMM expandido, ¿cómo podemos verificar que de hecho es mejor que el modelo de un solo nucleótido? Anteriormente demostramos que el algoritmo hacia adelante o hacia atrás se puede utilizar para calcular P (x) para un determinado
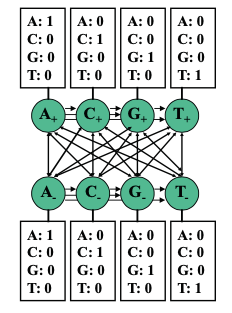
Figura 8.4: HMM para islas CpG
modelo. Si la probabilidad de nuestro conjunto de datos es mayor dado el segundo modelo que el primer modelo, lo más probable es que capture el comportamiento subyacente de manera más efectiva.
Sin embargo, existe un riesgo en complicar el modelo, que es el sobreajuste. Aumentar el número de parámetros para un HMM hace que el HMM sea más probable que sobreajuste los datos y sea menos preciso en la captura del comportamiento subyacente. Una solución común a esto en el aprendizaje automático es usar la regularización, que es esencialmente usar menos parámetros. En este caso, es posible reducir el número de parámetros a aprender al restringir que todas las probabilidades de transición +/- sean el mismo valor y todas las probabilidades de transición -/+ sean el mismo valor, ya que las transiciones de ida y vuelta de los estados + y - son lo que nos interesa modelar, y lo real bases donde ocurrió la transición no son tan importantes para nuestro modelo. Por lo tanto, para este modelo restringido tenemos que aprender menos parámetros lo que conduce a un modelo más simple y puede ayudar a evitar el sobreajuste.
FAQ
P: ¿Hay otras formas de codificar la memoria para la detección de islas CpG? R: Otras ideas con las que se puede experimentar incluyen
- Emitir dinucleótidos y encontrar una manera de lidiar con el solapamiento.
- Agregar un estado especial que va de C a G.


