24.2: Técnicas Experimentales
- Page ID
- 54758
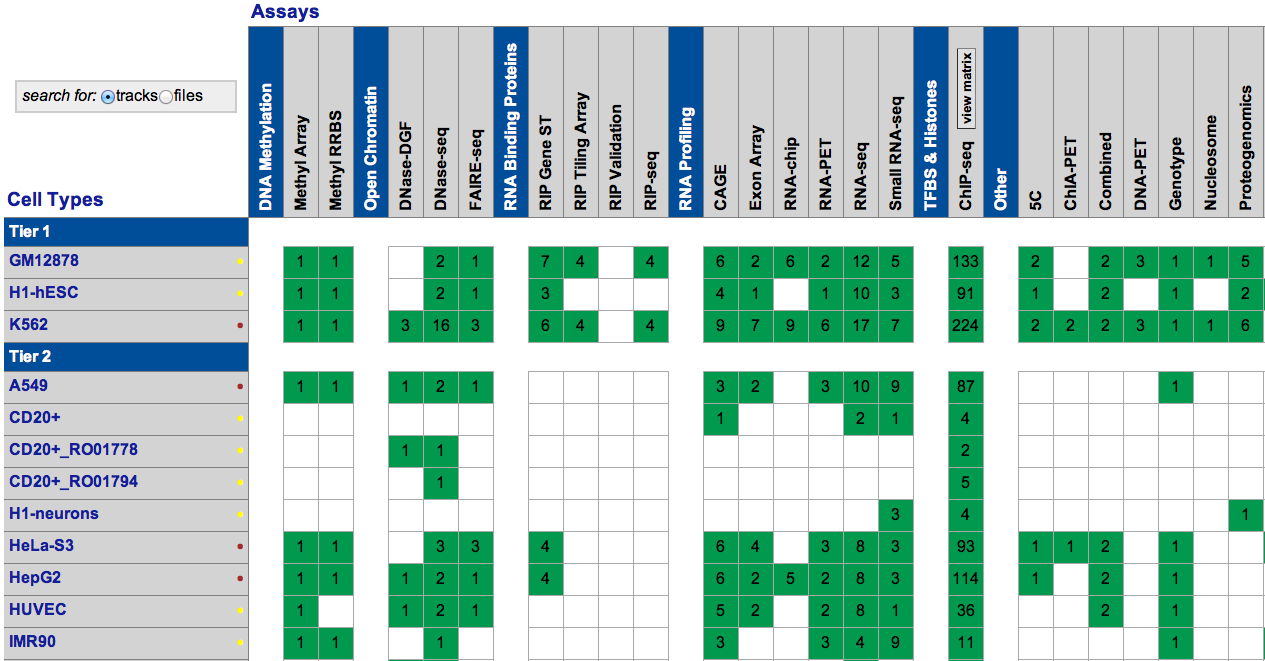
El proyecto ENCODE utilizó una amplia gama de técnicas experimentales, que van desde RNA-seq, Cage-seq, Exon Arrays, Maine-seq, Chromatin Chip-seq, DNase-seq y muchas más.
Proyecto ENCODE. Todos los derechos reservados. Este contenido está excluido de nuestro Creativo
Licencia Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
Una de las técnicas más importantes utilizadas fue Chip-seq (inmunoprecipitación de cromatina seguida de secuenciación). El primer paso en un experimento ChIP es apuntar a fragmentos de ADN asociados con una proteína específica. Esto se hace mediante el uso de un anti-cuerpo que se dirige a la proteína específica y se utiliza para inmunoprecipitar el complejo ADN-proteína. El paso final es ensayar el ADN. Esto determinará las secuencias unidas a las proteínas.
Chip-seq tiene varias ventajas sobre las técnicas anteriores (por ejemplo, Chip-chip). Por ejemplo, Chip-seq tiene resolución de un solo nucleótido y su alineabilidad aumenta con la longitud de lectura. Sin embargo, Chip-seq tiene varias desventajas. Los errores de secuenciación tienden a aumentar sustancialmente cerca del final de las lecturas. Además, con un bajo número de lecturas, la sensibilidad y especificidad tienden a disminuir al detectar regiones enriquecidas. Ambos problemas surgen al procesar los datos y muchas de las técnicas computacionales buscan rectificarlo.
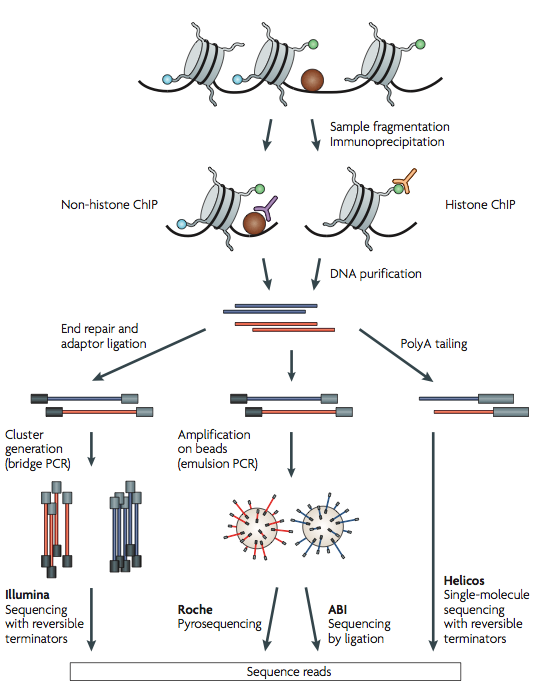
Cortesía de Macmillan Publishers Limited. Usado con permiso.
Fuente: Park, Peter J. “Chip-seq: Ventajas y Desafíos de una
Tecnología de Madurado”. Nature Reviews Genetics 10, núm. 10 (2009): 669-80

