11.10: ARN antisentido
- Page ID
- 57156
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)El ARN mensajero (ARNm) es monocatenario. Su secuencia de nucleótidos se llama “sentido” porque da como resultado un producto génico (proteína). Normalmente, sus nucleótidos desapareados son “leídos” mediante anticodones de ARN de transferencia a medida que el ribosoma procede a traducir el mensaje.

Sin embargo, el ARN puede formar dúplex tal como lo hace el ADN. Todo lo que se necesita es una segunda cadena de ARN cuya secuencia de bases sea complementaria a la primera hebra.
Ejemplo
5' C A U G 3' ARNm
3' G U A C 5' ARN antisentido
La segunda cadena se llama la cadena antisentido porque su secuencia de nucleótidos es el complemento del sentido del mensaje.
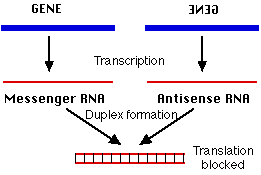
Cuando el ARNm forma un dúplex con una secuencia de ARN antisentido complementaria, se bloquea la traducción. Esto puede ocurrir porque el ribosoma no puede acceder a los nucleótidos en el ARNm o porque el ARN dúplex se degrada rápidamente por ribonucleasas en la célula. Con métodos de ADN recombinante, se pueden introducir genes sintéticos (ADN) que codifican moléculas de ARN antisentido en el organismo.
Ejemplos
El tomate Flavr Savr
La mayoría de los tomates que tienen que ser enviados al mercado se cosechan antes de madurar. De lo contrario, el etileno sintetizado por el tomate hace que maduren y se echen a perder antes de que lleguen al cliente. Se han construido tomates transgénicos que portan en su genoma un gen artificial (ADN) que se transcribe en un ARN antisentido complementario al ARNm para una enzima involucrada en la producción de etileno. Estos tomates producen sólo el 10% de la cantidad normal de la enzima.
El objetivo de este trabajo fue dotar a los tomates de supermercado algo más cercano a la apariencia y sabor de los tomates cosechados al madurar. Sin embargo, estos tomates a menudo se dañaron durante el envío y manejo y han sido retirados del mercado.
Tabaco transgénico

Fig.11.9.3 Flor de tabaco
Flor de una planta de tabaco que porta un transgén cuyo transcrito es antisentido a uno de los ARNm necesarios para la pigmentación normal de la flor.
Flor transgénica

Flor de otra planta transgénica que no logró alterar su pigmentación normal.
Elaboración de plantas transgénicas
Existen varios métodos para introducir genes en plantas, incluyendo
- infectar células vegetales con vectores plasmídicos que portan el gen deseado
- disparar gránulos microscópicos que contienen el gen directamente en la célula
A diferencia de los animales, no existe una distinción real entre células somáticas y células germinales. Tejidos somáticos de plantas, por ejemplo, células de raíz cultivadas en cultivo,
- se puede transformar en el laboratorio con el gen deseado
- crecido en plantas maduras con flores.
Si todo va bien, el transgén se incorporará al polen y a los huevos y se transmitirá a la siguiente generación.
En este sentido, es más fácil producir plantas transgénicas que animales transgénicos.
El ARN antisentido también se produce de forma natural
¿Las células contienen genes que se traducen naturalmente en moléculas de ARN antisentido capaces de bloquear la traducción de otros genes en la célula? La respuesta es sí, y estos parecen representar otro método de regulación de la expresión génica. Tanto en ratones como en humanos, el gen para el receptor del factor de crecimiento insulínico 2 (IgF2R) que se hereda del padre sintetiza un ARN antisentido que parece bloquear la síntesis del ARNm para IgF2R. Una diferencia heredada en la expresión de un gen dependiendo de si se hereda de la madre o del padre se llama impronta genómica o parental.
Interferencia de ARN (ARNi)
Al probar los efectos del ARN antisentido, se debe usar ARN sentido de la misma región codificante como control. Sorprendentemente, las preparaciones de ARN sentido a menudo resultan ser un inhibidor tan efectivo como el ARN antisentido.
¿Por qué? Parece que las preparaciones de ARN sentido a menudo están contaminadas con híbridos: hebras sentido y antisentido que forman una doble hélice de ARN bicatenario (ARNds). El ARN bicatenario correspondiente a un gen en particular es un poderoso supresor de ese gen. De hecho, el efecto supresor del ARN antisentido probablemente también depende de su capacidad para formar ARNbc (usando el ARNm correspondiente como molde).
La capacidad del ARNbc para suprimir la expresión de un gen correspondiente a su propia secuencia se denomina ARN de interferencia (ARNi). También se llama silenciamiento génico postranscripcional o PTGS.
Mecanismo de ARNi
Las únicas moléculas de ARN que normalmente se encuentran en el citoplasma de una célula son moléculas de ARN monocatenario. Si la célula encuentra moléculas de ARN bicatenario (ARNds), usa una enzima llamada Dicer para cortarlas en fragmentos que contienen ~21 pares de bases (~2 vueltas de una doble hélice). Luego, las dos cadenas de cada fragmento se separan, liberando la hebra antisentido. Con la ayuda de una proteína, se une a una secuencia sentido complementaria en una molécula de ARNm. Si el emparejamiento de bases es exacto, se destruye el ARNm. Por su acción, estos fragmentos de ARN se han denominado “ARN interferente pequeño (o corto)” (ARNip). El complejo de ARNip y proteína se llama el "R NA- i nduced s ilence c omplex” (RISC).
Los ARNIp también pueden interferir con la transcripción
Cada vez hay más evidencia de que los ARNIp también pueden inhibir la transcripción de genes
- tal vez mediante la unión a secuencias complementarias en el ADN o
- tal vez uniéndose al transcrito de ARN naciente a medida que se está formando.
En levaduras de fisión, al menos, el ARNip está complejado con una molécula de cada una de tres proteínas diferentes. Todo el complejo se llama complejo RITS (” RNA - i nduced iniciación de la ilenciación del gen t ranscripcional”)
Se desconoce cómo estos ARNIs —sintetizados en el citosol— obtienen acceso al ADN en el núcleo.
Las moléculas sintéticas de ARNip que se unen a promotores génicos pueden —en el laboratorio— reprimir la transcripción de ese gen. La represión está mediada por la metilación del ADN en el promotor y, tal vez, la metilación de las histonas en las proximidades.
Existe una cepa de arroz (LGC-1) que produce niveles anormalmente bajos de proteínas llamadas glutelinas. Resulta que de varios genes de glutelina encontrados en el arroz
- dos genes de glutelina estrechamente similares se localizan espalda con espalda en el mismo cromosoma.
- En LGC-1, se ha producido una deleción entre los dos genes que elimina la señal que normalmente detendría la transcripción después del primer gen.
- Así, la ARN polimerasa II se transcribe justo después del primer gen y en el segundo.
- El resultado es un ARN mensajero con secuencias casi idénticas que se ejecutan en direcciones opuestas.
- Esto hace que el ARNm se pliegue en una molécula de ARN bicatenario (ARNbc).
- Una enzima similar a Dicer corta el ARNbc en pequeños ARN interferentes (ARNip) que suprimen la transcripción adicional de esos genes, así como otros genes de glutelina.
¿Por qué ARNi?
Se ha encontrado que la ARNi opera en organismos tan diversos como plantas, hongos y animales como Drosophila melanogaster, Caenorhabditis elegans, e incluso ratones y peces cebra. Tal respuesta celular universal debe tener una función importante. ¿Qué podría ser?
Algunas posibilidades:
- Algunos virus tanto de plantas como de animales tienen un genoma de ARNbc. Y muchos otros virus tanto de plantas como de animales tienen un genoma de ARN que en la célula hospedadora se convierte brevemente en ARNbc. Por lo que la ARNi puede ser un arma para contrarrestar las infecciones por estos virus destruyendo sus ARNm y bloqueando así la síntesis de proteínas virales esenciales.
- Los transposones pueden transcribirse en moléculas de ARN con regiones que son bicatenarias. El ARNi podría entonces destruir estos.
- La interferencia de ARN puede ser el dividendo inesperado de otro proceso básico de control de la expresión génica.
ARNi como herramienta
En cualquier caso, el descubrimiento de ARNi agrega una herramienta prometedora a la caja de herramientas de los biólogos moleculares. La introducción del ARNbc correspondiente a un gen particular noqueará la propia expresión celular de ese gen. (Alimentar a C. elegans en E. coli que fabrica el ARNbc incluso hará el truco).
Ejemplo Heroico
En la edición del 24 de marzo de 2005 de Nature, Sönnichsen et al informaron que han inyectado ARNds correspondientes a 20,326 de los genes de C. elegans (¡98% del total!) y monitoreó el efecto de cada uno sobre el desarrollo embrionario desde la finalización de la meiosis (después de la fecundación) hasta la segunda división mitótica que produce el embrión de 4 células.
Encontraron que al menos 661 genes diferentes alteraron algún proceso durante este periodo:
- aproximadamente la mitad de ellos participan en la división celular y
- la mitad en el metabolismo celular general.
(Otros mil genes produjeron efectos fenotípicos que se observaron en etapas posteriores de desarrollo).
Debido a que la ARNi se puede hacer en tejidos particulares en un momento elegido, a menudo proporciona una ventaja sobre los “knockouts” de genes convencionales donde el gen faltante se porta en la línea germinal y por lo tanto cuya ausencia puede matar al embrión antes de que pueda estudiarse.
Otro ejemplo: detección de genes para determinar su efecto sobre la sensibilidad a los medicamentos
- Distribuya sus células en miles de pocillos y agregue, desde una “biblioteca” de miles de ARNip que representan todo el genoma, moléculas de ARNip dirigidas a la expresión de un gen a cada pocillo
- Añadir el medicamento a todos los pozos
- Ver qué pocillos tienen células que responden
Algunas otras aplicaciones prometedoras de ARNi
En células de mamíferos
En las células de mamíferos, la introducción de fragmentos de ARNbc solo reduce temporalmente la expresión génica. Sin embargo, las células de mamíferos pueden infectarse con un vector de ADN que codifica una molécula de ARN de 50-80 nucleótidos llamada “ARN de horquilla pequeña” (ARNhp) que contiene una secuencia correspondiente al gen que se desea suprimir. A medida que se sintetiza el ARNh, Dicer lo convierte en una molécula típica de ARNIp. Debido a que la célula puede sintetizar ARNh continuamente, la interferencia es duradera. De hecho, con vectores que se integran en el genoma del huésped, la ARNi puede transmitirse a los descendientes.
En plantas
El número del 19 de junio de 2003 de Nature informó sobre las plantas de café que fueron diseñadas para expresar un transgén que produce ARNip que interfiere —por ARNi— con la expresión de un gen necesario para producir cafeína. Entonces tal vez el café “descafeinado” algún día ya no requerirá la eliminación química de la cafeína de los granos de café.
Monsanto está desarrollando un maíz transgénico (maíz) que expresa un ARNds correspondiente a la secuencia de un gen esencial en el gusano de la raíz del maíz occidental, una plaga devastadora del cultivo. Después de ingerir este ARNbc, las propias células del insecto lo procesan en un ARNip que se dirige al ARNm del gen para su destrucción y mata al gusano en pocos días.
Amplificación de ARNi
En C. elegans, plantas y Neurospora, la introducción de algunas moléculas de ARNbc tiene un efecto potente y duradero. En las plantas, el silenciamiento génico se propaga a las células adyacentes (a través de plasmodesmas) e incluso a otras partes de la planta (a través del floema). La iARN dentro de una célula puede continuar después de la mitosis en la progenie de esa célula. La activación de ARNi en C. elegans puede incluso pasar a través de la línea germinal hacia sus descendientes.
Tal amplificación de una señal inicial de activación sugiere un efecto catalítico. Resulta que estos organismos tienen ARN polimerasas dependientes de ARN (RDRPs) que utilizan el ARNm dirigido por el ARNip antisentido inicial como molde para la síntesis de más ARNip. La síntesis de estos ARNIp “secundarios” incluso ocurre en regiones adyacentes del ARNm. Por lo tanto, estos ARNip secundarios no solo pueden dirigirse a áreas adicionales del ARNm original, sino que son potencialmente capaces de silenciar los ARNm de otros genes que pueden portar la misma secuencia de nucleótidos.
Este fenómeno, denominado “ARNi transitivo”,
- puede complicar la interpretación de los experimentos de supresión génica ya que la expresión de otros genes puede suprimirse además del gen diana;
- plantea una bandera de advertencia para el uso de ARNi para suprimir genes individuales en terapia humana (aunque no se han observado RDRP y amplificación en células de mamíferos).
ARNi en terapia humana
Debido a que su diana es tan específica, la posibilidad de usar ARNi para detener la expresión de un solo gen ha creado una gran emoción de que una nueva clase de agentes terapéuticos esté en el horizonte. Muchos ensayos clínicos están en marcha explorando el uso de moléculas de ARNip en el tratamiento de una amplia variedad de enfermedades. Hasta la fecha, los resultados más prometedores han sido el uso de ARNi para dirigirse a una enfermedad hereditaria en la que el hígado secreta una forma mutante de transtiretina que conduce a la acumulación de depósitos amiloides en las neuronas y en otros lugares.
MicroARN (miARN)
En C. elegans, el desarrollo exitoso a través de sus estadios larvarios y en el adulto requiere la presencia de al menos dos "microARN" (“miRNAs”), moléculas de ARN monocatenario que contienen aproximadamente 22 nucleótidos y por lo tanto aproximadamente del mismo tamaño que los ARNip.
Estos pequeños transcritos monocatenarios son generados por la escisión de precursores más grandes usando la versión C. elegans de Dicer.
Actúan destruyendo o inhibiendo la traducción de varios ARN mensajeros en el gusano (generalmente uniéndose a una región de secuencia complementaria en la región 3' no traducida [3'-UTR] del ARNm).
Los microARN (miARN) en C. elegans (que primero se llamaron “pequeños ARN temporales”) resultan ser representantes de una gran clase de ARN que son codificados por los propios genes del organismo.
- El producto inicial de la transcripción génica es una molécula grande llamada pr i -miARN.
- Mientras aún está dentro del núcleo, una enzima llamada Drosher corta el pri-miARN en una molécula más corta (~70 nucleótidos) llamada pr e -miARN.
- El pre-miARN se exporta al citosol donde se escinde (por Dicer en animales) en el miARN.
MicroRNAs
- se encuentran en todos los animales (los humanos generan unos 1000 miARN) y plantas pero no en hongos.
- contienen 19—25 nucleótidos;
- están codificados en el genoma
- algunos por genes independientes (que pueden codificar varios miARN)
- algunas por porciones de un intrón del gen cuyo ARNm regularán.
- puede expresarse en
- solo ciertos tipos de células y
- sólo en ciertos momentos en la diferenciación de un tipo celular particular.
Si bien queda por descubrir la evidencia directa de la función de muchos de estos productos génicos recién descubiertos, regulan la expresión génica regulando el ARN mensajero (ARNm), ya sea
- destruir el ARNm cuando las secuencias coinciden exactamente (la situación habitual en las plantas) o
- reprimiendo su traducción cuando las secuencias son sólo una coincidencia parcial. En este último caso, probablemente requiera varios miARN para unirse simultáneamente en la 3'-UTR.
Los microARN tienen dos rasgos ideales para esto:
- Al ser tan pequeños, se pueden transcribir rápidamente a partir de sus genes.
- No necesitan traducirse en un producto proteico para actuar (en contraste, por ejemplo, con factores de transcripción).
Los microARN regulan (reprimen) la expresión de genes en mamíferos también. El análisis del genoma ha revelado miles de genes humanos cuyos transcritos (m ARN) contienen secuencias a las que uno o más de nuestros ARN mi podrían unirse. Probablemente cada miARN puede unirse a hasta 200 dianas de ARNm diferentes, mientras que cada ARNm tiene sitios de unión para múltiples miARN. Tal sistema proporciona muchas oportunidades para la traducción coordinada de ARNm.
Un estudio reportado en Nature (Lim, et al. , 433: 769, 17 feb 2005) utilizaron análisis de chips de ADN para mostrar que cuando un miARN particular se expresaba en células HeLa,
- un miARN normalmente expresado en el cerebro reprimió la producción de ARNm por 174 genes diferentes, mientras
- un miARN normalmente expresado en el músculo cardíaco y esquelético reprimió la producción de ARNm por 96 genes, todos menos 8 de ellos diferentes de los reprimidos por el miARN cerebral.
A medida que el trabajo avanza rápidamente en este campo, el patrón que comienza a emerger es el siguiente:
- Muchos genes —especialmente aquellos involucrados en tales actividades de mantenimiento (por ejemplo, respiración celular) comunes a todas las células— no tienen 3'-UTR que puedan ser bloqueadas por cualquiera de los miARN codificados en el genoma.
- Los genes que deben expresarse en un tipo particular de célula diferenciada y/o en un momento determinado de la vida de esa célula
- no expresan ninguno de los genes miARN que podrían bloquear su expresión pero
- expresan genes miARN que bloquean la expresión de otros genes para funciones especializadas que no serían apropiadas en esa célula en ese momento.
- En lugar de ser simples interruptores que activan o desactivan la expresión génica, los miARN parecen ejercer un efecto más sutil: elevar o disminuir el nivel de expresión génica (al igual que los factores de transcripción de proteínas).
Así, la represión de la expresión génica por miARN parece ser un mecanismo para asegurar la expresión génica regulada y coordinada a medida que las células se diferencian a lo largo de caminos particulares Por ejemplo, cuando los genes cigotos comienzan a encenderse en la blástula de pez cebra, uno de ellos codifica un miARN que desencadena la destrucción de los ARNm maternos que han estado ejecutando las cosas hasta entonces.
Por lo tanto, los miARN pueden desempeñar un papel tan importante como los factores de transcripción en la regulación y coordinación de la expresión de múltiples genes en un tipo particular de célula en momentos particulares.
¿Los miARN terapéuticos?
La facilidad con la que los miARN pueden introducirse en las células y sus efectos generalizados sobre la expresión génica han dado lugar a esperanzas de que podrían ser útiles en el control de trastornos genéticos, por ejemplo, cáncer.
A la fecha, algunos estudios de laboratorio han sido bastante prometedores.
- Un miARN que bloquea la expresión de las ciclinas en fase G 1 y S —y así detiene el ciclo celular en sus huellas— protege a los ratones del cáncer de hígado.
- un miARN que inhibe los genes necesarios para la metástasis suprime la metástasis de células de cáncer de mama humano tratadas.
Resumen
Además de los factores de transcripción de proteínas, los eucariotas utilizan pequeñas moléculas de ARN para regular la expresión génica —casi siempre reprimiéndola— por lo que el fenómeno se denomina silenciamiento de ARN.
Hay dos fuentes de pequeñas moléculas de ARN:
- ARN interferentes pequeños (ARNIp)
- Las células vegetales los producen a partir del ARN bicatenario (ARNbc) de los virus invasores.
- Científicos y compañías farmacéuticas hacen estos como agentes para desactivar la expresión de genes específicos (llamados ARN de interferencia o ARNi).
- micro ARN (miARN s)
- Estos están codificados en los genomas de todas las plantas y animales.
- Tanto los ARNIp como los miARN se procesan de la misma manera en el citosol de la célula.
- Ambos son generados por Dicer.
- Ambos se incorporan en un R NA- i nduced s ilenciante c omplex (RISC).
- Si la secuencia de nucleótidos del ARN pequeño coincide exactamente con la del ARNm, el ARNm se corta y destruye.
- Si solo hay una coincidencia parcial (generalmente en su 3' UTR), se reprime la traducción (es decir, síntesis de proteínas). Ambas actividades tienen lugar en el citosol —quizás en los cuerpos P.
- Sin embargo, para algunos ARN pequeños, el complejo RISC ingresa al núcleo y desactiva la transcripción del (de los) gen (s) correspondiente (s)
- unión a la secuencia de ADN desenrollada (o quizás al transcrito de ARN a medida que se está formando)
- convertir la eucromatina en heterocromática
- metilación de la lisina-9 histona H3 en los nucleosomas alrededor del gen (es)
Aparte de su uso como herramientas de laboratorio —y quizás terapéuticas—, los ARN pequeños son claramente esenciales para los organismos que los fabrican.
Algunos ejemplos:
- Las plantas y los animales los utilizan para defenderse de los virus.
- Ejemplo: Cuando las células humanas son infectadas por el virus de la hepatitis C (VHC), producen miARN que interfieren con la expresión génica por este virus de ARN y por lo tanto su capacidad para replicarse.
- Algunos herpesvirus los utilizan para mantener viva su célula hospedadora el tiempo suficiente para completar la replicación viral (embotando una respuesta inmune del huésped contra la célula infectada y previniendo su muerte prematura por apoptosis).
- De los 46 miARN expresados en el embrión de Drosophila, 25 han demostrado ser esenciales para el desarrollo normal.
- El correcto desarrollo embrionario en otros animales (p. ej., C. elegans, pez cebra, ratones) también los requiere.
- Protegen contra el peligro de mutaciones causadas por transposones que se mueven en el genoma.
- También son necesarios para regular el tamaño del charco de al menos algunos tipos de células madre.
- Los ratones transgénicos con un solo gen de miARN inactivado desarrollan inmunodeficiencia severa que afecta a las células dendríticas, las células T auxiliares y las células B.
- La expresión reducida, o nula, de ciertos miARN es característica de varios cánceres diferentes en humanos.


