8.4: Detección, identificación y cuantificación de ácidos nucleicos y proteínas específicos
- Page ID
- 53253
Si bien la electroforesis en gel puede usarse para resolver moléculas en una mezcla, por sí misma, la técnica no permite la detección e identificación de secuencias de ácidos nucleicos o proteínas específicas. Por ejemplo, el gel 2-D mostrado anteriormente separa claramente una gran cantidad de proteínas en una muestra en manchas individuales. No obstante, si quisiéramos saber si una proteína específica estaba presente, no podríamos decirlo simplemente mirando el gel. De igual manera, en un gel de agarosa, mientras que a bandas de ADN se les podría asignar un tamaño, no se pudo distinguir entre dos ADN de secuencia diferente si ambos fueran de la misma longitud en pares de bases. “Una forma de detectar la presencia de un ácido nucleico o proteína particular depende de transferir las moléculas separadas de los geles a una membrana hecha de nitrocelulosa o nylon para crear una “" transferencia "” y sondear la (s) molécula (s) de interés usando reactivos que se unen específicamente a esas moléculas.” En la siguiente sección se discutirá cómo se puede hacer esto tanto para los ácidos nucleicos como para las proteínas.
Transferencias del sur y del norte
El Southern blot lleva el nombre de su inventor, el profesor de Oxford, Edwin Southern, a quien se le ocurrió un protocolo para transferir fragmentos de ADN de un gel a una lámina de nitrocelulosa y detectar una secuencia de ADN específica entre las bandas de la mancha. Como se muestra en la Figura 8.21, el método funciona de la siguiente manera. Una mezcla de moléculas de ADN (a menudo ADN que ha sido cortado en fragmentos más pequeños usando endonucleasas de restricción) se carga en un gel de agarosa, como es habitual. Una vez completada la ejecución del gel, las bandas de ADN se transfieren del gel a una membrana. Esto se puede lograr mediante transferencia capilar, donde el gel se pone en contacto con un trozo de membrana y se tira del tampón a través del gel absorbiéndolo hacia arriba en una pila de papel absorbente colocada encima de la membrana. A medida que el tampón se mueve, lleva consigo los fragmentos de ADN. El ADN se une a la membrana dejando una “huella” de fragmentos de ADN que refleja exactamente sus posiciones en el gel. La membrana de transferencia puede tratarse con luz UV, calor o productos químicos para unir firmemente el ADN a la membrana.
A continuación, se agrega a la membrana una sonda o agente de visualización específico para la molécula de interés. En la Figura 8.21, esto se denomina sonda marcada. Las sondas en una transferencia Southern son piezas de ADN diseñadas para ser complementarias a la secuencia diana deseada. Si la secuencia de interés está presente en la transferencia, la sonda, que es complementaria a la misma, puede emparejarse (hibridarse) con ella. Luego se lava la mancha para eliminar toda la sonda no unida. Las sondas se marcan con radiactividad o con otros reactivos químicos que permiten detectarlas fácilmente cuando se unen a la transferencia, por lo que es posible determinar visualmente si la sonda se ha unido a alguna de las bandas de ADN en la transferencia. Dado que la transferencia Southern se basa en el apareamiento de bases específico entre la sonda y la secuencia diana, es fácil adaptar la técnica para detectar moléculas específicas de ARN, también. La modificación de este método para detectar ARN se denominó en broma una transferencia “northern”.
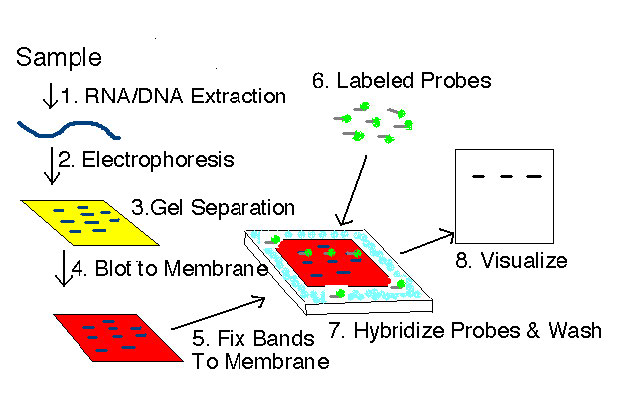
Figura 8.21 - Esquema de transferencia Northern o Southern. La transferencia Southern agrega desnaturalización de la hebra entre las etapas 4 y 5. Wikipedia
Western Blots
Las proteínas no pueden, por razones obvias, detectarse a través del emparejamiento de bases con una sonda de ADN, pero las transferencias de proteínas, hechas transfiriendo proteínas, separadas en un gel, sobre una membrana, se pueden sondear usando anticuerpos específicos contra una proteína particular de interés. La detección de proteínas suele emplear dos anticuerpos, el primero de los cuales no está marcado. El marcador está en el segundo anticuerpo, el cual está diseñado para reconocer solo el primer anticuerpo de una manera piggyback. El primer anticuerpo se une específicamente a la proteína de interés en la transferencia y el segundo anticuerpo reconoce y se une al primer anticuerpo. El segundo anticuerpo lleva comúnmente una enzima o reactivo que puede causar una reacción para producir un color tras el tratamiento posterior. Al final, si la molécula de interés está en la mezcla original, se “iluminará” y se revelará en la mancha. Esta variación sobre el tema de la transferencia se denominó Western blot (Figura 8.22).
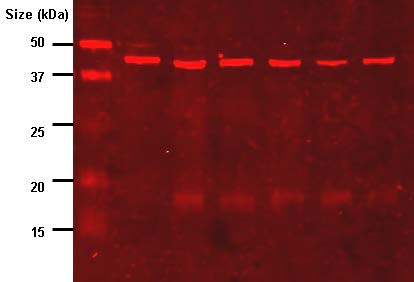
Figura 8.22 - Resultado de un análisis de transferencia Western. Wikipedia
En cada una de las transferencias descritas anteriormente, la unión de la sonda a la molécula diana permite determinar si la secuencia diana o proteína estaba en la muestra. Aunque las transferencias están diseñadas para ser utilizadas para la detección, más que para la cuantificación precisa, es posible obtener estimaciones de la abundancia de la molécula diana a partir de mediciones de densitometría de intensidad de señal.
Microarrays
Los geles 2-D son una forma de estudiar un amplio espectro de moléculas de proteína simultáneamente. Un enfoque para hacer algo similar con ADN o ARN involucra lo que se llama microarrays. Las micromatrices son especialmente útiles para monitorear las expresiones de miles de genes, simultáneamente. Donde una transferencia Northern permitiría la identificación de un solo ARNm a partir de una mezcla de ARNm, un experimento de micromatrices puede permitir la identificación simultánea de miles de ARNm que pueden ser realizados por una célula en un momento dado. También es posible realizar la cuantificación de manera mucho más confiable que con una transferencia.
Las micromatrices emplean un portaobjetos de vidrio, o chip, al que se unen secuencias cortas de ADN monocatenario, dispuestas en una cuadrícula, o matriz (Figura 8.23) Cada posición en la cuadrícula corresponde a un gen único. Es decir, la secuencia de ADN en este punto es parte de la secuencia de un gen específico. Cada punto de la cuadrícula tiene múltiples copias idénticas de la misma secuencia. Se registra la secuencia génica inmovilizada en cada posición de la cuadrícula.
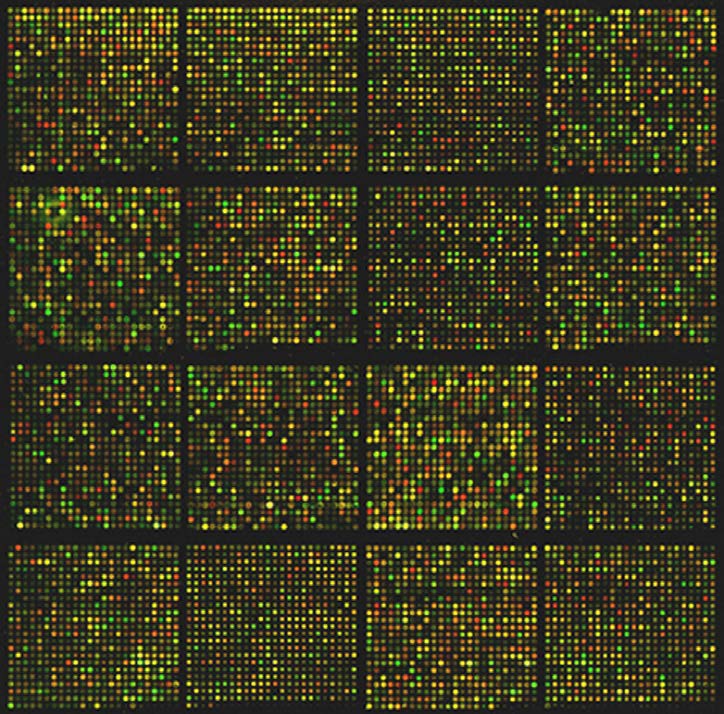
Figura 8.23 - Diseño de micromatrices. Imagen de Taralyn Tan
Al portaobjetos se le agrega una mezcla de moléculas de muestra, algunas de las cuales reconocerán y se unirán específicamente a las secuencias en el portaobjetos. La unión entre las moléculas de muestra y las secuencias unidas al portaobjetos se produce por emparejamiento de bases, en el caso de micromatrices de ADN. Luego se lava el portaobjetos para eliminar las moléculas de muestra que no están específicamente unidas a las secuencias en la cuadrícula.
Las moléculas de muestra se etiquetan con un colorante fluorescente, permitiendo identificar las manchas donde se unen. La cuadrícula se analiza punto por punto para la unión de las moléculas de muestra a las secuencias inmovilizadas. Cuantas más moléculas de muestra estén unidas en un punto, mayor será la intensidad de fluorescencia del colorante que se observará. La información de este análisis puede dar información sobre la presencia/ausencia/abundancia de moléculas en la muestra que se unen a las secuencias en la cuadrícula.
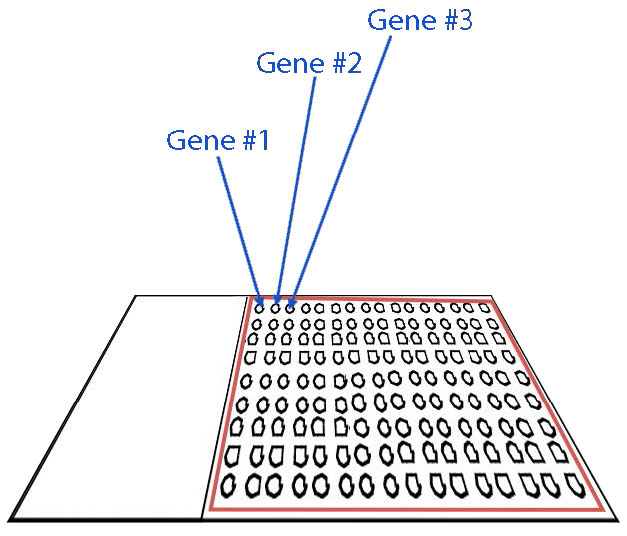
Figura 8.24 - Análisis de microarrays a gran escala del transcriptoma de ratón. Wikipedia


