4.3: Bibliotecas de presentación de fagos M13
- Page ID
- 57294
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Sistemas microbianos desarrollados para la evolución molecular experimental
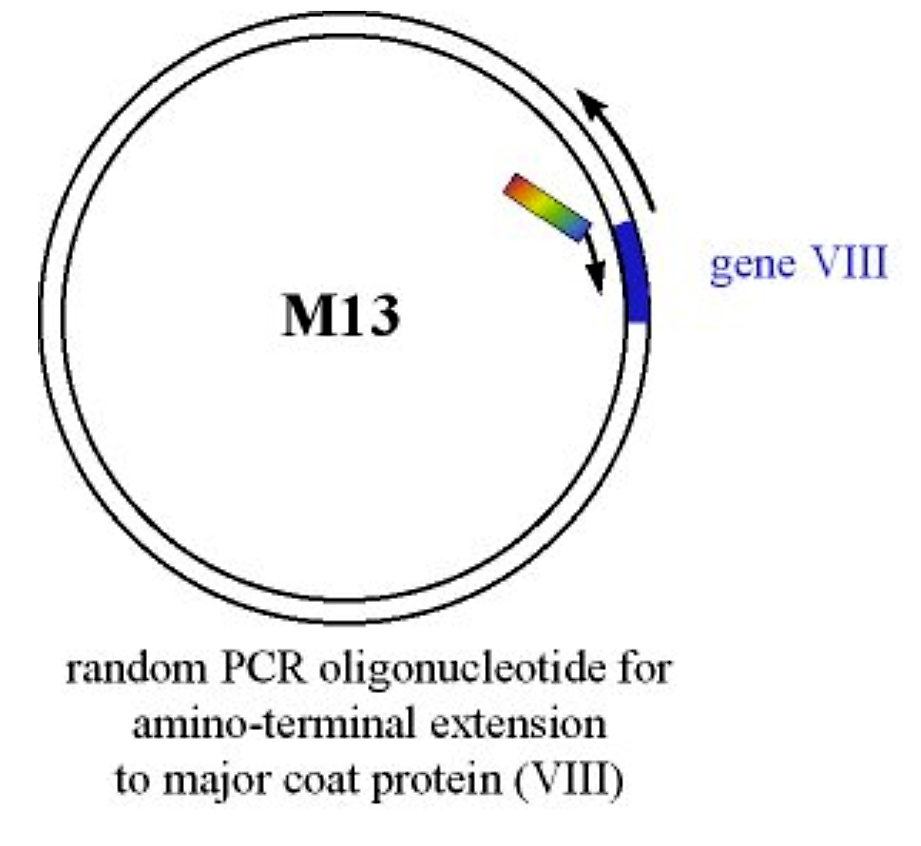
Se introduce una proteína o péptido en el extremo amino de la proteína principal de la cubierta (Gen VIII) mediante mutagénesis basada en PCR
.png)
Figura 4.3.1: Introducción de proteína en el gen VIII
El producto de PCR lineal se amplifica para regenerar un genoma viral circular intacto cuya proteína del gen VIII tiene ahora una extensión amino terminal. El M13 mutagenizado se introduce en células de E. coli. Es ADN desnudo (no empaquetado con proteínas de cubierta viral) por lo que se introduce en E. coli como un plásmido (es decir, mediante electroporación o usando células de E. coli competentes CaCl 2).
- La progenie viral extruida:
- Ser algo más largo ya que el genoma es más grande pero debe ser empaquetado o.k.
- Expresar la proteína mutante del gen VIII (cubierta mayor) como parte del ensamblaje de fagos filamentosos
.png)
Figura 4.3.2: Progenie M13 mutada
Selección
El péptido amino-terminal puede tener afinidad por un ligando específico, posiblemente un anticuerpo. Podemos usar esta afinidad de unión para purificar el fago mutante. Podemos construir una columna de cromatografía donde la matriz de cromatografía le haya unido el ligando de interés. Si pasamos nuestro fago a través de esta columna, el fago mutante que contiene la proteína mutante del gen VIII se unirá a, y se inmovilizará, en la columna
.png)
Figura 4.3.3: ligando inmovilizado
- Cualquier fago de tipo silvestre no se unirá y pasará a través de la columna
- El fago unido puede eluirse y usarse directamente para reinfectar E. coli para propagar virus con la proteína mutante del gen VIII.
La proteína mutante del gen VIII que ha sido purificada lleva consigo el ADN que codifica para sí mismo. En otras palabras, hemos seleccionado específicamente para el ADN que codifica para el gen VIII de interés (es decir, el mutante)
Cambio de la especificidad de unión
Supongamos que tenemos un ligando ligeramente diferente unido a la matriz...
.png)
Nuestro fago con proteína mutante del gen VIII no se unirá muy bien a este ligando diferente. Si introducimos los cambios de aminoácidos apropiados en nuestra proteína mutante del gen VIII, podemos mejorar la afinidad de unión (especificidad) y lograr que el fago se una al nuevo ligando
.png)
- Después de la PCR y la ligación, esto producirá una biblioteca de genomas de fagos, cada uno con una secuencia potencialmente diferente para la extensión amino terminal de la proteína del gen VIII
- En términos generales, cuando estos se introducen en E. coli, la eficiencia es típicamente tan baja que, como máximo, cada transformación exitosa da como resultado solo un genoma de fago por E. coli.
- Los fagos de la progenie de nuestra biblioteca tendrán, por lo tanto, un solo tipo de proteína del gen VIII por fago (un solo fago tendrá la proteína del gen VIII para la que codifica su ADN).
- Si pasamos la colección de fagos de progenie de nuestra biblioteca sobre nuestra columna de ligandos, nos uniremos selectivamente a aquellos fagos cuya proteína mutante del gen VIII tiene afinidad por el ligando.
.png)
Figura 4.3.5: Vinculación específica
- Si aislamos el fago unido podemos reinfectar E. coli, y aislar el fago de la progenie
- En esta primera selección podemos tener muchos otros fagos que podrían ser ligantes de ligandos débiles, o generalmente no selectivos. Entonces, el proceso se repite hasta que tenemos una biblioteca de mutantes específicos de alta afinidad (esto a veces se llama “Bio-mining” o “Bio-panning”)
- Luego podemos secuenciar el ADN y averiguar qué secuencia (s) en el mutante del gen VIII proporcionan una alta especificidad de unión a nuestro nuevo ligando
Limitaciones prácticas
- ¿Sobre qué longitud de ADN/proteína podemos aleatorizar?
Ejemplo: un tramo aleatorio de 10 aminoácidos
- 20 10 posibles secuencias polipeptídicas únicas diferentes. Cada aminoácido es de 110 g/mol o 1.8 x 10-22 g por aminoácido. Esto significaría que una biblioteca aleatoria completa de péptidos 10mer tendría una masa de 1.8 x 10-9 g.
Ejemplo: un tramo aleatorio de 20 aminoácidos
- 20 20 posibles secuencias polipeptídicas únicas diferentes. Cada aminoácido es de 110 g/mol o 1.8 x 10-22 g por aminoácido. Esto significaría que una biblioteca aleatoria completa de péptidos 20mer tendría una masa de 18.9 Kg!
Esto ni siquiera considera la masa significativamente mayor de la partícula del virus de la que formaría parte. Este tipo de cálculo demuestra que la generación de bibliotecas de secuencias de ADN aleatorias se limita a tramos muy cortos de aproximadamente media docena de posiciones de codones.
Muagénesis Aleatoria
Podríamos introducir aleatoriamente las cuatro bases en cada posición (NNN) mientras intentábamos sintetizar un codón aleatorio mutagénico, pero ¿qué nos daría esto?
| Leu, Arg, Ser (6 codones/64) | 9.4% |
| Val, Pro, Thr, Ala, Gly (4 codones/64) | 6.3% |
| Ile (3 codones/64) | 4.7% |
| Phe, Tyr, His, Gln, Asn, Glu, Asp, Cys (2 codones/64) | 3.1% |
| Met, Trp (1 codon/64) | 1.6% |
| Stop (3 codones/64) | 4.7% |
- Así, no todos los aminoácidos están presentes en cantidades iguales, y tendremos 4.7% de codones de parada
- Podemos mejorar esto usando las cuatro bases en las dos primeras posiciones, y una mezcla igual de bases G y C en la tercera posición (bamboleo). Esto daría como resultado 32 posibles codones y la siguiente distribución de aminoácidos y codones:
| Phe, Tyr, Cys, Trp, His, Gln, Ile, Asn, Met, Lys, Asp, Glu, Stop (1 codon/32) | 3.1% |
| Gly, Val, Ala, Pro, Thr (2 codones/32) | 6.3% |
| Leu, Arg, Ser (3 codones/32) | 9.4% |
Otro problema general con el método anterior es el de la unión no específica
- Esto se debe en parte a la gran cantidad de proteínas mutantes del gen VIII (~2500) en la superficie del virus.
- “La unión al ligando puede deberse a la interacción de dos o más proteínas del gen VIII que contribuyen a la unión del ligando (es decir, unión “" polivalente "”).”
- En este caso, el péptido mutante aislado puede mostrar una afinidad de unión débil o nula en absoluto.
Enfoque #1 - Mutar la proteína del gen III en lugar del gen VIII
- Si bien hay ~2,500 copias del gen VIII (proteína de la cubierta mayor) por fago, hay 5 copias del gen III (proteína de la cubierta menor) por fago
.png)
Figura 4.3.6: Mutación del gen III
- Podemos introducir la secuencia mutagénica en el extremo amino de la proteína del gen III y así limitar el número de copias en la partícula del fago
- esto todavía puede dar como resultado una unión polivalente
Enfoque #2 - Suplemento con proteína del gen III de tipo silvestre durante el ensamblaje de fagos
- Si el fago M13 contiene una segunda copia del gen III que no mutamos entonces cuando el fago se ensambla (ya que extruye a través de la membrana externa) las cinco proteínas del gen III que recoge serán una mezcla de mutante y tipo silvestre (en cantidades aproximadamente iguales). En este caso, el fago de la progenie tendrá, en promedio, 2-3 copias de la proteína mutante del gen III
- Si podemos aumentar la expresión de la proteína del gen III de tipo silvestre, hasta el punto en que esté presente en un exceso de cuatro veces sobre el mutante, entonces el fago de la progenie contendrá, en promedio, 1 copia de la proteína del gen III mutante (y la unión al ligando, por definición, será monovalente)
- Podemos poner un promotor fuerte frente al gen III de tipo silvestre
- Podríamos colocar el gen III de tipo silvestre cerca de uno de los terminadores (después del gen IV, por ejemplo). Esta posición daría como resultado una mayor expresión del gen de tipo silvestre debido a los efectos acumulativos de los promotores aguas arriba
Variación
Expresión de proteína mutagénica como fusión de una proteína de membrana externa de la propia E. coli
- Podemos unir nuestra secuencia mutagénica a un gen para una proteína de membrana externa (por ejemplo, OmpT, LamB)
- La superficie de la propia célula de E. coli muestra el péptido mutagénico
- Las proteínas de la membrana externa pueden estar presentes a un nivel de 10.000 moléculas o más
- Nuestra columna de ligandos se unirá selectivamente a células de E. coli que se unen específicamente. Estos pueden eluirse y cultivarse y el proceso repetido para seleccionar la secuencia mutante con afinidad de unión.
Expresión de proteína mutagénica como fusión con una proteína de unión a ADN
- Podemos unir nuestra secuencia mutagénica a un gen para una proteína que se une al ADN (por ejemplo, represor lac)
- El represor lac mutagénico será codificado por un plásmido que a su vez contiene dos secuencias operadoras lac.
- La molécula represora mutante expresada por el plásmido se unirá fuertemente a las regiones operadoras lac en el mismo plásmido.
- Las células se lisan (el represor permanece unido a su ADN plasmídico asociado) y se realiza la etapa de unión al ligando para enriquecer las secuencias de unión al ligando.
- El ADN plasmídico asociado se aísla y se transforma de nuevo en E. coli para amplificación y otra ronda de selección.
.png)
Figura 4.3.7: Fusión de proteína mutagénica y proteína b inding


