13.2: Midiendo la Similaridad/Disimilitud
( \newcommand{\kernel}{\mathrm{null}\,}\)
Podríamos intentar evaluar qué nodos son más similares a qué otros nodos intuitivamente mirando una gráfica. Notaríamos algunas cosas importantes. Parecería que los actores 2, 5 y 7 podrían ser estructuralmente similares en el sentido de que parecen tener vínculos recíprocos entre sí y con casi todos los demás. Los actores 6, 8 y 10 son “regularmente” similares en que están bastante aislados, pero no son estructuralmente similares porque están conectados con conjuntos de actores bastante diferentes. Pero, más allá de esto, es realmente bastante difícil evaluar rigurosamente la equivalencia con solo mirar un diagrama.
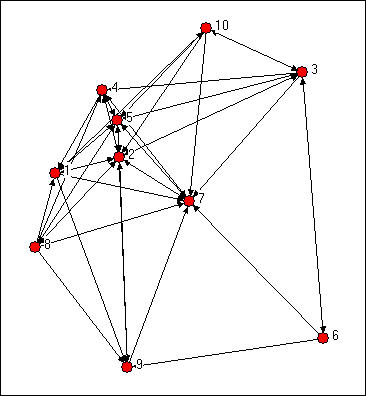
Figura 13.1: Red de información dirigida por Knoke
Podemos ser mucho más precisos al evaluar la similitud si usamos la representación matricial de la red en lugar del diagrama. Esto también nos permite usar la computadora para hacer algunos de los trabajos bastante tediosos involucrados en el cálculo de números de índice para evaluar la similitud. La matriz de datos original se ha reproducido a continuación como Figura 13.2. Muchas de las características que fueron evidentes en el diagrama también son fáciles de comprender en la matriz. Si miramos a través de las filas y contamos los grados, y si miramos hacia abajo las columnas (para contar en grados) podemos ver quiénes son los actores centrales y quiénes son los aislados. Pero, aún más generalmente, podemos ver que dos actores son estructuralmente equivalentes en la medida en que el perfil de las partituras en sus filas y columnas es similar. Encontrar equivalencia automórfica y equivalencia regular no es tan sencillo. Pero, como estas otras formas son menos restrictivas (y de ahí simplificaciones de las clases estructurales), comenzamos por medir cuán similares son los lazos de cada actor con todos los demás actores.
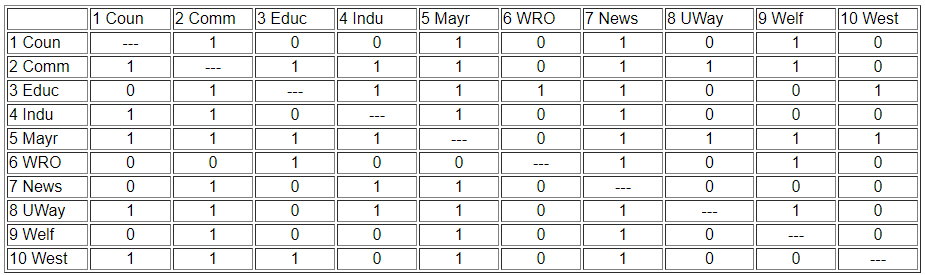
Figura 13.2: Matriz de adyacencia para la red de información Knoke
Se puede decir que dos actores son estructuralmente equivalentes si tienen los mismos patrones de vínculos con otros actores. Esto significa que las entradas en las filas y columnas para un actor son idénticas a las de otro. Si la matriz fuera simétrica, solo necesitaríamos escanear pares de filas (o columnas). Pero, dado que estos datos son sobre vínculos dirigidos, debemos examinar la similitud de envío y recepción de vínculos (claro, podríamos estar interesados en la equivalencia estructural con respecto a solo enviar, o solo recibir lazos). Podemos ver la similitud de los actores si ampliamos la matriz en la Figura 13.2 enumerando los vectores de fila seguidos de los vectores de columna para cada actor como una sola columna, como tenemos en la Figura 13.3.

Figura 13.3: Adyacencias de fila y columna concatenadas para la red de información Knoke
Los lazos de cada actor (tanto dentro como fuera) ahora se representan como una columna de datos. Ahora podemos medir la similitud de cada par de columnas para indexar la similitud de los dos actores, formando una matriz de similitudes por pares. También podríamos llegar a la misma idea a la inversa, indexando la disimilitud o “distancia” entre las puntuaciones en dos columnas cualesquiera.
Hay cualquier número de formas de indexar similitud y distancia. En las dos secciones siguientes revisaremos brevemente los enfoques más utilizados cuando los lazos se miden como valores (es decir, fuerza o costo o probabilidad) y como binarios.
El objetivo aquí es crear una matriz actor por actor de las medidas de similitud (o distancia). Una vez hecho esto, podemos aplicar otras técnicas para visualizar las similitudes en los patrones de relaciones del actor con otros actores.
Relaciones Valoradas
Un enfoque común para indexar la similitud de dos variables valoradas es el grado de asociación lineal entre las dos. Exactamente el mismo enfoque se puede aplicar a los vectores que describen las fortalezas de relación de dos actores con todos los demás actores. Al igual que con cualquier medida de asociación lineal, la linealidad es una suposición clave. A menudo es prudente, incluso cuando los datos están en el nivel de intervalo (por ejemplo, volumen de comercio de una nación a todas las demás) considerar medidas con supuestos más débiles (como medidas de asociación diseñadas para variables ordinales).
Coeficientes de correlación, covarianzas y productos cruzados de Pearson
La medida de correlación de similitud es particularmente útil cuando se “valoran” los datos sobre vínculos, es decir, nos hablan de la fuerza y dirección de la asociación, más que de simple presencia o ausencia. Las correlaciones de Pearson van desde -1.00 (es decir, que los dos actores tienen exactamente los vínculos opuestos entre sí), pasando por cero (es decir, que conocer el vínculo de un actor con un tercero no nos ayuda en absoluto a adivinar cuál podría ser el vínculo del otro actor con el tercero), hasta +1.00 (es decir, que los dos actores siempre tienen exactamente el mismo vínculo con otros actores - equivalencia perfecta). Las correlaciones de Pearson se utilizan a menudo para resumir la equivalencia estructural por pares porque la estadística (llamada “pequeña r”) es ampliamente utilizada en las estadísticas sociales. Si los datos sobre los lazos son verdaderamente nominales, o si la densidad es muy alta o muy baja, las correlaciones a veces pueden ser un poco problemáticas, y también se deben examinar las coincidencias (ver abajo). Estadísticas diferentes, sin embargo, suelen dar muy las mismas respuestas. La Figura 13.4 muestra las correlaciones de los diez perfiles de la organización Knoke de vínculos de entrada y salida de información. Estamos aplicando correlación, a pesar de que los datos de Knoke son binarios. El algoritmo UCINET Herramientas>Similitudes calculará correlaciones para filas o columnas.

Figura 13.4: Correlaciones de Pearson de filas (envío) para la red de información Knoke
Podemos ver, por ejemplo, que el nodo 1 y el nodo 9 tienen patrones idénticos de vínculos; existe una tendencia moderadamente fuerte para que el actor 6 tenga vínculos con actores que el actor 7 no tiene, y viceversa.
La medida de correlación de Pearson no presta atención a la prevalencia general de vínculos (la media de la fila o columna), y no presta atención a las diferencias entre actores en las varianzas de sus vínculos. A menudo esto es deseable, enfocarse únicamente en el patrón, más que en la media y la varianza como aspectos de similitud entre actores.
Sin embargo, a menudo, podríamos querer que nuestra medida de similitud refleje no solo el patrón de lazos, sino también las diferencias entre los actores en su densidad general de vínculos. Herramientas>Similitudes también calculará la matriz de covarianza. Si queremos incluir diferencias en varianzas entre actores como aspectos de (des) similitud, así como medias, se podría utilizar la relación entre productos calculada en Herramientas>Similitudes.
Distancias Euclideanas, Manhattan y Cuadradas
Un enfoque alternativo a la correlación lineal (y sus parientes) es medir la “distancia” o “disimilitud” entre los perfiles de empate de cada par de actores. Varias medidas de “distancia” se utilizan con bastante frecuencia en el análisis de redes, particularmente la distancia euclidiana o la distancia euclidiana cuadrada. Estas medidas no son sensibles a la linealidad de asociación y pueden ser utilizadas tanto con datos valorados como binarios.
La Figura 13.5 muestra las distancias euclidianas entre las organizaciones Knoke calculadas usando Herramientas>Disimilitudes y Distancias>Disimilaridades/distancias de vectores Std.

Figura 13.5: Distancias euclidianas en el envío para la red de información Knoke
La distancia euclidiana entre dos vectores es igual a la raíz cuadrada de la suma de las diferencias cuadradas entre ellos. Es decir, la fuerza del empate del actor A con C se resta de la fuerza del empate del actor B con C, y la diferencia es cuadrada. Esto se repite entonces a través de todos los demás actores (D, E, F, etc.), y se resume. Luego se toma la raíz cuadrada de la suma.
Una medida estrechamente relacionada es la distancia “Manhattan” o bloque entre los dos vectores. Esta distancia es simplemente la suma de la diferencia absoluta entre los lazos del actor con cada alter, sumada a través de los alters.
Relaciones Binarias
Si la información que tenemos sobre los vínculos entre nuestros actores es binaria, se pueden utilizar medidas de correlación y distancia, pero puede que no sean óptimas. Para datos que son binarios, es más común mirar los vectores de los lazos de dos actores, y ver qué tan de cerca las entradas en una “coinciden” con las entradas en la otra.
Existen varias medidas útiles de similitud de perfil de empate basadas en la idea de coincidencia que se calculan mediante Herramientas>Similitudes.
Partidos: Exact, Jaccard, Hamming
Un enfoque muy simple y a menudo efectivo para medir la similitud de dos perfiles de empate es contar el número de veces que el empate del actor A para alterar es lo mismo que el empate del actor B para alterar, y expresarlo como un porcentaje del total posible.
La figura 13.6 muestra el resultado de la relación columnas (recepción de información) de las burocracias de Knoke.
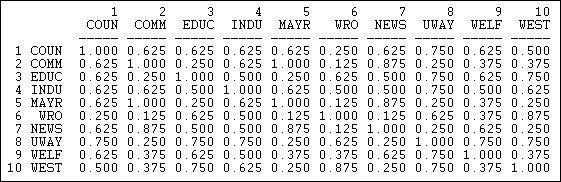
Figura 13.6: Proporción de coincidencias para recibir información de Knoke
Estos resultados muestran similitud de una manera bastante fácil de interpretar. El número 0.625 en la celda 2,1 significa que, al comparar el actor #1 y #2, tienen el mismo vínculo (presente o ausente) con otros actores62.5% de la época. La medida es particularmente útil con medidas nominales multicategoría de vínculos; también proporciona un buen escalado para datos binarios.
En algunas redes las conexiones son muy escasas. En efecto, si uno estuviera mirando lazos de conocimiento personal en organizaciones muy grandes, los datos podrían tener una densidad muy baja. Donde la densidad es muy baja, las medidas de “coincidencias”, “correlación” y “distancia” pueden mostrar relativamente poca variación entre los actores, y pueden causar dificultades para discernir conjuntos de equivalencias estructurales (por supuesto, en redes muy grandes y de baja densidad, realmente puede haber niveles muy bajos de estructuras equivalencia).
Un enfoque para resolver este problema es calcular el número de veces que ambos actores reportan un empate (o el mismo tipo de empate) con los mismos terceros actores como porcentaje del número total de vínculos reportados. Es decir, ignoramos casos en los que ni X ni Y están vinculados a Z, y preguntamos, de los lazos totales que están presentes, qué porcentaje hay en común. La Figura 13.7 muestra los coeficientes Jaccard para la recepción de información en la red Knoke, calculados mediante Herramientas>Similitudes, y seleccionando “Jaccard”.
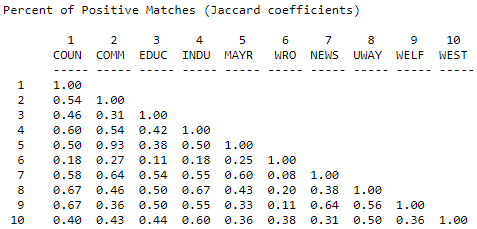
Figura 13.7: Coeficientes Jaccard para perfiles de recepción de información en la red Knoke
Nuevamente surge el mismo cuadro básico. Sin embargo, se enfatiza la singularidad del actor #6. El actor 6 es más único por esta medida debido al número relativamente pequeño de vínculos totales que tiene, esto da como resultado un menor nivel de similitud cuando se ignora la “ausencia conjunta” de vínculos. Donde los datos son escasos, y donde hay diferencias muy sustanciales en los grados de puntos, el coeficiente de coincidencia positivo es una buena opción para los datos binarios o nominales.
Otra interesante medida “coincidente” es la distancia Hamming, que se muestra en la Figura 13.8.

Figura 13.8: Distancias Hamming de recepción de información en la red Knoke
La distancia de Hamming es el número de entradas en el vector para un actor que habría que cambiar para que sea idéntico al vector del otro actor. Estas diferencias podrían ser ya sea sumar o dejar caer un empate, por lo que la distancia de Hamming trata la ausencia conjunta como similitud.
Con algo de inventiva, probablemente se pueda pensar en otras formas razonables de indexar el grado de similitud estructural entre actores. Podrías mirar el programa “Proximidades” de SPSSx, que ofrece una gran colección de medidas de similitud. La elección de una medida debe estar impulsada por una noción conceptual de “qué pasa”, la similitud de dos perfiles de lazo es lo más importante para los fines de un análisis particular. Muchas veces, francamente, hace poca diferencia, pero eso no es motivo suficiente para ignorar la cuestión.

