11.5: Uso de R para un Análisis de Cluster
( \newcommand{\kernel}{\mathrm{null}\,}\)
Para ilustrar cómo podemos usar R para completar un análisis de clúster: use este enlace y guarde el archivo allSpec.csv en su directorio de trabajo. Los datos de este archivo constan de 80 filas y 642 columnas. Cada fila es una muestra independiente que contiene uno o más de los siguientes cationes de metales de transición: Cu 2 +, Co 2 +, Cr 3 + y Ni 2 +. Las primeras siete columnas proporcionan información sobre las muestras:
- un id de muestra (en la forma custd_1 para un único estándar de Cu 2 + o nicu_mix1 para una mezcla de Ni 2 + y Cu 2 +)
- una lista de los analitos en la muestra (en la forma cuco para una muestra que contiene Cu 2 + y Co 2 +)
- el número de analitos en la muestra (un número de 1 a 4 y etiquetado como dimensiones)
- la concentración molar de Cu 2 + en la muestra
- la concentración molar de Co 2 + en la muestra
- la concentración molar de Cr 3 + en la muestra
- la concentración molar de Ni 2 + en la muestra
Las columnas restantes contienen valores de absorbancia a 635 longitudes de onda entre 380.5 nm y 899.5 nm.
Primero, necesitamos leer los datos en R, lo que hacemos usando la función read.csv ()
spec_data <- read.csv (” allSpec.csv “, check.names = FALSO)
donde la opción check.names = FALSE anula el valor predeterminado de la función para no permitir que el nombre de una columna comience con un número. A continuación, crearemos un subconjunto de este gran conjunto de datos para trabajar con
id_longitud de onda = seq (8, 642, 40)
sample_ids = c (1, 6, 11, 21:25, 38:53)
cluster_data = spec_data [sample_ids, wavelength_ids]
donde wavelength_ids es un vector que identifica las 16 longitudes de onda igualmente espaciadas, sample_ids es un vector que identifica las 24 muestras que contienen uno o más de los cationes Cu 2 +, Co 2 + y Cr 3 +, y cluster_data es una trama de datos que contiene los valores de absorbancia para estas 24 muestras en estas 16 longitudes de onda.
Antes de poder completar el análisis de conglomerados, primero debemos calcular la distancia entre los24×16=384 puntos que componen nuestros datos. Para ello, utilizamos la función dist (), que toma la forma general
dist (objeto, método)
donde objeto es un marco de datos o matriz con nuestros datos. Hay una serie de opciones para el método, pero usaremos la predeterminada, que es euclidiana.
cluster_dist = dist (cluster_data, method = “euclidean”)
cluster_dist
1 6 11 21 22 23 24 25
6 1.53328104
11 1.73128979 0.96493008
21 1.48359716 0.24997370 0.77766228
22 1.49208058 0.32863786 0.68852029 0.09664215
23 1.49457333 0.42903074 0.57495499 0.21089686 0.11755129
24 1.51211374 0.52218072 0.47457024 0.31016429 0.21830998 0.10205547
25 1.55862311 0.61154277 0.39798649 0.39406580 0.30194838 0.191251 0.09771283
38 1.17069314 0.38098750 0.96982420 0.34254297 0.38830178 0.45418483 0.53114050 0.61729900
Aquí solo se muestra una pequeña porción de los valores en cluster_dist; cada entrada muestra la distancia entre dos de las 24 muestras.
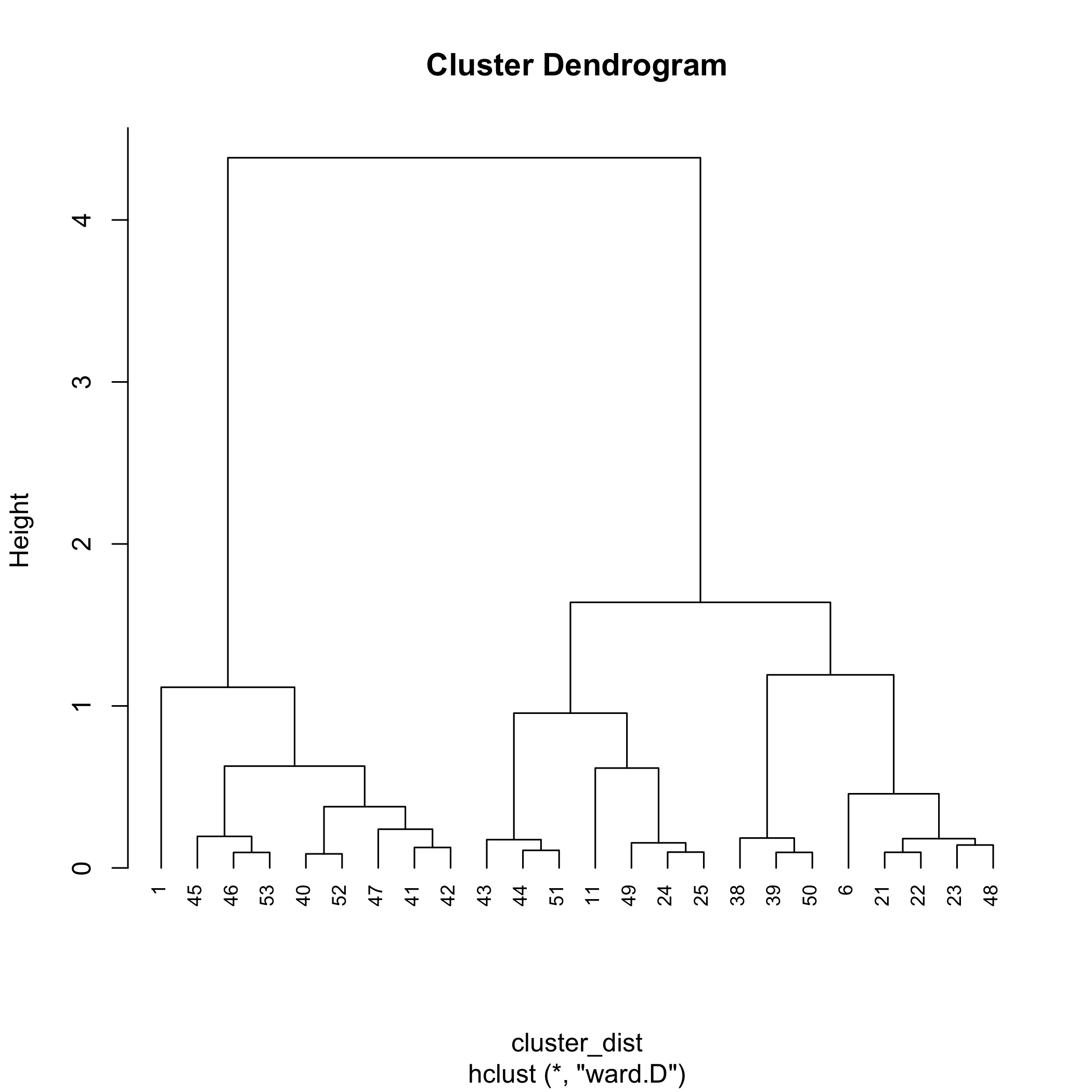
Con las distancias calculadas, podemos usar la función hclust () de R para completar el análisis de conglomerados. La forma general de la función es
hclust (objeto, método)
donde object es la salida creada usando dist () que contiene las distancias entre puntos. Hay una serie de opciones para método—aquí usamos el métodoWard.d — guardando la salida en el objeto cluster_results para que tengamos acceso a los resultados.
cluster_results = hclust (cluster_dist, método = “ward.d”)
Para ver el diagrama de clúster, pasamos el objeto cluster_results a la función plot () donde hang = -1 extiende cada línea vertical a una altura de cero. Por defecto, las etiquetas en la parte inferior del dendrograma son los identificadores de muestra; cex ajusta el tamaño de estas etiquetas.
plot (cluster_results, hang = -1, cex = 0.75)
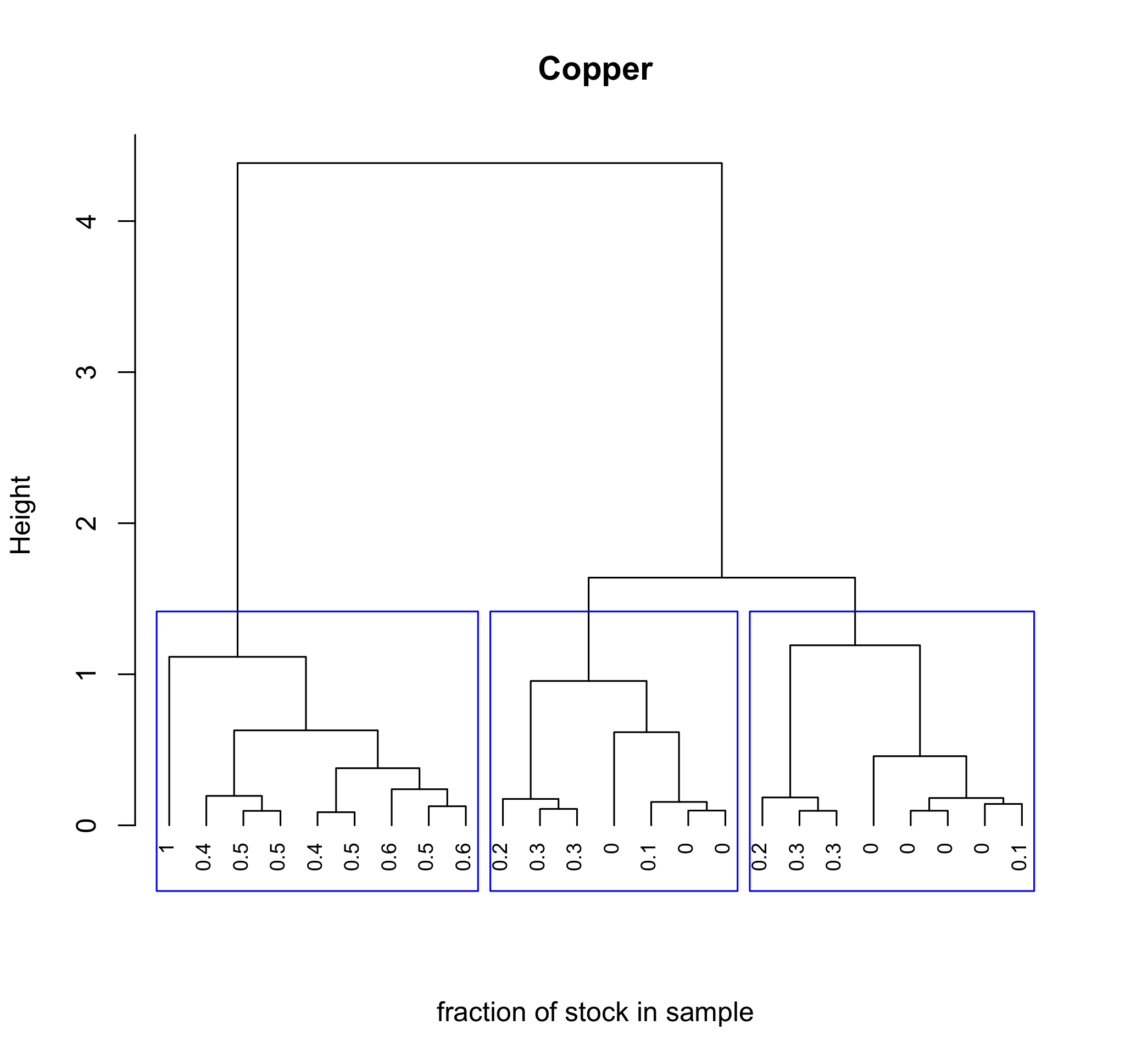
Con unas pocas líneas de código podemos agregar detalles útiles a nuestra trama. Aquí, por ejemplo, determinamos la fracción de la solución madre de Cu 2 + en cada muestra y usamos estos valores como etiquetas, y dividimos las 24 muestras en tres racimos grandes usando la función rect.clust () donde k es el número de clústeres a resaltar y que indica cuál de estos clústeres mostrar usando una caja rectangular.
cluster_copper = spec_data$conccu/spec_data$ conccu [1]
plot (cluster_results, hang = -1, labels = cluster_copper [sample_ids], main = “Cobre”, xlab = “fracción de stock en muestra”, sub = “”, cex = 0.75)
rect.hclust (cluster_results, k = 3, que = c (1,2,3), borde = “azul”)
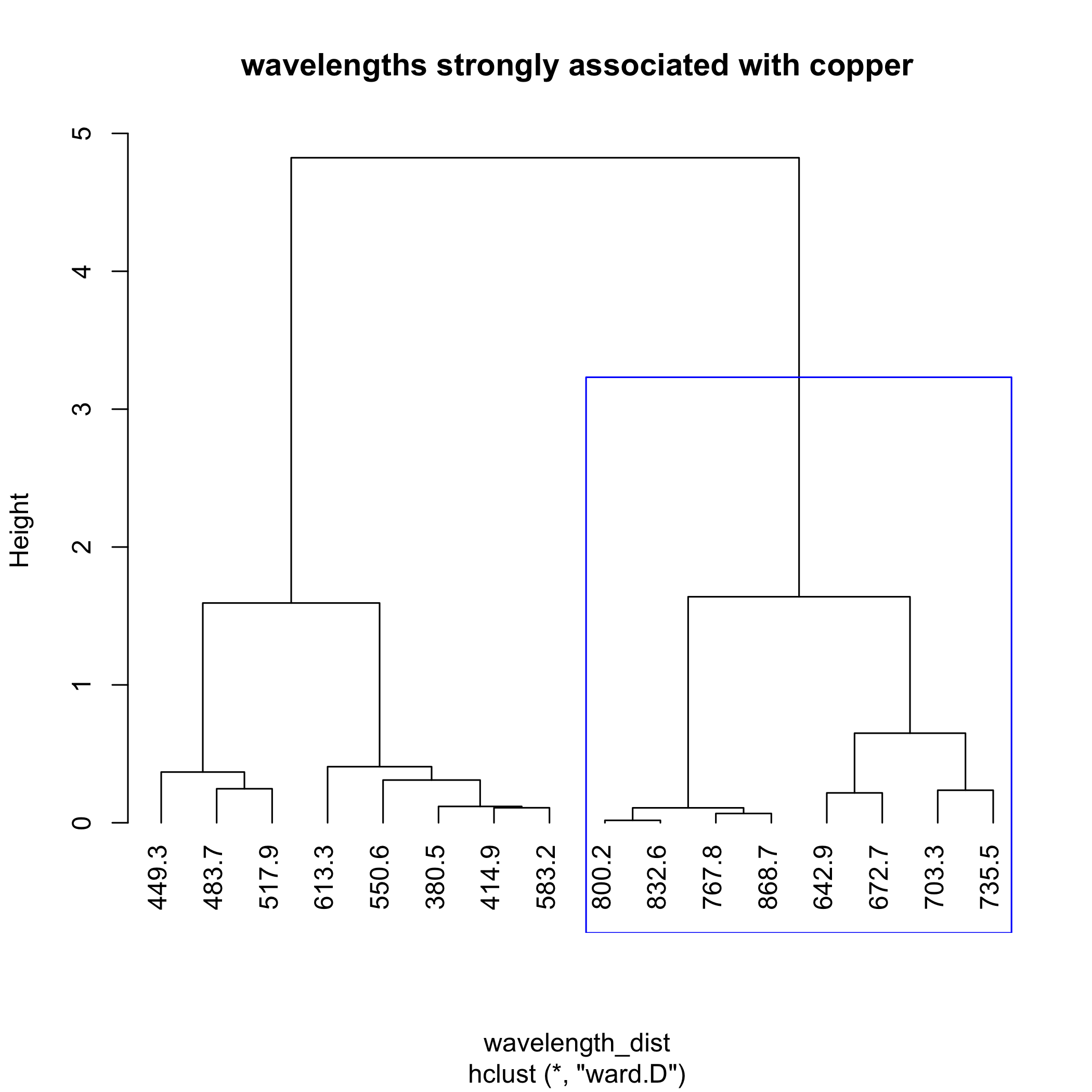
El siguiente código muestra cómo podemos usar el mismo conjunto de datos de 24 muestras y 16 longitudes de onda para completar un diagrama de clúster para las longitudes de onda. El uso de la función t () dentro de la función dist () toma la transposición de nuestros datos para que las filas sean las 16 longitudes de onda y las columnas sean las 24 muestras. Hacemos esto porque la función dist () calcula distancias usando las filas.
wavelength_dist = dist (t (cluster_data))
wavelength_clust = hclust (wavelength_dist, method = “ward.d”)
plot (wavelength_clust, hang = -1, main = “longitudes de onda fuertemente asociadas con cobre”)
rect.hclust (wavelength_clust, k = 2, which = 2, border = “blue”)
La siguiente figura destaca el cúmulo de longitudes de onda más fuertemente asociadas con la absorción por Cu 2 +.