21.3: Hibridación de ADN
- Page ID
- 69565
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Para ilustrar el uso de la termodinámica estadística para describir la unión, se discuten modelos simples para la hibridación o fusión del ADN. Estos modelos son similares a nuestra descripción de la transición hélice-bobina en su enfoque. Estas no distinguen las diferentes nucleobases, solo considerando nucleótidos a lo largo de una cadena que están emparejados (pb) o libres (f).
Consideremos el caso del emparejamiento entre oligonucleótidos autocomplementarios.
\[S+S \rightleftharpoons D \nonumber \]
S se refiere a cualquier ssADN completamente disociado y D a cualquier forma de dímero que involucre dos cadenas que tienen al menos un par de bases formado. Luego podemos seguir las expresiones para los equilibrios monómero-dímero anteriores. La constante de equilibrio para la asociación de hebras simples es
\[K_a = \dfrac{c_D}{c_S^2}\]
Esta constante de equilibrio está determinada por la barrera de energía libre dependiente de la concentración para que dos hebras se difundan en contacto y creen el primer par de bases. Si la concentración total de moléculas presentes es monómero o dímero, la forma es
\[ C_{TOT} = c_S + 2c_D \]
entonces la fracción de las cadenas de ADN en la forma dímera es
\[ \theta_D = \dfrac{2c_D}{C_{tot}} \]
y eq. (10) lleva a
\[ \theta_D = 1+(4K_aC_{tot})^{-1}-\sqrt{(1+(4K_aC_{tot})^{-1})^2-1} \]
Vemos que a la concentración total, que resulta en una fracción dímera\(\theta_D = 0.5\), se obtiene de la constante de asociación\(K_a=(9C_{tot})^{-1} \). Esta es una descripción tradicional de la termodinámica de un equilibrio monómero-dímero.
Podemos calcular K a partir de las funciones de partición molecular para los estados S y D:
\[K_a = \dfrac{q_D}{q_S^2} \nonumber \]
Diferentes modelos de hibridación variarán en la forma de estas funciones de partición. Para cualquier estado, podemos separar la función de partición en contribuciones de los grados conformacionales de libertad relevantes para el emparejamiento de bases y la hibridación, y otros grados de libertad, qi = qi, confqi, ext. Suponiendo que los grados externos de libertad serán en gran parte de naturaleza entrópica, descuidamos un cálculo explícito y factorizamos los grados externos de libertad definiendo la variable γ:
\[\gamma = \dfrac{q_{D,ext}C_{tot}}{q_{S,ext}^2} \]
entonces
\[ \theta_D = 1+\dfrac{q_{S,int}^2}{4\gamma q_{D,int}}-\sqrt{\left( 1+ \dfrac{q_{S,int}^2}{4\gamma q_{D,int}} \right)^2-1} \]
Oligonucleótidos cortos: El modelo de cremallera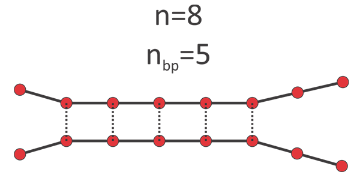
Para la hibridación de oligonucleótidos cortos, una aproximación común (y razonable) es el modelo de estiramiento único, que asumió que el apareamiento de bases solo ocurrirá como un solo tramo continuo de pares de bases. Esto es razonable para oligómeros cortos (n < 20) donde dos tramos helicoidales distintos separados por una burbuja (bucle) son improbables dada la longitud de persistencia del ADNds. El modelo de cremallera se refiere a la caja de estiramiento único con “coincidencia perfecta”, en la que solo se cuenta el emparejamiento entre las bases en hebras de ADN alineadas con precisión de secuencia. Como resultado de estas dos aproximaciones, los únicos pares de bases disociados observados en este modelo aparecen al final de una cadena (deshilachado).
El número de bases en una sola cadena es n y el número de bases que se emparejan es n pb. Para el dímero, consideramos todas las configuraciones que tengan al menos un par de bases formado. La función de partición de dímero se puede escribir como
\[ \begin{aligned} q_{D,int}(n) &=\sigma \sum_{n_{bp}=1}^ng(n,n_{bp})s^{n_{bp}} \\ &=\sigma \sum_{n_{bp}=1}^n (n-n_{bp} +1)s^{n_{bp}} \end{aligned} \]
Aquí g es el número de formas de disponer nbp pares de bases continuas en una hebra con longitud n; σes el peso estadístico para nuclear el primer par de bases; y s es el peso estadístico para formar un par de bases junto a un segmento ya emparejado:\( s=e^{-\Delta \varepsilon_{bp}/k_BT}\). Por lo tanto, en el modelo de cremallera, la constante de equilibrio en la ecuación (23) entre ADNss y dímeros que involucran al menos un par de bases intacto es: K zip = σs. En el caso de cadenas de polinucleótidos homogéneas, en las que se permite el deslizamiento de registro entre cadenas:\( q_{D,int}(n) =\sigma \sum_{n_{bp}=1}^n (n-n_{bp} +1)^2s^{n_{bp}} \). La suma en eq. (27) se puede evaluar exactamente, dando
\[q_{D,int}(n) = \dfrac{\sigma_S}{(s-1)^2}\left[ s^{n+1} -(n+1)s+n \right] \]
En el caso de que s > 1 (\( \Delta \varepsilon_{bp} < 0 \)) y n1, q D, int →σs n. Además, la distribución de probabilidad de los segmentos helicoidales es
\[ P_{bp}(n,n_{bp}) = \dfrac{(n-n_{bp}+1)\sigma s^{n_{bp}}}{q_{D,int}}\qquad 1\leq n_{bp} \leq n \]
La siguiente gráfica muestra ilustraciones de la densidad de probabilidad y el paisaje energético asociado para un rango estrecho de s a través de la transición hélice-bobina. Estas figuras ilustran un estado dúplex que siempre tiene un único mínimo de energía libre caracterizado por configuraciones deshilachadas.

Además de la fracción de moléculas que se asocian para formar un dímero, también debemos considerar la fracción de contactos que forman exitosamente un par de bases en el estado dímero
\[ \theta_{bp} = \dfrac{ \langle n_{bp} \rangle }{n} \nonumber \]
Podemos evaluar esto usando la identidad
\[ \langle n_H \rangle = \dfrac{s}{q} \dfrac{\partial q}{\partial s} \nonumber \]
Usando eq. (28) tenemos
\[ \theta_{bp} = \dfrac{ns^{n+2}-(n+2)s^{n+1}+(n+2)s - n}{n(s-1)(s^{n+1}-s(n+1)+n)} \nonumber \]
Similar a la transición hélice-espiral en polipéptidos, θ bp muestra un comportamiento cooperativo con una transición centrada en s = 1, que se vuelve más pronunciada con n creciente y decreciente σ.

Finalmente, podemos escribir la fracción total de nucleobases que participan en un par de bases como el producto de la fracción de las cadenas de ADN que están asociadas en forma de dímero, y la fracción promedio de bases del dímero que se emparejan.
\[\theta_{tot} = \theta_D \theta_{bp} \nonumber \]

