17.2: Fosfato de Piridoxal (Vitamina B6)
- Page ID
- 72302
La coenzima piridoxal fosfato (comúnmente abreviado PLP) es la forma activa de la vitamina\(B_6\), o piridoxina.
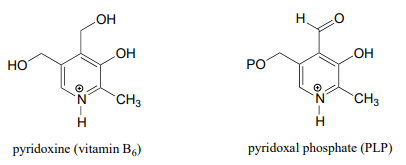
El PLP es necesario para más de 100 reacciones diferentes en el metabolismo humano, principalmente en las diversas vías de biosíntesis y degradación de aminoácidos. La función esencial del PLP es actuar como un 'sumidero electrónico', estabilizando una carga formal negativa que se desarrolla en intermedios clave de reacción. Algunas de las reacciones te serán familiares a partir del capítulo 12 y 13 veremos ejemplos, por ejemplo, de -dependientes\(\alpha \)-carbon racemization, as well as aldol- and Claisen-type reactions. Other reactions will be less familiar: for example, the participation of allows for decarboxylation of amino acids, a chemical step which would be highly unlikely without the coenzyme, and PLP is also required for a very important class of biochemical transformation called 'transamination', in which the amino group of an amino acid is transferred to an acceptor molecule. Before we dive into the reactions themselves, though, we need to begin by looking at a key preliminary step that is common to all of the PLP reactions we will see in this section.
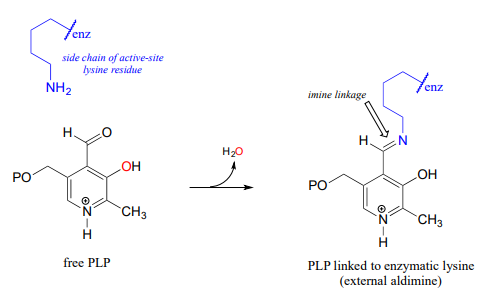
PLP in the active site: the imine linkage
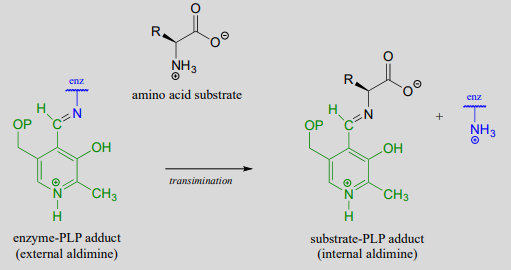
The common catalytic cycle of a PLP-dependent enzyme begins and ends with the coenzyme covalently linked to the enzyme's active site through an imine linkage between the aldehyde carbon of PLP and the amine group of a lysine residue (see section 10.5 to review the mechanism for imine formation). For a PLP-dependent enzyme to become active, a PLP molecule must first enter the active site of an enzyme and form an imine link to the lysine. This state is often referred to as an external aldimine.
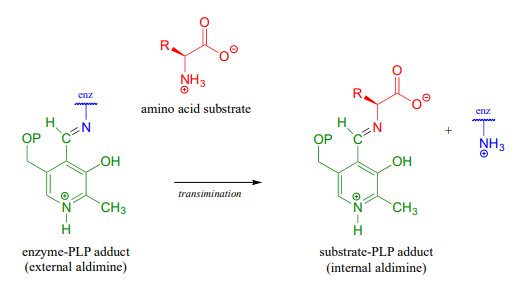
El primer paso de prácticamente todas las reacciones dependientes de PLP es la transiminación (sección 10.5), ya que el grupo amino en el sustrato aminoácido desplaza al grupo amino de la lisina enzimática. Este estado -donde la coenzima está unida covalentemente al sustrato o producto de la reacción- suele denominarse aldimina interna.
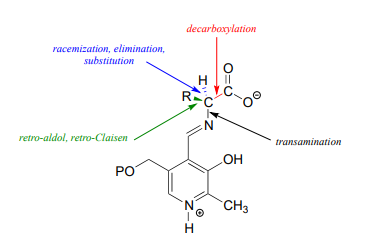
Con la transiminación preliminar lograda, la química PLP real está lista para comenzar. La versatilidad del PLP en términos de su capacidad para ayudar con una amplia variedad de tipos de reacción se ilustra en la siguiente figura, mostrando cómo, dependiendo de la reacción/enzima en cuestión, PLP puede ayudar en la escisión de cualquiera de los cuatro enlaces al\(\alpha \)-carbon of the amino acid substrate.
Veamos primero la reacción catalizada por alanina racemasa dependiente de PLP. (EC 5.1.1.1).
Racemización de aminoácidos dependiente de PLP
En la sección 12.2 vimos un ejemplo de una reacción de racemización de aminoácidos independiente de PLP, en la que el intermedio cargado negativamente era simplemente la forma enolada de un carboxilato:

Muchas otras reacciones de aminoácidos racemasas, sin embargo, requieren la participación de PLP.
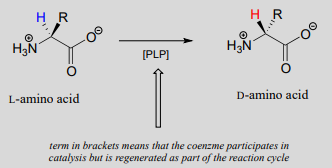
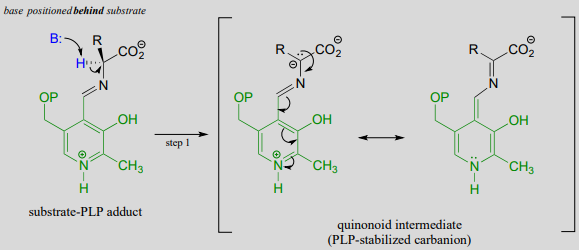
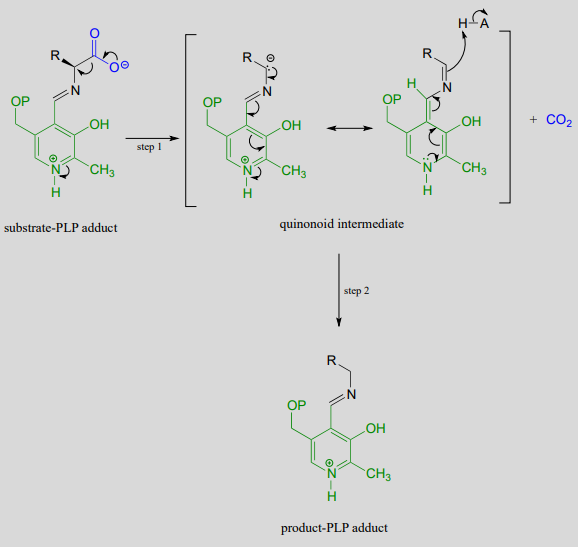
Al igual que todas las demás reacciones dependientes de PLP que veremos en esta sección, la racemización de aminoácidos dependiente de PLP comienza con una etapa preliminar en la que el sustrato se une a la coenzima a través de una transiminación. Una vez que se une a PLP en el sitio activo, el protón a de un sustrato de aminoácido es abstraído por una base de sitio activo (paso 1 a continuación). La carga negativa en el intermedio carbanión puede, por supuesto, deslocalizarse en el grupo carboxilato. La coenzima PLP, sin embargo, proporciona una red expandida de conjugados\(\pi \)-bonds over which the electron density can be delocalized all the way down to the PLP nitrogen. This is what we mean when we say that the job of PLP is to act as an ‘electron sink’: the coenzyme is very efficient at absorbing, or delocalizing, the excess electron density on the deprotonated \(\alpha \)-carbon of the reaction intermediate. PLP is helping the enzyme to increase the acidity of the \(\alpha \)-hydrogen by stabilizing the conjugate base. A PLP-stabilized carbanion intermediate is commonly referred to as a quinonoid intermediate. Note that in the overall reaction equation below, PLP appears below the reaction arrow in brackets, indicating that it participates in the mechanism but is regenerated as part of the reaction cycle.
PLP-dependent amino acid racemization:
Mechanism:
Preliminary step - transimination
First step - deprotonation:
Second step - reprotonation from the opposite side:
Final step - transimination:
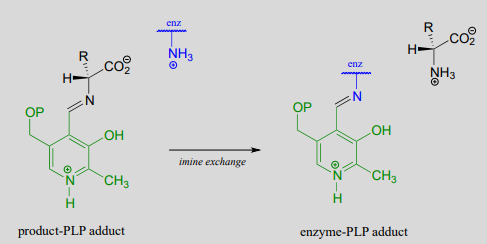
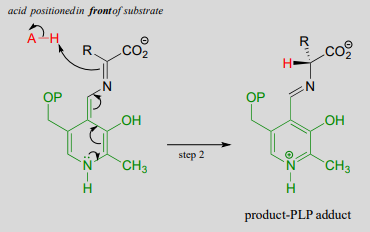
Just as in the PLP-independent racemase reactions, reprotonation occurs on the opposite side of the substrate (step 2), leading to the \(D\)-amino acid product.
All that remains is the final imine exchange which frees the \(D\)-amino acid product and re-attaches the coenzyme to the enzymatic lysine side-chain, ready to begin another catalytic cycle.
To simplify matters, from here on we will not include the preliminary and final transimination steps in our PLP reaction figures - we will only show mechanistic steps that occur while the substrate is attached to the coenzyme (the internal aldimine forms).
PLP-dependent decarboxylation
In the amino acid racemase reaction above, PLP assisted in breaking the \(\alpha \)-carbon to \(\alpha \)-proton bond of the amino acid. Other PLP-dependent enzymes can catalyze the breaking of the bond between the \(\alpha \)-carbon and the carboxylate carbon by stabilizing the resulting carbanion intermediate: these are simply decarboxylation reactions.
PLP-depended amino acid decarboxylation:

Mechanism:
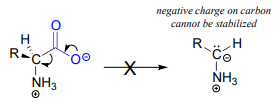
Notice something very important here: while in racemization reactions the assistance of PLP can be seen as 'optional' (in the sense that some racemase enzyme use PLP and others do not), the coenzyme is essential for amino acid decarboxylation steps. Without PLP, there is no way to stabilize the carbanion intermediate, and decarboxylation is not a chemically reasonable step.
Un ejemplo de una reacción de descarboxilación facilitada por PLP es el paso final en la ruta de biosíntesis de lisina: (EC 4.1.1.20).

Dibuje flechas mecanicistas para la etapa de ruptura de enlaces carbono-carbono de la reacción de descarboxilación dependiente de PLP anterior.
Escisión retroaldólica y retroclaisen dependiente de PLP
(Sería una buena idea antes de leer esta sección revisar los mecanismos de reacción aldol/retro-aldol y claisen/retro-claisen en las secciones 12.3 y 13.3, respectivamente)
Hasta el momento hemos visto al PLP jugar un papel en romper el vínculo entre los\(\alpha \)-carbon and its \(\alpha \)-proton (in the racemization reaction), and the bond between the \(\alpha \)-carbon and carboxylate carbon (in the decarboxylation reaction).

Otras enzimas dependientes de PLP catalizan la escisión del enlace entre el\(\alpha\) carbono y el primer carbono en la cadena lateral del aminoácido, también conocido como el\(\beta \) carbono. En la vía de degradación de la serina, la serina se convierte primero en glicina mediante una reacción de escisión retro-aldólica. (). Aunque se podría proponer un mecanismo razonable sin la participación de PLP, esta reacción de hecho requiere que la coenzima ayude en la estabilización de la carga negativa en el intermedio carbanión.
Una reacción de escisión retro-aldólica dependiente de PLP
(serina hidroximetil transferasa, EC 2.1.2.1)
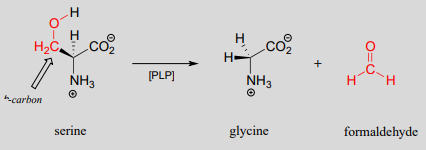
Mecanismo:
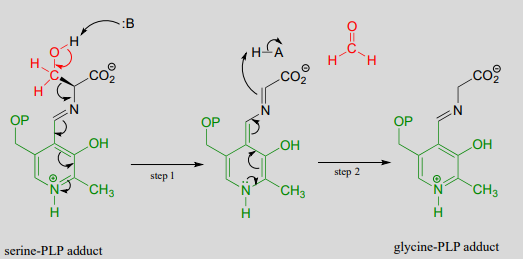
Obsérvese que, en esta reacción al igual que en la reacción racemasa descrita anteriormente, el intermedio clave es un carbanión o quinonoide estabilizado con PLP.
¿Qué pasa con el (¡tóxico!) formaldehído producido en esta reacción? Veremos más adelante en este capítulo cómo la enzima serina hidroximetiltransferasa pasa a usar otra coenzima llamada tetrahidrofolato para evitar que el formaldehído salga del sitio activo y cause daño a la célula.
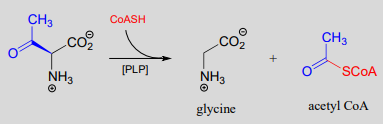
El PLP también ayuda en las reacciones de escisión de retro-claisen (sección 13.3), como esta etapa en la degradación de la treonina. (EC 2.3.1.29)
Una reacción retro-claisen dependiente de PLP:
Mecanismo:
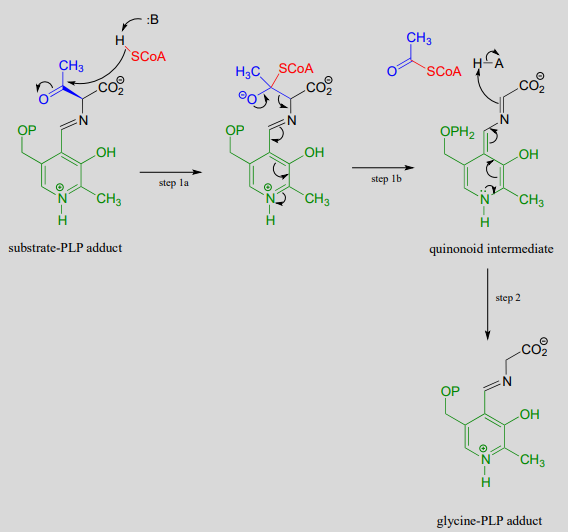
Observe cómo, al igual que la reacción retro-aldólica, se rompe el enlace entre el\(\alpha \)\(\beta \) -carbono y el -carbono del sustrato aminoácido (en la etapa 1b).
Transaminación dependiente de PLP
Uno de los tipos de reacción más importantes en el metabolismo de los aminoácidos es la transaminación, en la que un grupo amino de una molécula donante (a menudo un aminoácido) se transfiere a una molécula aceptora de cetona o aldehído.
Una reacción de transaminación:

Transaminación fase 1 (transferencia del grupo amino del sustrato de aminoácido a la coenzima)
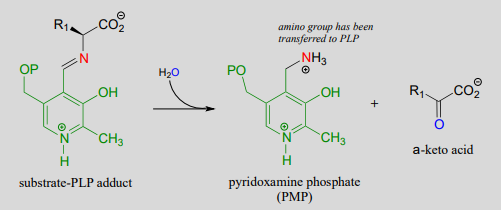
Mecanismo:
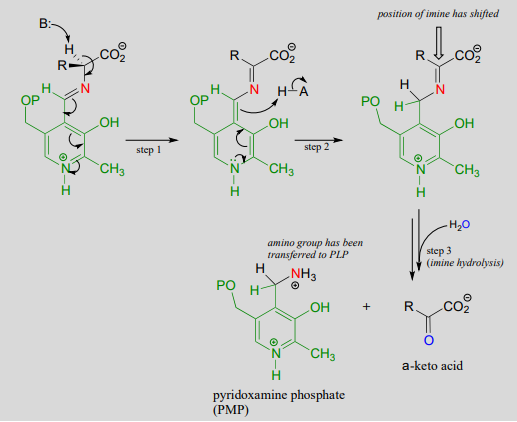
Una vez más, el paso 1 es la abstracción del\(\alpha \) protón -protón del aducto PLP-sustrato. Sin embargo, en una reacción de transaminasa esta etapa inicial de desprotonación va seguida inmediatamente de reprotonación en lo que originalmente era el carbono aldehído de PLP (paso 2 anterior), lo que da como resultado un nuevo doble enlace carbono-nitrógeno (es decir, una imina) entre el átomo\(\alpha \) -carbono y el átomo de nitrógeno del aminoácido original. Luego se hidroliza el grupo imina reposicionado (paso 3 anterior), rompiendo el enlace carbono-nitrógeno, transfiriendo el grupo amino a la coenzima y liberando un\(\alpha \) -ceto ácido.
La coenzima, que ahora porta un grupo amina y se llama piridoxamina fosfato (\(PMP\)), luego transfiere el grupo amina a\(\alpha \) -cetoglutarato (para formar glutamato) a través de una inversión de todo el proceso descrito anteriormente.
Reacción de transaminación, fase 2
(transferencia del grupo amino de la coenzima a la molécula aceptora)
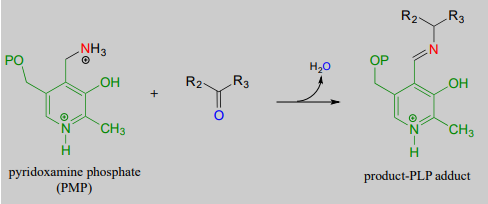
Mecanismo:
ver ejercicio a continuación
En una reacción de transaminación, la coenzima PLP no solo proporciona un sumidero de electrones, también sirve como un 'lugar de estacionamiento' temporal para un grupo amino ya que se transfiere de donante a aceptor.
Mostrar un mecanismo completo paso a paso para la 'fase 2' de la reacción de transaminación anterior.
Aquí hay un ejemplo de una reacción de transaminación en la ruta de biosíntesis de arginina: EC 2.6.1.11

- Dibuje flechas para el primer paso mecanicista de la 'fase 2' de la reacción de transaminasa anterior.
- Qué carbono en el lado del sustrato de la reacción eventualmente se convertirá en el\(\alpha \)-carbon of arginine?
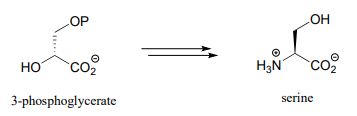
Propose a pathway, with three enzymatic steps, for the biosynthesis of serine from 3-phosphoglycerate. Include a generalized ('-ase') enzyme name for each step. Glutamate plays a role in the process as an amino group donor.
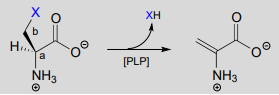
PLP-dependent \(beta \)-elimination and \(\beta \)-substitution
(Before starting this section, it would be a good idea to review \(E1cb\) \(\beta \)-elimination and conjugate addition reaction mechanisms in section 13.4)
By now it should be pretty apparent that PLP is a pretty versatile coenzyme! Two more reaction types in the PLP toolbox are \(\beta \)-elimination and \(\beta \)-substitution on amino acid substrates.
In a PLP-dependent \(\beta \)-elimination reaction, the coenzyme simply helps to stabilize the carbanion intermediate of the \(E1cb\) mechanism:
PLP-dependent \(\beta \)-elimination reaction
Mechanism:
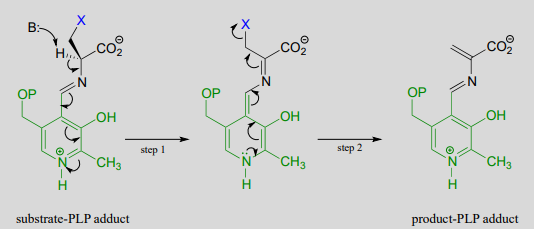
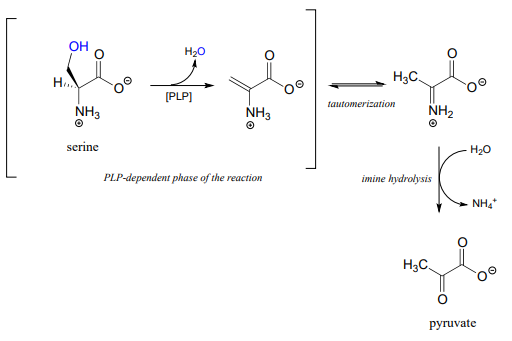
Serine dehydratase (EC 4.2.1.13) catalyzes a PLP-dependent \(\beta \)-elimination in the first step of the serine degradation pathway:
Una reacción\(\beta \) de sustitución es simplemente\(E1cb\) eliminación seguida directamente por la reacción inversa (adición de conjugado) con un nucleófilo diferente (Y en la figura siguiente):
Una reacción de\(\beta \) sustitución:
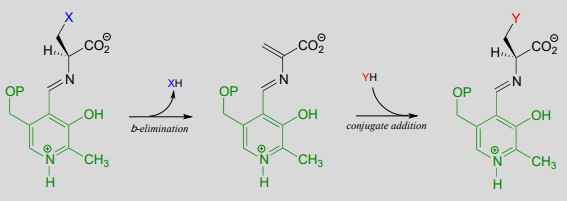
En muchas bacterias, la síntesis de cisteína a partir de serina incluye una etapa de\(\beta \) sustitución dependiente de PLP (EC 2.5.1.47).
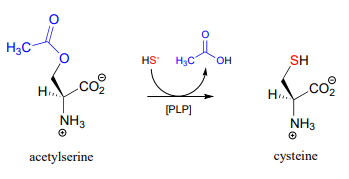
Dibujar un mecanismo para la fase de adición de conjugado de la reacción anterior (terminar con el aducto cisteína-PLP).
Dependiente de PLP\(\gamma \)-elimination and \(\gamma \)-substitution reactions
The electron sink capability of PLP allows some enzymes to catalyze eliminations at the \(\gamma \)-carbon of some amino acid side chains, rather than at the \(\beta \)-carbon. secret to understanding the mechanism of a \(\gamma \)-elimination is that PLP essentially acts as an electron sink twice - it absorbs the excess electron density from not one but two proton abstractions.
\(\gamma \)-elimination

Mechanism:

In a familiar first step, the \(\alpha \)-proton of the amino acid is abstracted by an enzymatic base, and the electron density is absorbed by PLP. Next comes the new part - before anything happens to the electron density from the first proton abstraction, a second proton, this time from the \(\beta \)-carbon on the side chain, is abstracted, forming an enamine intermediate (step 2). The phenolic proton on the pyridoxal ring of PLP donates a proton to the nitrogen. step 3, the leaving is expelled and a new \(\pi \)-bond forms between the \(\beta \) and \(\gamma \) carbons (step 3). This \(\pi \)-bond is short-lived, however, as the electron density from the first proton abstraction, which has been 'stored' in PLP all this time, flows back up to protonate the \(\alpha \)-carbon (step 4), leaving the \(\gamma \)-elimination product linked to PLP via the usual imine connection.
An example is the cystathionine \(\gamma \)-lyase reaction in the methionine degradation pathway (EC 4.4.1.1):
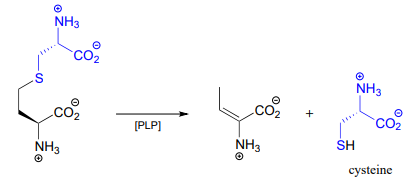
higo 2
Una reacción relacionada es la\(\gamma \) sustitución dependiente de PLP, que de nuevo es simplemente\(\gamma \) -eliminación de un grupo saliente (X en la figura siguiente) seguida directamente por el proceso inverso (a\(\gamma \) -adición) con un nucleófilo diferente ('Nu' en la figura siguiente).
\(\gamma \)Sustitución dependiente de PLP:

Mecanismo:

A continuación se muestra una reacción de\(\gamma \) sustitución dependiente de PLP en la vía de degradación de metionina (EC 4.2.1.22):

Racemasa a aldolasa: alteración del curso de una reacción PLP
Hemos visto cómo las enzimas dependientes de PLP catalizan una variedad de tipos de reacción -racemización, escisión retroaldol/retro-claisen, transaminación, eliminación y sustitución- que, a pesar de su aparente diversidad, se caracterizan por la formación de un intermedio carbanión crítico que se estabiliza mediante la propiedad de “sumidero de electrones” de la coenzima PLP. Dada esta característica mecanicista común, sería razonable proponer que la arquitectura del sitio activo de estas enzimas también podría ser bastante cercana. Esta idea fue muy bien ilustrada por un experimento en el que los investigadores encontraron que cambiar un solo aminoácido de sitio activo de alanina racemasa dependiente de PLP era suficiente para convertirlo en una retro-aldolasa (J. Am. Chem. Soc. 2003, 125, 10158).
En la reacción de alanina racemasa de tipo silvestre (natural), una histidina de sitio activo (roja en la figura a continuación) desprotonata un residuo de tirosina vecino (azul), que a su vez actúa como la base catalítica abstrayendo el\(\alpha \)-proton of the substrate. When researchers changed the tyrosine to an alanine (using a technique called 'site-directed mutagenesis'), and substituted \(\beta \)-hydroxytyrosine for the alanine substrate, the new 'mutant' enzyme catalyzed a retro-aldol reaction.
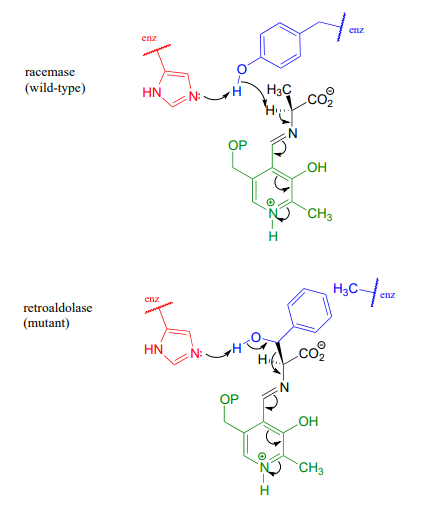
Observe lo que ha sucedido aquí: la histidina básica, sin tirosina para desprotonar por la mutación, se posiciona para abstraer un protón del grupo\(\beta \) -hidroxilo del nuevo sustrato, estableciendo una escisión retroaldólica. Eso fue todo lo que se necesitó para cambiar una racemasa en una retroaldolasa, porque el necesario sistema de sumidero de electrones PLP quedó en su lugar. Los investigadores predijeron correctamente que el anillo de fenilo de\(\beta \) -hidroxi tirosina encajaría muy bien en el espacio dejado vacío debido al cambio de tirosina a alanina en la estructura de la enzima mutante
Estos resultados subrayan la estrecha relación mecanicista entre dos reacciones dependientes de PLP que, a primera vista, parecen ser bastante diferentes, y sugieren que las racemasas y aldolasas dependientes de PLP pueden haber evolucionado a partir de una enzima “ancestro” común.
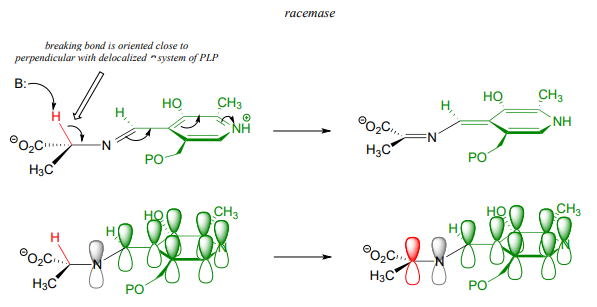
Consideraciones estereoelectrónicas de las reacciones dependientes de PLP
Recordemos que todas las reacciones dependientes de PLP implican la escisión de uno de los enlaces provenientes del\(\alpha \) -carbono de un sustrato de aminoácido, sirviendo la coenzima como un 'sumidero electrónico' para estabilizar el intermedio que resulta. Las enzimas dependientes de PLP aceleran esta etapa de ruptura de enlaces al unir el aducto Sustrato-PLP en una conformación tal que el enlace que se rompe está cerca de perpendicular al plano formado por el\(\pi \) sistema conjugado de PLP: de esta manera, como el\(\alpha \) -carbono transita de\(sp^3\) a\(sp^2\) hibridación, el\(\pi \) orbital no hibridado ya está orientado para superponerse con el resto del sistema conjugado. Por ejemplo, en alanina racemasa el primer paso es la escisión del\(C\alpha -H\) enlace, por lo que debe ser ese enlace el que se posicione casi perpendicular al plano PLP:
Asimismo, en una aminoácido descarboxilasa, el\(C\alpha \)-carboxylate bond is held near-perpendicular to the PLP plane, and in hydroxymethyltransferase, the \(C\alpha -C\beta \) bond is in the perpendicular orientation:
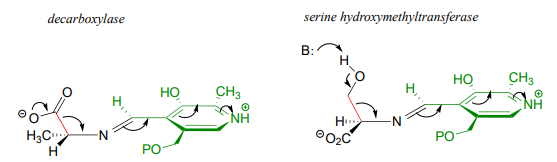
These are all good examples of how enzymatic catalysis is achieved, in part, by the ability of the active site to bind the substrate molecule in a specific conformation which contributes to the lowering of the activation energy of a key reaction step.


