7.11: Espectros infrarrojos como evidencia de estructura derivada de ácido carboxílico
( \newcommand{\kernel}{\mathrm{null}\,}\)
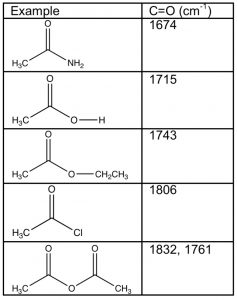
Los ácidos carboxílicos y sus derivados tienen una serie de características en común, siendo la más obvia que el carbono carbonilo tiene tres enlaces a elementos electronegativos, por lo tanto, el carbono carbonilo se encuentra en un estado de oxidación más alto que los aldehídos y cetonas. Sin embargo, la naturaleza del átomo electronegativo unido al carbonilo sí impacta las propiedades de la molécula como un todo. Por ejemplo, si examinamos las absorciones de carbonilo de estos derivados, encontramos un rango bastante amplio de frecuencias. Recordemos que la mayoría de los aldehídos y cetonas absorben alrededor1710−30cm−1, similar al valor que muestran los ácidos carboxílicos. Los derivados de ácidos carboxílicos, sin embargo, varían de alrededor1670cm−1 para amidas a 1810-30 cm —1 para cloruros y anhidridos de ácido. ¿Cómo podemos explicar la diferencia y utilizarla para predecir (y explicar) las diferencias en las propiedades de estos derivados?
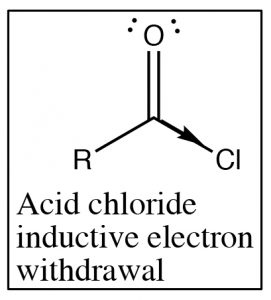
Recordemos que la frecuencia de absorción IR de los carbonilos depende de la energía requerida para estirar el enlace (que está determinada por la energía del enlace). Dado que la frecuencia de absorción de amida es mucho menor que la frecuencia de absorción del cloruro de ácido podemos concluir que el grupo carbonilo en la amida requiere menos energía para estirarse que el cloruro de ácido (el enlace es más débil). Si observamos las estructuras de estos dos grupos funcionales vemos que ambos involucran un elemento electronegativo unido al carbono carbonilo, lo que lleva a la retirada de electrones por inducción a través del enlace sigma al carbono. La diferencia entre los dos (amida y cloruro de ácido) surge porque el nitrógeno de la amida también puede donar su par de electrones al oxígeno carbonílico. El resultado es que en las amidas existe un solapamiento significativo entre el par solitario del nitrógeno amida y el sistema de enlaces carbonilo pi. ElC−O enlace ahora tiene menos carácter de doble enlace y, por lo tanto, se necesita menos energía para estirarse. En contraste, el cloro (o cualquier halógeno) no es básico y por lo tanto no participa en este tipo de resonancia a través del sistema pi. En los cloruros de ácido, el cloro está eliminando la densidad electrónica del carbono por inducción a través del sistema pi; hay más carácter deC=O doble enlace que en una amida y se necesita más energía para estirar el enlace carbonilo (lo que lleva a una mayor absorción de IR).
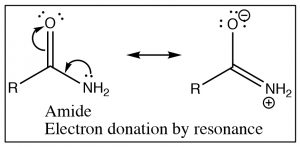
La donación de electrones a partir del nitrógeno significa que el par solitario en el nitrógeno de amida no está disponible para la donación a los ácidos. Esto significa que las amidas no son básicas, o más bien, ni siquiera tan básicas como las aminas en las que el par solitario está disponible gratuitamente para su donación a un ácido. Esto tiene importantes ramificaciones en los sistemas biológicos. En los polipéptidos, que son aminoácidos unidos por grupos funcionales amida (rodeados a continuación), las aminas se protonan a fisiológicaspH (existen comoRNH3+) [8]. Es un experimento de pensamiento interesante para predecir cómo se comportarían las proteínas y los péptidos si los nitrógenos de amida fueran más básicos.
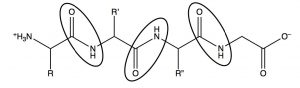
Si bien hay relativamente pocas cadenas laterales ácidas o básicas de origen natural en los polipéptidos (a partir de aminoácidos como el ácido glutámico o la lisina), cada péptido está unido por innumerables enlaces amida entre los aminoácidos individuales. Si todos estos nitrógenos de amida fueran protonados, los péptidos y proteínas tomarían estructuras muy diferentes (ya que tendrían una gran carga positiva que, en ausencia de contraiones, repelería otras partes de la molécula).


