25.7: Péptidos y Proteínas
- Page ID
- 72632
Clasificación
Los aminoácidos son los bloques de construcción de las estructuras de poliamida de péptidos y proteínas. Cada aminoácido está unido a otro por un enlace amida (o péptido) formado entre el\(\ce{NH_2}\) grupo de uno y el\(\ce{CO_2H}\) grupo del otro:

De esta manera, se construye una estructura polimérica de enlaces amida repetitivos en una cadena o anillo. Los grupos amida son planos y la configuración alrededor del\(\ce{C-N}\) enlace suele ser, pero no siempre, trans (Sección 24-1). El patrón de enlaces covalentes en un péptido o proteína se llama su estructura primaria:
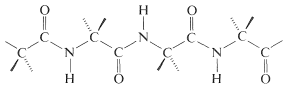
La distinción entre una proteína y un péptido no está completamente clara. Una elección arbitraria es llamar a proteínas solo a aquellas sustancias con pesos moleculares mayores a 5000. La distinción también podría hacerse en términos de diferencias en las propiedades físicas, particularmente la hidratación y conformación. Así, las proteínas, a diferencia de los péptidos, tienen cadenas muy largas que se enrollan y se pliegan de formas particulares, con moléculas de agua llenando los huecos en las bobinas y pliegues. El enlace de hidrógeno entre los grupos amida juega un papel decisivo en mantener las cadenas en yuxtaposición entre sí, en lo que a veces se llama la estructura secundaria y terciaria. \(^5\)Bajo la influencia del calor, disolventes orgánicos, sales, etc., las moléculas de proteína sufren cambios, a menudo irreversiblemente, llamados desnaturalización. De esta manera se alteran las conformaciones de las cadenas y el grado de hidratación, con el resultado de que disminuye la solubilidad y la capacidad de cristalización. Lo más importante es que las propiedades fisiológicas de la proteína generalmente se destruyen permanentemente en la desnaturalización. Por lo tanto, si se planifica una síntesis de una proteína, sería necesario duplicar no sólo las secuencias de aminoácidos sino también las conformaciones exactas de las cadenas y la forma de hidratación característica de la proteína nativa. Con los péptidos, las propiedades químicas y fisiológicas de los materiales naturales y sintéticos suelen ser idénticas, siempre que la síntesis duplique todos los elementos estructurales y configuracionales. Lo que esto significa es que un péptido asume automáticamente la estructura secundaria y terciaria característica del péptido nativo en la cristalización o disolución en disolventes.
La representación de estructuras peptídicas de cualquier longitud con fórmulas estructurales convencionales es engorrosa. Como resultado, se usan universalmente abreviaturas que emplean símbolos de tres letras para los aminoácidos componentes. Es importante que conozcas las convenciones para estas abreviaturas. Los dos posibles dipéptidos compuestos por un glicieno y una alanina son
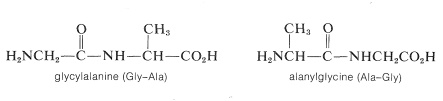
Observe que en las convenciones utilizadas para nombres y fórmulas abreviadas el aminoácido con el grupo amino libre (el aminoácido\(\ce{N}\) -terminal) siempre está escrito a la izquierda. El aminoácido con el grupo carboxilo libre (el aminoácido\(\ce{C}\) -terminal) siempre está escrito a la derecha. El guión entre las abreviaturas de tres letras para los ácidos indica que están unidos entre sí por un enlace amida.
Determinación de Secuencias de Aminoácidos
El procedimiento general para determinar la estructura primaria de un péptido o proteína consiste en tres etapas principales. Primero, se debe determinar el número y tipo de unidades de aminoácidos en la estructura primaria. Segundo, se identifican los aminoácidos en los extremos de las cadenas, y tercero, se determina la secuencia de los aminoácidos componentes en las cadenas.
La composición de aminoácidos generalmente se obtiene por hidrólisis ácida completa del péptido en sus aminoácidos componentes y análisis de la mezcla por cromatografía de intercambio iónico (Sección 25-4C). Este procedimiento se complica por el hecho de que el triptófano se destruye en condiciones ácidas. Además, la asparagina y la glutamina se convierten en ácidos aspártico y glutámico, respectivamente.
La determinación del ácido\(\ce{N}\) -terminal en el péptido se puede hacer por tratamiento del péptido con 2,4-dinitrofluorobenceno, sustancia muy reactiva en desplazamientos nucleofílicos con aminas pero no amidas (ver Sección 14-6B). El producto es un derivado\(\ce{N}\) -2,4-dinitrofenilo del péptido que, después de la hidrólisis de los enlaces amida, produce un\(\ce{N}\) -2,4-dinitrofenilaminoácido:
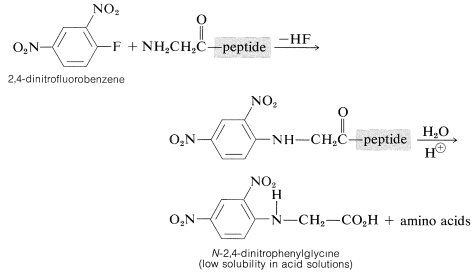
Estos derivados de aminoácidos pueden separarse de los aminoácidos ordinarios resultantes de la hidrólisis del péptido debido a que la baja basicidad del nitrógeno 2,4-dinitrofenil-sustituido (Sección 23-7C) reduce en gran medida la solubilidad del compuesto en solución ácida y altera su cromatografía comportamiento. La principal desventaja del método es que todo el péptido debe ser destruido para identificar el ácido\(\ce{N}\) uno-terminal.
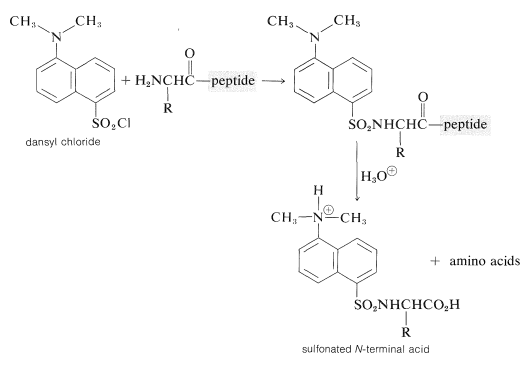
Un método relacionado y más sensible hace una sulfonamida del\(\ce{NH_2}\) grupo terminal con un reactivo llamado “cloruro de dansilo”. Al igual que con el 2,4-dinitrofluorobenceno, el péptido debe ser destruido por hidrólisis para liberar el aminoácido\(\ce{N}\) sulfonado, el cual puede identificarse espectroscópicamente en cantidades de microgramos:
Un método poderoso para secuenciar un péptido desde el extremo\(\ce{N}\) -terminal es la degradación de Edman en la que el isotiocianato de fenilo\(\ce{C_6H_5N=C=S}\), reacciona selectivamente con el aminoácido terminal en condiciones ligeramente básicas. Si la mezcla de reacción se acidifica entonces, el aminoácido terminal se escinde del péptido como una tiohidantoína cíclica,\(8\):
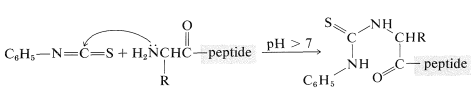
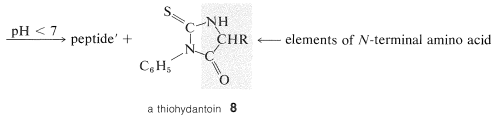
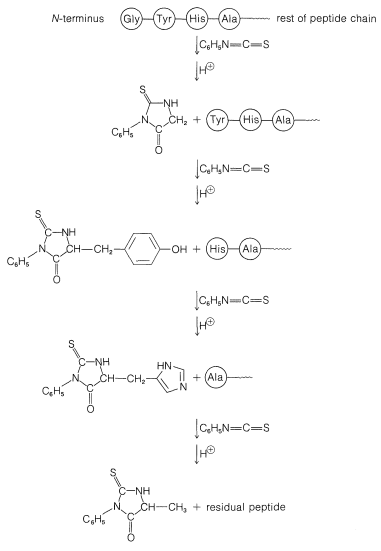
Existen reactivos simples que reaccionan selectivamente con el extremo carboxilo de un péptido, pero no han demostrado ser tan útiles en general para el análisis de los aminoácidos\(\ce{C}\) terminales como lo ha hecho la enzima carboxipeptidasa A. Esta enzima cataliza la hidrólisis del enlace peptídico que conecta el aminoácido con los grupos carboxilo terminales con el resto del péptido. Así, los aminoácidos en el extremo carboxilo se eliminarán uno por uno a través de la acción de la enzima. Siempre que se realicen correcciones apropiadas para diferentes tasas de hidrólisis de enlaces peptídicos para diferentes aminoácidos en el extremo carboxilo del péptido, la secuencia de hasta cinco o seis aminoácidos en el péptido puede deducirse del orden de su liberación por la carboxipeptidasa. Así, podría establecerse una secuencia tal como péptido-Ser-Leu-Tyr observando que la carboxipeptidasa libera aminoácidos del péptido en el orden Tyr, Leu, Ser:

Determinar las secuencias de aminoácidos de péptidos y proteínas grandes es muy difícil. Aunque la degradación de Edman e incluso la carboxipeptidasa se pueden usar para secuenciar completamente péptidos pequeños, no se pueden aplicar con éxito a cadenas peptídicas con varios cientos de unidades de aminoácidos. El éxito se ha obtenido con cadenas peptídicas largas mediante el empleo de reactivos, a menudo enzimas, para escindir selectivamente ciertos enlaces peptídicos. De esta manera la cadena se puede descomponer en varios péptidos más pequeños que se pueden separar y secuenciar. El problema entonces es determinar la secuencia de estos pequeños péptidos en la estructura original. Para ello, se llevan a cabo procedimientos alternativos para escisiones selectivas que producen diferentes conjuntos de péptidos más pequeños. Por lo general, no es necesario secuenciar completamente todos los conjuntos de péptidos. La composición global de aminoácidos y los respectivos grupos terminales de cada péptido pueden ser suficientes para mostrar secuencias solapantes a partir de las cuales se puede deducir lógicamente la secuencia completa de aminoácidos.
La mejor manera de mostrarte cómo funciona el método de superposición de secuenciación de péptidos es mediante un ejemplo específico. En este ejemplo, ilustraremos el uso de las dos enzimas más comúnmente utilizadas para la escisión selectiva de péptidos. Una es la tripsina, una enzima proteolítica del páncreas (PM 24,000) que cataliza selectivamente la hidrólisis de los enlaces peptídicos de aminoácidos básicos, lisina y arginina. La escisión ocurre en el lado carboxilo de la lisina o arginina:
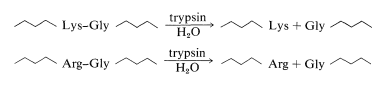
La quimotripsina es una enzima proteolítica del páncreas (PM 24.500) que cataliza la hidrólisis de enlaces peptídicos a los aminoácidos aromáticos, tirosina, triptófano y fenilalanina, más rápidamente que a otros aminoácidos. La escisión se produce en el lado carboxilo del aminoácido aromático:
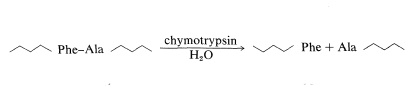
Nuestro ejemplo es la secuenciación de un péptido (P) derivado de la hidrólisis parcial de una proteína que, en la hidrólisis ácida completa, dio Ala, 3 Gly, Glu, His, 3 Lys, Phe, Tyr, 2 Val, y un equivalente molar de amoníaco.
1. El tratamiento del péptido (P) con carboxipeptidasa liberó alanina, y con 2,4-dinitrofluorobenceno seguido de hidrólisis dio el derivado 2,4-dinitrofenilo de valina. Estos resultados establecen el\(\ce{N}\) término -terminal como valina y el\(\ce{C}\) extremo -terminal como alanina. Los elementos estructurales conocidos ahora son
2. La hidrólisis parcial del péptido (P) con tripsina dio un hexapéptido, un tetrapéptido, un dipéptido y un equivalente molar de lisina. Los péptidos, que designaremos respectivamente como M, N y O, fueron secuenciados por degradación de Edman y se encontró que tenían estructuras:
\[\begin{array}{ll} \text{Gly}-\text{Ala} & \text{O} \\ \text{Val}-\text{Tyr}-\text{Glu}-\text{Lys} & \text{N} \\ \text{Val}-\text{Gly}-\text{Phe}-\text{Gly}-\text{His}-\text{Lys} & \text{M} \end{array}\]
Con esta información, se pueden escribir cuatro posibles estructuras para el péptido P original que son consistentes con los grupos finales conocidos y el hecho de que la tripsina escinde el péptido P en el lado carboxilo de la unidad de lisina. Por lo tanto
\[\begin{array}{cc} \text{N}-\text{M}-\text{Lys}-\text{O} & \text{M}-\text{N}-\text{Lys}-\text{O} \\ \text{N}-\text{Lys}-\text{M}-\text{O} & \text{M}-\text{Lys}-\text{N}-\text{O} \end{array}\]
3. La hidrólisis parcial del péptido P usando quimotripsina como catalizador dio tres péptidos, X, Y y Z. Estos no fueron secuenciados, pero se determinó su composición de aminoácidos:
\[\begin{array}{ll} \text{Gly, Phe, Val} & \text{X} \\ \text{Gly, His, Lys, Tyr, Val} & \text{Y} \\ \text{Ala, Glu, Gly, 2 Lys} & \text{Z} \end{array}\]
Esta información puede ser utilizada para decidir cuál de las estructuras alternativas deducidas anteriormente es la correcta. La quimotripsina escinde el péptido en el lado carboxilo de las unidades de fenilalanina y tirosina. Solo el péptido M contiene Phe, y si comparamos M con las composiciones de X, Y y Z, vemos que solo X e Y se superponen con M. El péptido Z contiene la única unidad Ala y debe ser el\(\ce{C}\) extremo -terminal. Si juntamos estas piezas para obtener un péptido, P' (que difiere de P por no tener el nitrógeno correspondiente al amoníaco formado en la hidrólisis completa) entonces P' debe tener la estructura X-Y-Z:
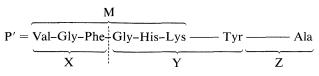
Esto puede no estar del todo claro, y será bueno considerar la lógica con cierto detalle. Los péptidos M y N tienen valinas\(\ce{N}\) -terminales, y una de ellas debe ser la unidad\(\ce{N}\) -terminal. El péptido M se solapa con X e Y, y debido a que X e Y se producen por una escisión en el lado carboxilo de Phe, las unidades X e Y tienen que estar conectadas en el orden X-Y. Debido a que el otro Val está en Y, el\(\ce{N}\) -terminal debe ser M. Esto reduce las posibilidades de
\[\begin{array}{c} \text{M}-\text{N}-\text{Lys}-\text{O} \\ \text{M}-\text{Lys}-\text{N}-\text{O} \end{array}\]
Hay dos unidades de Lys en Z, y esto significa que solo la secuencia M-N-Lys-O es consistente con la secuencia X-Y-Z, como se muestra:
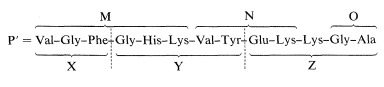
La pieza final del rompecabezas es la colocación del mol de amoníaco liberado del péptido original en la hidrólisis ácida. El amoníaco proviene de una función amida primaria:
\[\ce{R-CONH_2} \overset{\ce{H_3O}^\oplus}{\longrightarrow} \ce{RCO_2H} + \ce{NH_4^-}\]
El grupo amida no puede estar en el\(\ce{C}\) extremo terminal porque el péptido sería entonces inerte a la carboxipeptidasa. El único otro lugar posible es en el carboxilo de cadena lateral del ácido glutámico. La estructura completa puede escribirse como
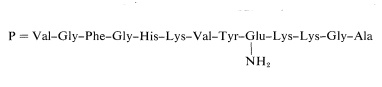
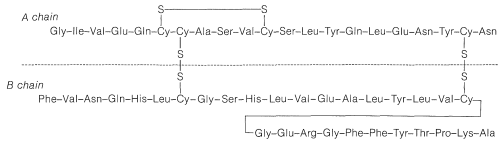
Mediante procedimientos como los señalados en esta sección se han secuenciado más de 100 proteínas. Este es un logro impresionante considerando la complejidad y el tamaño de muchas de estas moléculas (véase, por ejemplo, el Cuadro 25-3). Han pasado poco más de dos décadas desde que la primera secuencia de aminoácidos de una proteína fue reportada por F. Sanger, quien determinó la estructura primaria de la insulina (1953). Este trabajo sigue siendo un hito en la historia de la química porque estableció por primera vez que las proteínas tienen estructuras primarias definidas de la misma manera que otras moléculas orgánicas. Hasta ese momento, se cuestionaba abiertamente el concepto de estructuras primarias definidas para las proteínas. Sanger desarrolló el método de análisis de aminoácidos\(\ce{N}\) terminales utilizando 2,4-dinitrofluorobenceno y recibió el Premio Nobel en 1958 por su éxito en la determinación de la secuencia de aminoácidos de la insulina.
Métodos para formar enlaces peptídicos
Los problemas involucrados en la síntesis de péptidos son de mucha importancia práctica y han recibido considerable atención. La mayor dificultad para armar una cadena de digamos 100 aminoácidos en un orden particular es una de rendimiento global. Se requerirían al menos 100 etapas sintéticas separadas y, si el rendimiento en cada etapa fuera igual a\(n \times 100\%\), el rendimiento general sería\(\left( n^{100} \times 100\% \right)\). Si el rendimiento en cada paso fuera\(90\%\), el rendimiento global sería solo\(0.003\%\). Obviamente, una síntesis práctica de laboratorio de una cadena peptídica debe ser un proceso altamente eficiente. La extraordinaria capacidad de las células vivas para lograr síntesis de esta naturaleza, no de una sola sino de una amplia variedad de tales sustancias, es realmente impresionante.
Varios métodos para la formación de enlaces amida han sido discutidos en las Secciones 18-7A y 24-3A. A continuación se muestra la reacción más general, en la que X es algún grupo lábil reactivo (ver Tabla 24-1):
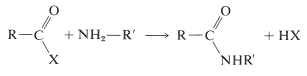
Cuando se aplica al acoplamiento de dos aminoácidos diferentes, es de esperar dificultad porque estas mismas reacciones pueden unir dos aminoácidos en un total de cuatro formas diferentes. Así, si empezamos con una mezcla de glicina y alanina, podríamos generar para dipéptidos, Gly-Ala, Ala-Gly, Gly-Gly y Ala-Ala.
Para evitar reacciones de acoplamiento no deseadas, se sustituye un grupo protector sobre la función amino del ácido que va a actuar como agente acilante. Además, todas las funciones amino, hidroxilo y tiol que pueden acilarse para dar productos no deseados normalmente deben protegerse. Por ejemplo, para sintetizar Gly-Ala libre de otros posibles dipéptidos, tendríamos que proteger el grupo amino de la glicina y el grupo carboxilo de la alanina:
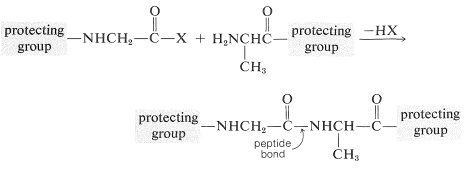
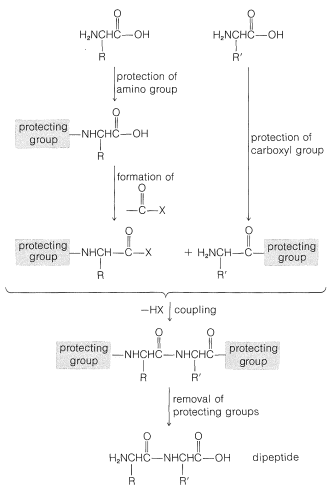
Algunos métodos para proteger las funciones amina e hidroxilo se discutieron previamente en las Secciones 23-13 y 15-9, respectivamente. Un resumen de algunos grupos protectores de uso común para\(\ce{NH_2}\),\(\ce{OH}\)\(\ce{SH}\), y\(\ce{CO_2H}\) funciones se encuentra en la Tabla 25-2, junto con las condiciones por las cuales los grupos protectores pueden eliminarse. Los mejores grupos protectores para\(\ce{NH_2}\) las funciones son fenilmetoxicarbonilo (benciloxicarbonilo) y terc - butoxicarbonilo. Ambos grupos se pueden eliminar por tratamiento con ácido, aunque el grupo terc - butoxicarbonilo es más reactivo. El grupo fenilmetoxicarbonilo se puede eliminar por reducción con hidrógeno sobre un catalizador metálico o con sodio en amoníaco líquido. Este método es más útil cuando, en la etapa de eliminación, es necesario evitar el tratamiento con ácido:
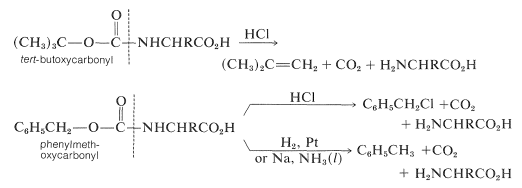
Tabla 25-2: Algunos grupos protectores de amina y carboxilo utilizados en síntesis de péptidos

En la mayoría de los casos, la formación del éster etílico proporciona un grupo protector satisfactorio para la función carboxilo.
La conversión del grupo carboxilo en un grupo más reactivo y el acoplamiento son pasos clave en la síntesis de péptidos. La reacción de acoplamiento debe ocurrir fácil y cuantitativamente, y con un mínimo de racemización de los centros quirales en la molécula. Este último criterio es el talón de Aquiles de muchas secuencias de acoplamiento posibles. La importancia de la no racemización se puede apreciar mejor con un ejemplo. Considere la síntesis de un tripéptido a partir de tres\(L\) aminoácidos protegidos, A, B y C, en dos etapas de acoplamiento secuencial,\(\text{C} \overset{\text{B}}{\rightarrow} \text{B}-\text{C} \overset{\text{A}}{\rightarrow} \text{A}-\text{B}-\text{C}\). Supongamos que el rendimiento de acoplamiento es cuantitativo, pero hay\(20\%\) formación del\(D\) isómero en el componente acilante en cada etapa de acoplamiento. Entonces el tripéptido consistirá en una mezcla de cuatro diastereómeros, solo\(64\%\) de los cuales será el\(L\) diastereómero deseado\(L\) (Ecuación 25-7):\(L\)
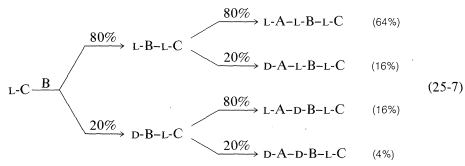 \(\tag{25-7}\)
\(\tag{25-7}\)
Esto es claramente inaceptable, especialmente para los péptidos de cadena más larga. Nueve etapas\(20\%\) de acoplamiento con el isómero incorrecto formado en cada uno darían solo\(13\%\) del decapéptido con la estereoquímica correcta.
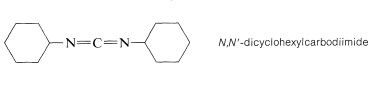
Los derivados carboxilo más utilizados en el acoplamiento de amida son azidas\(\ce{RCO-N_3}\)\(\ce{RCO-O-COR'}\), anhídridos mixtos y ésteres de fenoles moderadamente ácidos\(\ce{RCO-OAr}\) (ver Cuadro 24-1). También es posible acoplar ácido libre con un grupo amina usando una diimida\(\ce{R-N=C=N-R}\), la mayoría de las veces\(\ce{N}\),\(\ce{N'}\) -diciclohexilcarbodiimida.
El reactivo de diimida puede considerarse como un agente deshidratante. Los “elementos de agua” eliminados en el acoplamiento son consumidos por la diimida para formar una urea sustituida. La reacción general es
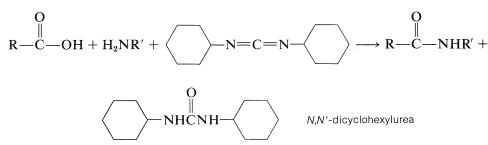
Esta reacción tiene lugar porque las diimidas\(\ce{-N=C=N}-\),, tienen sistemas de doble enlace acumulados reactivos como los de cetenos,\(\ce{-C=C=O}\)\(\ce{-N=C=O}\) isocianatos e isotiocianatos,\(\ce{-N=C=S}\) y son susceptibles al ataque nucleofílico en el carbono central. En la primera etapa de la reacción de acoplamiento de diimidas, la función carboxilo se añade a la imida para dar un intermedio acilo,\(9\). Este intermedio es un derivado de carboxilo activado\(\ce{RCO-X}\) y es mucho más reactivo hacia una función amino que el ácido parental. Por lo tanto, la segunda etapa es la aminólisis de\(9\) para dar el producto acoplado y\(\ce{N}\),\(\ce{N'}\) -diciclohexilurea:
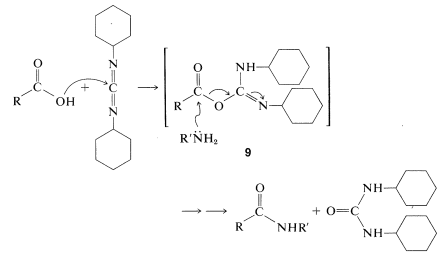
Después de completar una reacción de acoplamiento, y antes de que se pueda añadir otro aminoácido al\(\ce{N}\) extremo terminal, es necesario eliminar el grupo protector. Esto debe hacerse mediante reacciones selectivas que no destruyan los enlaces peptídicos o los grupos protectores de la cadena lateral. Esta parte de la síntesis peptídica se discute en la Sección 23-13, y algunas reacciones útiles para la eliminación\(\ce{N}\) de los grupos protectores terminales se resumen en la Tabla 25-2.
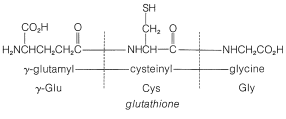
A pesar del gran número de etapas independientes involucradas en la síntesis de péptidos incluso pequeños, cada uno con sus problemas de rendimiento, racemización y selectividad, se ha logrado un éxito notable en la síntesis de péptidos grandes y algunos de los péptidos más pequeños. La síntesis de insulina (Figura 25-8) con sus 51 unidades de aminoácidos y puentes 3-disulfuro ha sido lograda por varios investigadores. Se han sintetizado varios péptidos hormonales importantes, a saber, glutatión, oxitocina, vasopresina y hormona tirotrópica (ver Figura 25-9). Un logro importante ha sido la síntesis de una enzima con actividad ribonucleasa reportada independientemente por dos grupos de investigadores, liderados por R. Hirschman (Merck) y R. B. Merrifield (Rockefeller University). Esta enzima es una de las proteínas más simples, teniendo una estenosis lineal de 124 residuos de aminoácidos. Es como un péptido, no una proteína, ya que asume la estructura secundaria y terciaria apropiada sin intervención bioquímica (Sección 25-7A). Como ejemplo específico de la estrategia involucrada en la síntesis peptídica, la síntesis escalonada de oxitocina se describe en la Figura 25-10, usando la notación abreviada en uso común.
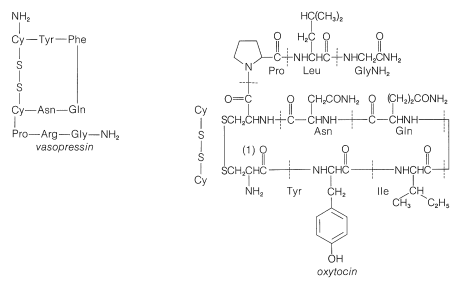
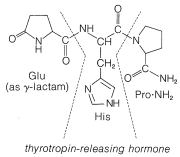
\ (\ ce {C_5}\), </em">no a\(\ce{C_1}\). La vasopresina (media izquierda) y la oxitocina (centro derecha) son hormonas peptídicas del lóbulo posterior de la glándula pituitaria. Funcionan principalmente para elevar la presión arterial (vasopresina), como antidiuréticos (vasopresina), y para promover la contracción del útero y los músculos de la lactancia (oxitocina). El aislamiento, identificación y síntesis de estas hormonas fue realizado por Vincent du Vigneaud, por lo que fue galardonado con el Premio Nobel de Química en 1965. La hormona liberadora de tirotropina (parte inferior) es una de varias hormonas peptídicas pequeñas secretadas por el lóbulo anterior de la glándula pituitaria. Estas son las hormonas “maestras” que funcionan para estimular la secreción hormonal de otras glándulas endocrinas. La tirotropina estimula el funcionamiento de la glándula tiroides.
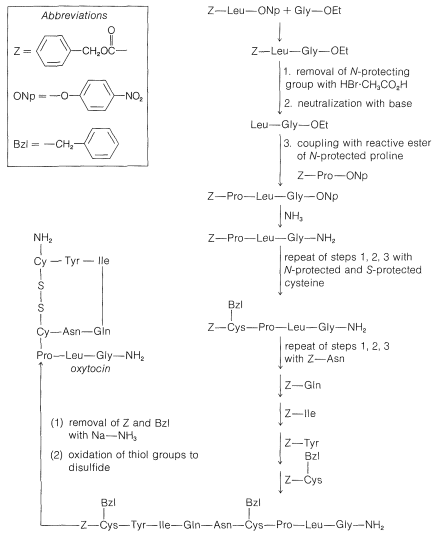
Figura 25-10: Síntesis paso a paso de oxitocina por el método del éster reactivo. En las abreviaturas utilizadas aquí\(-\text{Gly}-\ce{NH_2} = \ce{-NHCH_2CONH_2}\) y
Síntesis de péptidos en fase sólida
El rendimiento global en una síntesis de múltiples etapas de un péptido de tamaño incluso modesto es muy pobre a menos que cada etapa se pueda llevar a cabo de manera muy eficiente. Una elegante modificación de la síntesis clásica de péptidos ha sido desarrollada por R. B. Merrifield, la cual ofrece rendimientos mejorados al minimizar las pérdidas manipuladoras que normalmente asisten a cada etapa de una síntesis multietapa. La innovación clave es anclar el aminoácido\(\ce{C}\) -terminal a un soporte insoluble, y luego agregar unidades de aminoácidos por los métodos utilizados para la síntesis de soluciones. Después de que se haya logrado la secuencia deseada de aminoácidos, el péptido puede escindirse del soporte y recuperarse de la solución. Todas las reacciones involucradas en la síntesis deben, por supuesto, ser llevadas a su\(100\%\) conclusión esencialmente para que se pueda obtener un producto homogéneo. La ventaja de tener el péptido anclado a un soporte sólido es que prácticamente se eliminan laboriosas etapas de purificación; el material sólido se purifica simplemente lavando y filtrando sin transferir el material de un recipiente a otro. El método se ha conocido como síntesis de péptidos en fase sólida. A continuación se detallan más detalles de la síntesis en fase sólida.
La naturaleza del soporte polimérico es de gran importancia para una síntesis peptídica exitosa. Una que es ampliamente utilizada es una resina de poliestireno reticulado del tipo empleado en la cromatografía de intercambio iónico (Sección 25-4C). Es necesario que la resina sea insoluble pero tenga una estructura lo suficientemente suelta como para absorber solventes orgánicos. De lo contrario, los reactivos no podrán penetrar en los espacios entre las cadenas. Esto es indeseable porque las reacciones ocurren en la superficie de las partículas de resina y la mala penetración reduce en gran medida el número de equivalentes de sitios reactivos que se pueden obtener por gramo de resina. Finalmente, para anclar una cadena peptídica a la resina, se debe introducir en la resina un grupo funcional reactivo (generalmente un grupo clorometilo). Esto se puede hacer mediante una reacción de clorometilación de Friedel-Crafts, que sustituye al\(\ce{ClCH_2}-\) grupo en la posición 4 de los grupos fenilo en la resina:

Al inicio de la síntesis peptídica, el aminoácido\(\ce{C}\) -terminal se une a través de su grupo carboxilo a la resina por un ataque nucleófilo del ion carboxilato sobre los grupos clorometilo. El grupo\(\alpha\) -amino debe protegerse adecuadamente, como con el terc - butoxicarbonilo, antes de llevar a cabo esta etapa:
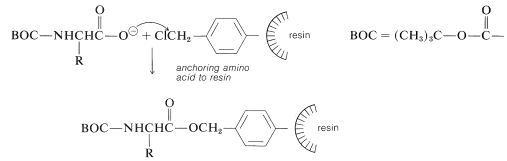
A continuación, el grupo protector de amina debe eliminarse sin escindir el enlace éster a la resina. La etapa de acoplamiento a un segundo aminoácido\(\ce{N}\) protegido sigue, con\(\ce{N}\),\(\ce{N'}\) -diciclohexilcarbodiimida como reactivo de acoplamiento de elección:
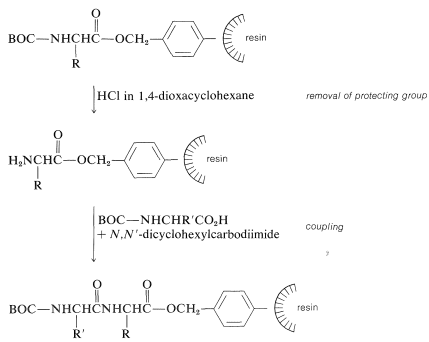
Las etapas de formación de enlaces peptídicos se repiten tantas veces como sea necesario para construir la secuencia deseada. En última instancia, la cadena peptídica se elimina de la resina, generalmente con\(\ce{HBr}\) ácido trifluoroetanoico anhidro\(\ce{CF_3CO_2H}\), o con anhidro\(\ce{HF}\). Este tratamiento también elimina los otros grupos protectores sensibles a los ácidos.
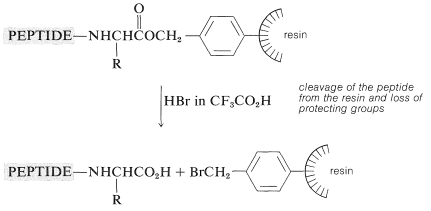
El método se presta maravillosamente al control automático, y se han desarrollado máquinas adecuadamente programadas para agregar reactivos y lavar el producto en los momentos apropiados. En la actualidad, la cadena se puede extender aproximadamente seis unidades de aminoácidos al día. Es necesario verificar la homogeneidad de la cadena peptídica en crecimiento a intervalos ya que si algún paso no avanza adecuadamente, el producto final puede estar seriamente contaminado con péptidos con la secuencia incorrecta.
En la síntesis de la enzima ribonucleasa por el método de Merrifield, los 124 aminoácidos se dispusieron en la secuencia de ribonucleasa a través de 369 reacciones y unas 12 mil operaciones individuales de la máquina automatizada de síntesis de péptidos sin aislamiento de ningún intermedio.
Separación de péptidos y proteínas
En muchos problemas de secuenciación de péptidos y síntesis de péptidos es necesario poder separar mezclas de péptidos y proteínas. Los principales métodos utilizados para este propósito dependen de las propiedades ácido-base o de los tamaños y formas moleculares.
La ultracentrifugación es ampliamente utilizada para la purificación, separación y determinación del peso molecular de proteínas. Un campo centrífugo, hasta 500,000 veces el de la gravedad, se aplica a la solución, y las moléculas se mueven hacia abajo en el campo según su masa y tamaño.
Las moléculas grandes también se pueden separar por filtración en gel (o cromatografía en gel), en donde las moléculas pequeñas se separan de las grandes pasando una solución sobre un gel que tiene poros de un tamaño en el que las moléculas pequeñas pueden penetrar y quedar atrapadas. Las moléculas mayores que el tamaño de poro se transportan con el disolvente. Esta forma de separación cromatográfica se basa en el “tamizado” más que en la afinidad química. Se dispone de una amplia gama de geles con diferentes tamaños de poro, y es posible fraccionar moléculas con pesos moleculares que van de 700 a 200,000. El peso molecular de una proteína se puede estimar por los tamaños de los poros que penetrará, o no, penetrará.
Las propiedades ácido-base, y por lo tanto el carácter iónico, de péptidos y proteínas también se pueden utilizar para lograr separaciones. La cromatografía de intercambio iónico, similar a la descrita para aminoácidos (Sección 25-4C), es un método de separación importante. Otro método basado en el carácter ácido-base y el tamaño molecular depende de las tasas diferenciales de migración de las formas ionizadas de una proteína en un campo eléctrico (electroforesis). Las proteínas, como los aminoácidos, tienen puntos isoeléctricos, que son los valores de pH a los que las moléculas no tienen carga neta. En todos los demás valores de pH habrá algún grado de carga iónica neta. Debido a que diferentes proteínas tienen diferentes propiedades iónicas, frecuentemente se pueden separar por electroforesis en soluciones tamponadas. Otro método, que se emplea para la separación y purificación de enzimas, es la cromatografía de afinidad, la cual fue descrita brevemente en la Sección 9-2B.
\(^5\)La distinción entre estructura secundaria y terciaria no es nítida. La estructura secundaria implica la consideración de las interacciones y relaciones espaciales de los aminoácidos en las cadenas peptídicas que están muy juntas en la estructura primaria, mientras que la estructura terciaria se refiere a aquellos que están muy separados en la estructura primaria.
Colaboradores y Atribuciones
- John D. Robert and Marjorie C. Caserio (1977) Basic Principles of Organic Chemistry, second edition. W. A. Benjamin, Inc. , Menlo Park, CA. ISBN 0-8053-8329-8. This content is copyrighted under the following conditions, "You are granted permission for individual, educational, research and non-commercial reproduction, distribution, display and performance of this work in any format."


